食管癌是起源于食管黏膜上皮的恶性肿瘤[1],2020年全球癌症统计数据显示,其发病例数在所有恶性肿瘤中居第7位,死亡例数居第6位[2]。食管癌的发病率及病理类型在不同国家和地区之间差异显著,东亚地区发病率最高,其中食管鳞状细胞癌(esophageal squamous cell carcinoma,ESCC)占比达90%以上;而欧美国家发病率相对较低,病理类型以食管腺癌为主[2]。我国食管癌年发病和死亡例数在恶性肿瘤中分别居第6位和第5位,占全球食管癌发病和死亡总例数的50%以上,疾病负担居全球首位[3]。确诊时的临床分期是决定食管癌预后的最主要因素,而我国食管癌早期诊断率不足15%,患者总体预后差,5年生存率仅为30.3%[4]。食管癌在早期阶段缺乏报警症状,大多数患者因进行性吞咽困难或出现转移症状就诊而发现,此时肿瘤往往已进展至中晚期。因此,在食管癌高发地区和高危人群中广泛开展筛查是提高早期诊断率、降低患者死亡率的关键。
上消化道内镜结合活检组织病理学是筛查和诊断食管癌及癌前病变的金标准,但并不适合大规模人群筛查。国内研究显示,即使在河南林州、河北磁县等食管癌的聚集性高发区,通过内镜筛查47~134例无症状受检者才能发现1例食管癌或高级别上皮内瘤变[5]。我国人口基数庞大,40~75岁人口近6亿,而消化内镜医师和诊疗设备相对缺乏,难以满足人群普查需要。此外,内镜检查属侵入性操作,部分受检者难以耐受,少部分甚至可能出现出血、穿孔等并发症。因此,采用相对简便、高效的非内镜筛查方式对目标人群进行风险分层并对高危者开展内镜精查,是更符合卫生经济学原则和我国国情的防控策略。近年来流行病学研究方法不断改进,体外诊断、生命组学和人工智能等技术不断进步,针对我国高发的ESCC催生出许多筛查新路径与新方法,如疾病风险评估模型,食管细胞学检测,以及血液、呼出气体和唾液标志物检测等,为食管癌的精准筛查和有效防控带来了新的曙光。
1 疾病风险预测模型疾病风险预测模型利用相对容易获得的危险因素信息定量评估疾病风险,是目前广泛应用的高危人群筛选方法,并已在结直肠癌[6]、胃癌[7]等消化道肿瘤的筛查中广泛应用。国内外学者已建立了多种ESCC发病风险预测模型,模型的建立多基于社区/医院人群为基础的横断面研究和队列研究,建模方法以多因素logistic回归和Cox比例风险回归模型为主,模型预测变量主要包括流行病学宏观暴露因素和遗传易感性标志物[8]。
1.1 基于宏观暴露因素的疾病风险预测模型基于宏观暴露因素的疾病风险预测模型具有调查简便、计算快捷、不产生额外花费等优点。国内外学者目前已构建了多个基于宏观暴露因素的ESCC发病风险预测模型(表 1)[9-18],模型的ROC AUC可达0.70~0.88。我国是全球ESCC发病和死亡例数最多的国家,构建上述模型所基于的人群大部分来自于我国高发区。Liu等[10]基于河南安阳地区横断面数据分别建立了≤60岁、>60岁人群的食管鳞状上皮重度不典型增生及ESCC发病风险预测模型,并采用交叉验证法对模型进行内部验证。在该研究中,≤60岁人群模型纳入了年龄、以煤或木头作为烹调燃料源、BMI、不明原因的上腹部疼痛、进食速度5个预测变量,>60岁人群模型纳入了年龄、ESCC家族史、吸烟、BMI、杀虫剂暴露史、饮食习惯规律性、烫热饮食习惯、进食速度、霉变食物暴露史9个变量。该模型在保证阳性病例检出灵敏度达100%的前提下,可以分别减少≤60岁人群、>60岁人群27%和9%的内镜检查需求。该模型的优势在于预测价值较好(尤其是≤60岁人群模型),建模人群来源于高发地区社区人群,但未经外部验证,且进食速度、进食习惯、餐饮温度等变量难以在短时间内客观、准确地评估。该团队在后续研究中基于河南安阳和广东深圳2家医院食管癌机会性筛查人群建立并验证了食管高级别病变预测模型[11],其中来自安阳的5 624名内镜受检者为训练集,来自深圳的5 765名为外部验证集。该模型纳入了年龄、吸烟、BMI、吞咽困难、胸骨后疼痛5个变量,评分简单且预测价值高,在训练集和验证集中AUC分别可达0.871和0.843,有望在机会性筛查人群的食管癌风险预测中发挥作用。Chen等[12]基于我国农村地区2007-2012年食管癌筛查项目参与者3年随访期间食管癌发病数据建立了ESCC发病风险预测模型,纳入年龄、性别、上消化道癌家族史、吸烟状态、背部或胸骨后疼痛、高盐饮食和新鲜水果摄入、消化性溃疡病史、食管炎病史8个变量,预测3年内食管癌发病的AUC为0.81,并能在保证较高灵敏度和特异度的情况下减少3.11%的上消化道内镜筛查需求。该模型的推广应用有望进一步提高我国食管癌高发农村地区的筛查效率,提高内镜检查阳性率。
|
|
表 1 ESCC发病风险预测模型汇总 Tab 1 Risk prediction models for ESCC |
伊朗戈勒斯坦省是全球ESCC发病率最高的地区之一。Etemadi等[9]基于2003-2007年开展的伊朗戈勒斯坦病例对照研究人群,分别构建了ESCC和食管鳞状上皮重度不典型增生发病风险预测模型。ESCC与重度不典型增生预测模型均纳入10个预测变量(年龄、种族、吸烟、鸦片使用史、受教育程度、婚姻状况、口腔健康、家族史、茶温、水源),其中ESCC预测模型的AUC为0.77,重度不典型增生预测模型的AUC为0.71。欧美国家ESCC发病率相对较低,但由于其公共卫生数据系统完善,已有学者基于瑞典[15]、挪威和英国[16]等国家的人口登记数据或大型队列研究构建了ESCC发病风险预测模型,模型AUC与高发地区类似,可达0.70~0.81。
1.2 基于遗传易感性的疾病风险预测模型遗传易感性是指由于个体遗传结构不同,在环境暴露因素影响下呈现出易患某些疾病的倾向。ESCC存在遗传易感性基因和单核苷酸多态性位点,因此遗传易感性标志物具有疾病风险预测的潜能。Yokoyama等[17]基于病例对照研究设计,纳入来自日本3家医院的868名男性受试者,以乙醛脱氢酶2基因表达状态和4个非遗传性预测因素(饮酒、吸烟、蔬菜摄入量、水果摄入量)构建了ESCC发病风险预测模型。该模型的AUC可达0.86,如果对模型评分前10%的高危人群开展内镜筛查,阳性预测值可较日本现行方案提高1.7%。但由于该模型的构建仅基于医院ESCC男性患者,且未经外部验证,模型的人群代表性和外推性受到一定限制。Chang等[18]采用病例对照研究设计,基于中国5个省市(北京市、河北省、河南省、江苏省和广东省)5家医院9 805名ESCC患者与10 493名正常对照,通过全基因组关联研究找出了25个与食管癌易感性相关的单核苷酸多态性位点,采用加权遗传风险评分方法计算遗传因素风险得分,并联合4个非遗传变量(性别、年龄、吸烟、饮酒)构建了ESCC发病风险预测模型,其AUC为0.709。该模型的构建基于多中心、大样本研究,并考虑了ESCC潜在的遗传因素与非遗传因素,值得进一步开展外部验证研究。
2 食管细胞学检测食管细胞学检查是指采用特定器械收集食管脱落细胞,并对收集的细胞学样本进行形态学观察或联合生物标志物测定的过程。由于其操作相对简便且可直接获取食管细胞用于分析,因此在食管癌人群筛查中具有良好应用前景。国外已有研究报道利用食管细胞海绵(cytosponge,图 1A)联合生物标志物检查可有效地筛查巴雷特食管或对其进行风险分层。Kadri等[19]对英国504例长期服用制酸剂的受试者开展食管cytosponge联合三叶因子3(trefoil factor 3,TFF3)检测,以上消化道内镜检查为金标准,结果显示cytosponge诊断病灶长度>2 cm的巴雷特食管的灵敏度和特异度分别为90.0%和93.5%,并且患者接受度较高。Ross-Innes等[20]基于468例巴雷特食管患者构建了包含临床变量和细胞学结果的巴雷特食管风险分层模型,纳入腺上皮异型性、p53染色异常、极光蛋白激酶A染色3个细胞学结果,以及年龄、腰臀比、巴雷特食管病灶长度3个临床变量,经风险分层后,低危组、中危组和高危组检出腺上皮高级别上皮内瘤变和食管腺癌的概率分别为0、14%和87%。Chettouh等[21]和Li等[22]分别将cytosponge与DNA甲基化检测和miRNA检测相结合,2种模式在巴雷特食管的无创化筛查和诊断中均能达到较高的灵敏度和特异度。一项随机对照研究显示,在英国反酸症状人群中使用cytosponge联合TFF3检测与传统方式相比可将巴雷特食管检出率提升10.6倍[23]。
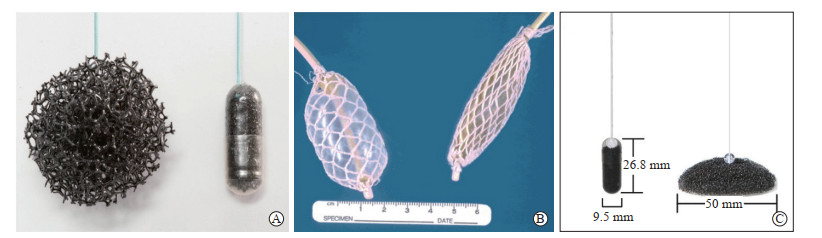
|
图 1 食管细胞采集装置 Fig 1 Esophageal cell collection device A: Cytosponge cell collection device; B: Traditional balloon cell collection device; C: Novel cell enrichment device invented by the author's study group. |
20世纪60年代,原河南医学院病理学教研室沈琼教授研发了食管拉网细胞学方法,并在河南林县开展食管癌筛查。20世纪80年代以来,国内外团队合作在我国ESCC高发区河南开展了一系列筛查研究,评价了食管脱落细胞学用于筛查的可行性。Roth等[24]对439例无症状受试者先后进行了球囊拉网细胞学(图 1B)、胶囊海绵细胞学和上消化道内镜结合活检检查,结果显示,球囊拉网细胞学和胶囊海绵细胞学诊断食管鳞状上皮异型增生与ESCC的灵敏度分别为47%和24%,特异度分别为81%和92%。Pan等[25]采用球囊拉网和液基细胞学制片方法对740例河南林州的受试者进行筛查,结果显示该方法诊断食管鳞状上皮异型增生与ESCC的灵敏度和特异度分别为46%和84%。上述研究结果提示,传统食管脱落细胞学诊断食管鳞状上皮异型增生和ESCC的灵敏度均较低,不适宜用于高发区无症状人群筛查。针对上述问题,本团队改良了我国传统拉网和国外cytosponge细胞采集器,细胞富集装置采用穹顶状设计,完全展开直径可达5 cm,保证其采样过程中对贲门和食管全长都贴合良好,增大接触面积(图 1C)。富集装置材料孔隙密度更高,进一步提升了采样效率,每位受检者可采集不少于300万个细胞,采用深度学习系统自动扫描全拨片辅助细胞病理医师判读可减少漏诊风险。在高发区社区人群中的初步应用结果显示,该方法筛查ESCC的灵敏度和特异度分别可达90.0%和93.7%[26]。进一步开展的多中心、前瞻性队列研究显示,综合细胞学图像特征和流行病学危险因素构建的食管-胃交界处的ESCC和食管腺癌筛查风险测评模型的灵敏度和特异度分别为94.5%和91.9%,经过该方法初筛后人群中90.3%的个体可归入低风险组而无需内镜检查[27]。
3 血液标志物检测 3.1 蛋白标志物及抗体蛋白标志物及其相关抗体是最早发现并应用于临床的肿瘤标志物,可用于食管癌诊断的主要包括癌胚抗原(carcinoembryonic antigen,CEA)、鳞状细胞癌相关抗原(squamous carcinoma-associated antigen,SCC)、细胞角蛋白19片段抗原21-1(cytokeratin 19 fragment antigen 21-1,Cyfra21-1)和p53抗体等。上述肿瘤标志物灵敏度均相对较低,单独应用于食管癌的诊断价值不大,多项肿瘤指标联合检测有助于提高诊断效能。Jing等[28]报道,CEA、糖类抗原19-9、糖类抗原24-2、SCC联合检测食管癌的灵敏度为68.4%,特异度为71.5%,但在Ⅰ期食管癌中阳性率较低。一项meta分析(纳入44项研究)结果显示CEA、Cyfra21-1、p53抗体和SCC诊断ESCC的阳性似然比分别为5.94、12.11、6.71和7.66,阴性似然比分别为0.76、10.59、0.75和0.68,但诊断早期癌的灵敏度均不理想[29]。一项系统评价归纳了35种抗体对ESCC的诊断效能,结果显示抗体的特异度均较高(中位数为98.3%),但灵敏度较低(中位数为26.7%),特别是对早期病变漏诊率较高[30]。Xu等[31]在388例ESCC和125例正常对照者中同时检测6种抗体(p53、NY-ESO-1、基质金属蛋白酶7、热激蛋白70、过氧化物还原蛋白Ⅵ和Bmi-1抗体),检出任意一种抗体即视为筛查阳性。此方法诊断ESCC的灵敏度和特异度分别为57%和95%,诊断早期ESCC的灵敏度和特异度分别为45%和95%。
3.2 非编码RNA 3.2.1 miRNAmiRNA是由20~24个核苷酸组成的高度保守的单链非编码RNA,通过对靶基因的转录后调控影响多种生命活动,包括肿瘤的发生和发展[32]。目前已有较多研究评价了血液miRNA对ESCC的诊断效能。Sudo等[33]采用病例对照研究设计,以566例ESCC患者和4 965例非癌症患者为研究对象(分别来源于日本国家癌症中心生物样本库和一项健康体检项目),并分成训练集和验证集。通过检测血清标本miRNA表达水平,研究者鉴定出6个与日本人群ESCC发病显著相关的miRNA(miRNA-8073、miRNA-6820-5p、miRNA-6794-5p、miRNA-3196、miRNA-744-5p和miRNA-6799-5p),以这6个miRNA为预测变量构建了ESCC发病风险预测模型,该模型在验证集中预测ESCC的AUC高达1.00(灵敏度和特异度分别为100%和98%),但由于建模样本主要由医院来源的病例和对照样本组成,该模型的预测效能有待在日本人群乃至其他种族人群中进一步验证。Miyoshi等[34]鉴定了8个在ESCC患者血浆中高表达的miRNA(miRNA-103、miRNA-106b、miRNA-151、miRNA-17、miRNA-181a、miRNA-21、miRNA-25、miRNA-93)并构建诊断预测模型,在几项大型、独立、多种族、多中心的回顾性和前瞻性队列中的验证结果显示,该模型预测ESCC的AUC可达0.80~0.93,且对早期ESCC具有良好区分度。Yao等[35]发表的一项系统评价和meta分析评价了43种不同miRNA对ESCC的诊断效能,总体灵敏度、特异度和AUC分别为0.794、0.779和0.86;其中报道较多、诊断效能较高的miRNA包括miRNA-21、miRNA-25、miRNA-31、miRNA-100、miRNA-223、miRNA-375和miRNA-1322,经meta分析合并后其诊断ESCC的AUC分别为0.84、0.80、0.91、0.70、0.81、0.84和0.88。
3.2.2 lncRNAlncRNA是指长度在200个核苷酸以上且不编码蛋白质的RNA,可在转录和翻译水平调控基因表达,与肿瘤的发生、发展密切相关[32]。目前在ESCC中研究较多的lncRNA包括POU3F3旁基因编码的长基因间非蛋白编码RNA(a long intergenic non-protein coding RNA encoded by a gene located next to POU3F3,linc-POU3F3)和HOX转录反义RNA(HOX transcript antisense RNA,HOTAIR)。Li等[36]发现并验证了linc-POU3F3在ESCC组织和正常组织中的表达差异,进一步的机制研究证实其通过诱导POU3F3基因CpG岛过度甲基化发挥致癌作用。Tong等[37]检测了147例食管癌患者和123名健康志愿者的循环linc-POU3F3,结果显示linc-POU3F3诊断ESCC的AUC、灵敏度和特异度分别为0.842、72.8%和89.4%。此外,联合检测血液linc-POU3F3和SCC可进一步提高对ESCC的诊断效能,并能检出80.8%的早期癌。Wang等[38]检测了50例ESCC患者和20例健康志愿者血液HOTAIR水平,结果显示血液HOTAIR诊断ESCC的AUC为0.793,HOTAIR浓度随TNM分期的增高而升高且在手术切除肿瘤后显著下降,因此有望成为ESCC早期诊断和手术后复发监测的可靠标志物。
3.3 循环肿瘤DNA(circulating tumor DNA,ctDNA)ctDNA指坏死或凋亡的肿瘤细胞释放到外周血中的肿瘤DNA片段,其带有肿瘤特异性突变或表观遗传学改变。ctDNA甲基化水平检测目前是ESCC非侵入性诊断研究中的热点。Kawakami等[39]研究显示,在52例未经治疗的食管腺癌患者中有13例(25%)血浆大肠腺瘤性息肉病(adenomatous polyposis coli,APC)基因甲基化水平增高,而在32例ESCC患者中只有2例(6%)血浆中可检测到高甲基化APC基因,并且血浆APC高甲基化与不良预后相关。Hibi等[40]研究显示23%(7/31)的ESCC患者血清p16基因存在异常甲基化。由于目前ctDNA诊断ESCC的研究尚不充分且检测费用相对较高,其临床应用尚未普及。
3.4 代谢产物代谢组学是定性和定量研究生物体系(细胞、组织或生物体)在病理状态下产生的所有代谢产物的动态变化的科学[41]。肿瘤细胞的代谢途径和代谢产物与正常细胞存在显著差异,进而导致肿瘤患者和健康人群血液代谢产物谱的差异,这些存在差异的代谢产物有望成为诊断肿瘤的新型标志物。Liu等[42]对53例ESCC患者和53名健康受试者的血浆样本进行代谢组学检测和分析,发现了25种浓度上调和5种浓度下调的代谢产物,并通过分层聚类分析验证了上述代谢产物对ESCC患者和健康人的区分效果。Ma等[43]采用HPLC测定了51例ESCC患者和60名健康受试者血浆游离氨基酸水平,结果显示与健康受试者相比,ESCC患者血浆天冬氨酸、谷氨酸、甘氨酸、组氨酸、苏氨酸、牛磺酸、丙氨酸、甲硫氨酸、亮氨酸、异亮氨酸和苯丙氨酸水平降低,半胱氨酸水平升高,利用血浆氨基酸代谢谱建立的多变量模型对ESCC患者与健康人群具有良好的区分度。目前利用代谢产物谱系差异诊断ESCC的研究仍不充分,尚无大样本临床研究定量评估代谢组学方法对ESCC诊断的准确性。
4 呼出气体标志物检测人类呼出的气体由2 000余个特异的挥发性有机化合物复合体(volatile organic compound,VOC)组成,对呼出气体进行分析是一种快速、无创且便捷的检测方法,VOC中可能包含潜在的肿瘤标志物。Markar等[44]评估了呼出气体检测对食管和胃腺癌的诊断价值,研究纳入英国3个中心163例食管胃腺癌患者和172名健康受试者,测定丁酸、戊酸、己酸、丁醛和癸醛5种VOC,以这5种VOC建立的模型诊断食管胃腺癌的灵敏度和特异度分别为80%和81%,AUC可达0.85,但该研究所纳入患者中早期病例较少,尚不能评估该模型对早期上消化道腺癌的诊断价值。Zou等[45]初步评估了呼出气体VOC检测对中国ESCC患者的诊断价值,运用质谱分析法来测定VOC,结果显示ESCC患者的全质谱图中有20个离子峰与对照组显著不同,最终确定了与ESCC高度相关的7个离子峰,其诊断ESCC的灵敏度和特异度分别可达86.2%和89.5%。该研究样本量较小,且绝大部分ESCC为中晚期患者,该方法能否用于人群筛查仍需进一步研究和验证。
5 唾液标志物检测唾液腺血供丰富,唾液被认为是血液循环的终末产物,血浆中存在的分子大多数也存在于唾液中,因此,唾液也被用于癌症、传染病、心血管疾病等疾病的检测。Xie等[46]通过微阵列分析从7例ESCC患者与3名健康受试者的全唾液样本中鉴定出6种差异表达miRNA(miRNA-144、miRNA-10b、miRNA-451、miRNA-486-5p、miRNA-634、miRNA-21),并在39例食管癌患者和19名健康人的全唾液和唾液上清液样本中通过qPCR对其表达水平进行了验证,结果发现全唾液中miRNA-10b、miRNA-144、miRNA-451及唾液上清液中miRNA-10b、miRNA-144、miRNA-21、miRNA-451的表达显著上调。暨南大学联合汕头大学和安阳市肿瘤医院团队在ESCC患者唾液外泌体中鉴定出2种特异性富集的转运RNA源性小RNA,并在大规模人群中验证了其诊断(灵敏度为90.50%,特异度为94.20%)和预后预测效能[47]。另有研究报道,唾液miRNA-114诊断ESCC的灵敏度为74.6%,特异度为92.0%[48];唾液miRNA-21诊断Ⅰ/Ⅱ期ESCC的灵敏度为89%,特异度为64%[49]。
6 总结与展望开展筛查和早期诊治是ESCC防控的关键举措,而研发和应用简便、有效的非侵入性初筛方法有助于提高筛查效率、普及度和患者依从性,进而提升防控效果。目前ESCC发病风险预测模型已有较多研究报道,其优势在于操作简便、价格低廉,但诊断效能仍有待进一步提高,且不同预测模型的外推性缺乏可靠验证,因此在筛查中尚未广泛应用。今后亟须开展针对已建立的ESCC发病风险预测模型的大样本、多地区外部验证和效果比较研究。新型食管细胞学检测的诊断准确性和患者耐受性较传统拉网细胞学检查有了质的提升,其优势在于能直接获取病变部位细胞,且对早期ESCC及癌前病变均具有较高诊断灵敏度,目前已应用于高发地区及高危个体的筛查,并在全国范围内逐步推广。人工智能图像识别、自动化风险测评、生物标志物检测等新技术手段有望进一步提升食管细胞学检测的效率和准确性。针对血液标志物已有较多研究报道,其中蛋白标志物、抗体和代谢产物检测对早期ESCC的诊断灵敏度和特异度较低,血浆miRNA组合在几项大样本研究中显示出良好的诊断效能。目前我国尚无获批上市的食管癌体外诊断试剂,因此需进一步加速产学研深度融合与创新成果转化。唾液和呼出气体标志物检测具有采样完全无创、方便快捷等优点,但目前相关研究尚不充分,其样本保存和快速检测方法的改进、肿瘤特异性标志物的筛选等工作仍有待多学科联合攻关。此外,目前尚无针对不同类别筛查手段联合应用于食管癌筛查的可行性和效果研究,值得关注和进一步探索。
| [1] |
THRUMURTHY S G, CHAUDRY M A, THRUMURTHY S S D, MUGHAL M. Oesophageal cancer: risks, prevention, and diagnosis[J/OL]. BMJ, 2019, 366: l4373. DOI: 10.1136/bmj.l5391.
|
| [2] |
SUNG H, FERLAY J, SIEGEL R, LAVERSANNE M, SOERJOMATARAM I, JEMAL A, et al. Global cancer statistics 2020:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA A Cancer J Clin, 2021, 71: 209-249. DOI:10.3322/caac.21660 |
| [3] |
ZHENG R, ZHANG S, ZENG H, WANG S, SUN K, CHEN R, et al. Cancer incidence and mortality in China, 2016[J]. J Nat Cancer Cent, 2022, 2: 1-9. DOI:10.1016/j.jncc.2022.02.002 |
| [4] |
ZENG H, CHEN W, ZHENG R, ZHANG S, JI J S, ZOU X, et al. Changing cancer survival in China during 2003-15: a pooled analysis of 17 population-based cancer registries[J/OL]. Lancet Glob Health, 2018, 6: e555-e567. DOI: 10.1016/S2214-109X(18)30127-X.
|
| [5] |
CHEN R, LIU Y, SONG G, LI B, ZHAO D, HUA Z, et al. Effectiveness of one-time endoscopic screening programme in prevention of upper gastrointestinal cancer in China: a multicentre population-based cohort study[J]. Gut, 2021, 70: 251-260. |
| [6] |
YEOH K G, HO K Y, CHIU H M, ZHU F, CHING J Y L, WU D C, et al. The Asia-Pacific colorectal screening score: a validated tool that stratifies risk for colorectal advanced neoplasia in asymptomatic Asian subjects[J]. Gut, 2011, 60: 1236-1241. DOI:10.1136/gut.2010.221168 |
| [7] |
CAI Q, ZHU C, YUAN Y, FENG Q, FENG Y, HAO Y, et al. Development and validation of a prediction rule for estimating gastric cancer risk in the Chinese high-risk population: a nationwide multicentre study[J]. Gut, 2019, 68: 1576-1587. DOI:10.1136/gutjnl-2018-317556 |
| [8] |
李贺, 陈万青. 食管癌发病风险评估模型的研究进展[J]. 中国肿瘤, 2019, 28: 883-890. |
| [9] |
ETEMADI A, ABNET C C, GOLOZAR A, MALEKZADEH R, DAWSEY S M. Modeling the risk of esophageal squamous cell carcinoma and squamous dysplasia in a high risk area in Iran[J]. Arch Iran Med, 2012, 15: 18-21. |
| [10] |
LIU M, LIU Z, CAI H, GUO C, LI X, ZHANG C, et al. A model to identify individuals at high risk for esophageal squamous cell carcinoma and precancerous lesions in regions of high prevalence in China[J]. Clin Gastroenterol Hepatol, 2017, 15: 1538-1546. DOI:10.1016/j.cgh.2017.03.019 |
| [11] |
LIU Z, GUO C, HE Y, CHEN Y, JI P, FANG Z, et al. A clinical model predicting the risk of esophageal high-grade lesions in opportunistic screening: a multicenter real-world study in China[J/OL]. Gastrointest Endosc, 2020, 91: 1253-1260.e3. DOI: 10.1016/j.gie.2019.12.038.
|
| [12] |
CHEN W, LI H, REN J, ZHENG R, SHI J, LI J, et al. Selection of high-risk individuals for esophageal cancer screening: a prediction model of esophageal squamous cell carcinoma based on a multicenter screening cohort in rural China[J]. Int J Cancer, 2021, 148: 329-339. DOI:10.1002/ijc.33208 |
| [13] |
SHEN Y, XIE S, ZHAO L, SONG G, SHAO Y, HAO C, et al. Estimating individualized absolute risk for esophageal squamous cell carcinoma: a population-based study in high-risk areas of China[J/OL]. Front Oncol, 2021, 10: 598603. DOI: 10.3389/fonc.2020.598603.
|
| [14] |
YANG X, SUO C, ZHANG T, YIN X, MAN J, YUAN Z, et al. A nomogram for screening esophageal squamous cell carcinoma based on environmental risk factors in a high-incidence area of China: a population-based case-control study[J/OL]. BMC Cancer, 2021, 21: 343. DOI: 10.1186/s12885-021-08053-7.
|
| [15] |
WANG Q L, LAGERGREN J, XIE S H. Prediction of individuals at high absolute risk of esophageal squamous cell carcinoma[J/OL]. Gastrointest Endosc, 2019, 89: 726-732.e2. DOI: 10.1016/j.gie.2018.10.025.
|
| [16] |
WANG Q L, NESS-JENSEN E, SANTONI G, XIE S H, LAGERGREN J. Development and validation of a risk prediction model for esophageal squamous cell carcinoma using cohort studies[J]. Am J Gastroenterol, 2021, 116: 683-691. DOI:10.14309/ajg.0000000000001094 |
| [17] |
YOKOYAMA T, YOKOYAMA A, KUMAGAI Y, OMORI T, KATO H, IGAKI H, et al. Health risk appraisal models for mass screening of esophageal cancer in Japanese men[J]. Cancer Epidemiol Biomarkers Prev, 2008, 17: 2846-2854. DOI:10.1158/1055-9965.EPI-08-0397 |
| [18] |
CHANG J, HUANG Y, WEI L, MA B, MIAO X, LI Y, et al. Risk prediction of esophageal squamous-cell carcinoma with common genetic variants and lifestyle factors in Chinese population[J]. Carcinogenesis, 2013, 34: 1782-1786. DOI:10.1093/carcin/bgt106 |
| [19] |
KADRI S R, LAO-SIRIEIX P, O'DONOVAN M, DEBIRAM I, DAS M, BLAZEBY J M, et al. Acceptability and accuracy of a non-endoscopic screening test for Barrett's oesophagus in primary care: cohort study[J/OL]. BMJ, 2010, 341: c4372. DOI: 10.1136/bmj.c4372.
|
| [20] |
ROSS-INNES C S, CHETTOUH H, ACHILLEOS A, GALEANO-DALMAU N, DEBIRAM-BEECHAM I, MACRAE S, et al. Risk stratification of Barrett's oesophagus using a non-endoscopic sampling method coupled with a biomarker panel: a cohort study[J]. Lancet Gastroenterol Hepatol, 2017, 2: 23-31. DOI:10.1016/S2468-1253(16)30118-2 |
| [21] |
CHETTOUH H, MOWFORTH O, GALEANO-DALMAU N, BEZAWADA N, ROSS-INNES C, MACRAE S, et al. Methylation panel is a diagnostic biomarker for Barrett's oesophagus in endoscopic biopsies and non-endoscopic cytology specimens[J]. Gut, 2018, 67: 1942-1949. DOI:10.1136/gutjnl-2017-314026 |
| [22] |
LI X, KLEEMAN S, COBURN S B, FUMAGALLI C, PERNER J, JAMMULA S, et al. Selection and application of tissue microRNAs for nonendoscopic diagnosis of Barrett's esophagus[J/OL]. Gastroenterology, 2018, 155: 771-778.e3. DOI: 10.1053/j.gastro.2018.05.050.
|
| [23] |
FITZGERALD R C, DI PIETRO M, O'DONOVAN M, MARONI R, MULDREW B, DEBIRAM-BEECHAM I, et al. Cytosponge-trefoil factor 3 versus usual care to identify Barrett's oesophagus in a primary care setting: a multicentre, pragmatic, randomised controlled trial[J]. Lancet, 2020, 396: 333-344. DOI:10.1016/S0140-6736(20)31099-0 |
| [24] |
ROTH M J, LIU S F, DAWSEY S M, ZHOU B, COPELAND C, WANG G Q, et al. Cytologic detection of esophageal squamous cell carcinoma and precursor lesions using balloon and sponge samplers in asymptomatic adults in Linxian, China[J]. Cancer, 1997, 80: 2047-2059. DOI:10.1002/(SICI)1097-0142(19971201)80:11<2047::AID-CNCR3>3.0.CO;2-U |
| [25] |
PAN Q J, ROTH M J, GUO H Q, KOCHMAN M L, WANG G Q, HENRY M, et al. Cytologic detection of esophageal squamous cell carcinoma and its precursor lesions using balloon samplers and liquid-based cytology in asymptomatic adults in Linxian, China[J]. Acta Cytologica, 2008, 52: 14-23. DOI:10.1159/000325430 |
| [26] |
GAO Y, XIN L, FENG Y D, YAO B, LIN H, SUN C, et al. Feasibility and accuracy of artificial intelligence-assisted sponge cytology for community-based esophageal squamous cell carcinoma screening in China[J]. Am J Gastroenterol, 2021, 116: 2207-2215. DOI:10.14309/ajg.0000000000001499 |
| [27] |
GAO Y, XIN L, LIN H, YAO B, ZHANG T, ZHOU A J, et al. Machine learning-based automated sponge cytology for screening of oesophageal squamous cell carcinoma and adenocarcinoma of the oesophagogastric junction: a nationwide, multicohort, prospective study[J/OL]. Lancet Gastroenterol Hepatol, 2023, 14: S2468-1253(23)00004-3. DOI: 10.1016/S2468-1253(23)00004-3.
|
| [28] |
JING J X, WANG Y, XU X Q, SUN T, TIAN B G, DU L L, et al. Tumor markers for diagnosis, monitoring of recurrence and prognosis in patients with upper gastrointestinal tract cancer[J]. Asian Pac J Cancer Prev, 2014, 15: 10267-10272. |
| [29] |
ZHANG J, ZHU Z, LIU Y, JIN X, XU Z, YU Q, et al. Diagnostic value of multiple tumor markers for patients with esophageal carcinoma[J/OL]. PLoS One, 2015, 10: e0116951. DOI: 10.1371/journal.pone.0116951.
|
| [30] |
ZHANG H, XIA J, WANG K, ZHANG J. Serum autoantibodies in the early detection of esophageal cancer: a systematic review[J]. Tumour Biol, 2015, 36: 95-109. DOI:10.1007/s13277-014-2878-9 |
| [31] |
XU Y W, PENG Y H, CHEN B, WU Z Y, WU J Y, SHEN J H, et al. Autoantibodies as potential biomarkers for the early detection of esophageal squamous cell carcinoma[J]. Am J Gastroenterol, 2014, 109: 36-45. DOI:10.1038/ajg.2013.384 |
| [32] |
CHU L Y, PENG Y H, WENG X F, XIE J J, XU Y W. Blood-based biomarkers for early detection of esophageal squamous cell carcinoma[J]. World J Gastroenterol, 2020, 26: 1708-1725. DOI:10.3748/wjg.v26.i15.1708 |
| [33] |
SUDO K, KATO K, MATSUZAKI J, BOKU N, ABE S, SAITO Y, et al. Development and validation of an esophageal squamous cell carcinoma detection model by large-scale microRNA profiling[J/OL]. JAMA Netw Open, 2019, 2: e194573. DOI: 10.1001/jamanetworkopen.2019.4573.
|
| [34] |
MIYOSHI J, ZHU Z, LUO A, TODEN S, ZHOU X, IZUMI D, et al. A microRNA-based liquid biopsy signature for the early detection of esophageal squamous cell carcinoma: a retrospective, prospective and multicenter study[J/OL]. Mol Cancer, 2022, 21: 44. DOI: 10.1186/s12943-022-01507-x.
|
| [35] |
YAO C, LIU H N, WU H, CHEN Y J, LI Y, FANG Y, et al. Diagnostic and prognostic value of circulating microRNAs for esophageal squamous cell carcinoma: a systematic review and meta-analysis[J]. J Cancer, 2018, 9: 2876-2884. DOI:10.7150/jca.25351 |
| [36] |
LI W, ZHENG J, DENG J, YOU Y, WU H, LI N, et al. Increased levels of the long intergenic non-protein coding RNA POU3F3 promote DNA methylation in esophageal squamous cell carcinoma cells[J/OL]. Gastroenterology, 2014, 146: 1714-1726.e5. DOI: 10.1053/j.gastro.2014.03.002.
|
| [37] |
TONG Y S, WANG X W, ZHOU X L, LIU Z H, YANG T X, SHI W H, et al. Identification of the long non-coding RNA POU3F3 in plasma as a novel biomarker for diagnosis of esophageal squamous cell carcinoma[J/OL]. Mol Cancer, 2015, 14: 3. DOI: 10.1186/1476-4598-14-3.
|
| [38] |
WANG W, HE X, ZHENG Z, MA X, HU X, WU D, et al. Serum HOTAIR as a novel diagnostic biomarker for esophageal squamous cell carcinoma[J/OL]. Mol Cancer, 2017, 16: 75. DOI: 10.1186/s12943-017-0643-6.
|
| [39] |
KAWAKAMI K, BRABENDER J, LORD R V, GROSHEN S, GREENWALD B D, KRASNA M J, et al. Hypermethylated APC DNA in plasma and prognosis of patients with esophageal adenocarcinoma[J]. J Natl Cancer Inst, 2000, 92: 1805-1811. DOI:10.1093/jnci/92.22.1805 |
| [40] |
HIBI K, TAGUCHI M, NAKAYAMA H, TAKASE T, KASAI Y, ITO K, et al. Molecular detection of p16 promoter methylation in the serum of patients with esophageal squamous cell carcinoma[J]. Clin Cancer Res, 2001, 7: 3135-3138. |
| [41] |
NICHOLSON J, LINDON J, HOLMES E. 'Metabonomics': understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data[J]. Xenobiotica, 1999, 29: 1181-1189. DOI:10.1080/004982599238047 |
| [42] |
LIU R, PENG Y, LI X, WANG Y, PAN E, GUO W, et al. Identification of plasma metabolomic profiling for diagnosis of esophageal squamous-cell carcinoma using an UPLC/TOF/MS platform[J]. Int J Mol Sci, 2013, 14: 8899-8911. DOI:10.3390/ijms14058899 |
| [43] |
MA H, HASIM A, MAMTIMIN B, KONG B, ZHANG H P, SHEYHIDIN I. Plasma free amino acid profiling of esophageal cancer using high-performance liquid chromatography spectroscopy[J]. World J Gastroenterol, 2014, 20: 8653-8659. DOI:10.3748/wjg.v20.i26.8653 |
| [44] |
MARKAR S R, WIGGINS T, ANTONOWICZ S, CHIN S T, ROMANO A, NIKOLIC K, et al. Assessment of a noninvasive exhaled breath test for the diagnosis of oesophagogastric cancer[J]. JAMA Oncol, 2018, 4: 970-976. DOI:10.1001/jamaoncol.2018.0991 |
| [45] |
ZOU X, ZHOU W, LU Y, SHEN C, HU Z, WANG H, et al. Exhaled gases online measurements for esophageal cancer patients and healthy people by proton transfer reaction mass spectrometry[J]. J Gastroenterol Hepatol, 2016, 31: 1837-1843. DOI:10.1111/jgh.13380 |
| [46] |
XIE Z, CHEN G, ZHANG X, LI D, HUANG J, YANG C, et al. Salivary microRNAs as promising biomarkers for detection of esophageal cancer[J/OL]. PLoS One, 2013, 8: e57502. DOI: 10.1371/journal.pone.0057502.
|
| [47] |
LI K, LIN Y, LUO Y, XIONG X, WANG L, DURANTE K, et al. A signature of saliva-derived exosomal small RNAs as predicting biomarker for esophageal carcinoma: a multicenter prospective study[J/OL]. Mol Cancer, 2022, 21: 21. DOI: 10.1186/s12943-022-01499-8.
|
| [48] |
吴伟东, 侯文进, 吴哲凡, 王跃军, 易永盛, 林苇嘉. 唾液中miRNA-144可作为早期诊断食道癌的基因标志物[J]. 南方医科大学学报, 2013, 33: 1783-1786. DOI:10.3969/j.issn.1673-4254.2013.12.16 |
| [49] |
叶敏华, 叶鹏辉, 张伟珠, 饶珈琦, 谢子钧. 唾液与血浆微小RNA-21对早期食管癌的诊断价值[J]. 南方医科大学学报, 2014, 34: 885-889. |
 2023, Vol. 44
2023, Vol. 44


