2. 海军军医大学(第二军医大学)国家肝癌科学中心, 上海 200438;
3. 海军军医大学(第二军医大学)第三附属医院国际合作生物信号转导研究室, 上海 200438
2. National Center for Liver Cancer, Naval Medical University (Second Military Medical University), Shanghai 200438, China;
3. International Cooperation Laboratory on Signal Transduction, The Third Affiliated Hospital of Naval Medical University (Second Military Medical University), Shanghai 200438, China
目前,二代测序(second-generation sequencing,NGS技术凭借其高通量、高准确性与低成本等优势在基因组测序市场占据主导地位。该技术将基因组打断为小片段,在对每个片段进行测序后生成一小段DNA序列,即读长(reads)[1]。但是,这种方法会导致基因组数据高度碎片化,产生不完整甚至错误的组装。
近年来,对读长的更高需求加速了长读长测序(long-read sequencing,LRS)技术的发展。与短读长测序技术相比,LRS技术无须将DNA片段化便可进行测序,能够跨越整个重复序列并实现连续和完整的组装。随着测序通量和准确性的提升,LRS技术可以测定上万至几兆碱基的连续序列,技术成熟度稳步提升。目前常使用的LRS技术主要有美国太平洋生物科技公司(Pacific Biosciences,PacBio)研发的单分子实时(single molecule real-time,SMRT)测序系统和牛津纳米孔科技有限公司(Oxford Nanopore Technologies,ONT)研发的纳米孔测序技术,它们均能跨越基因组中高重复性的区域,实现核酸分子更精确、完整的检测,从而识别基因组中大片段的变异和更多的转录本。
1 LRS技术原理 1.1 SMRT测序技术SMRT测序技术采用了一种拓扑环状的DNA分子模板,称为SMRTbell。该模板由两端带有单链发夹式接头的双链DNA插入物组成。组装好的SMRTbell与DNA聚合酶结合,并被装载到包含多达800万个零模波导孔(zero-mode waveguide,ZMW)的SMRT CELL芯片上[2]。在测序反应过程中,聚合酶围绕SMRTbell进行反应,用荧光标记的脱氧核苷三磷酸合成新生链。在每次掺入后,激光将会激发荧光基团,并利用相机记录荧光信号(图 1A)。
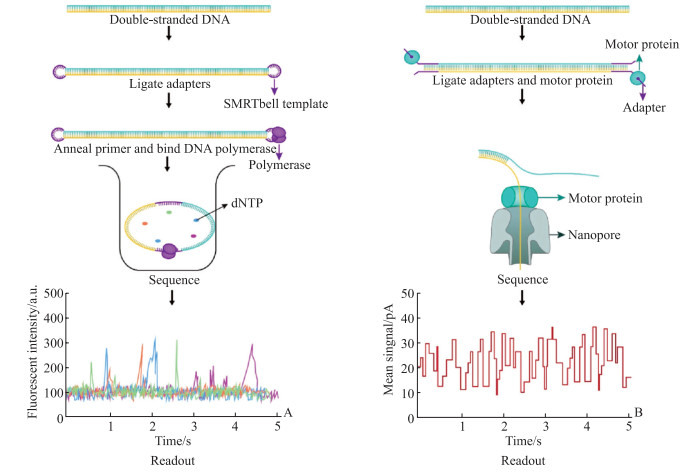
|
图 1 PacBio SMRT测序技术与ONT纳米孔测序技术工作原理 Fig 1 Schematics of PacBio SMRT and ONT nanopore sequencing procedure A: A schematic diagram of PacBio SMRT sequencing. A SMRTbell template consists of a double-stranded region flanked by 2 hairpin loops. The DNA template is replicated by polymerase chain reaction (PCR), then sequenced by PacBio SMRT. Bases can be identified by different fluorescence colors and intensities. B: A schematic diagram of ONT nanopore sequencing. A single-stranded DNA molecule is ligated with adapters and motor proteins so it can pass through the nanopore. Changes in electrical current or tunneling currents are used to read off the chain of bases. PacBio: Pacific Biosciences; SMRT: Single molecule real-time; ONT: Oxford Nanopore Technologies; SMRTbell: Single molecule real-time bell; dNTP: Deoxyribonucleoside triphosphate; a.u.: Absorbance unit. |
2019年,PacBio开发了循环共识测序(circular consensus sequencing,CCS),该方法通过从单个模板分子的多次检测中得出共有序列,从而实现高碱基准确度[3],在长度约为13 kb的序列中碱基准确度超过99%。但该测序过程需要约10 µg DNA样本,在检测样本量有限的微小肿瘤或早期肿瘤中应用困难。此外,这种测序方式虽然准确性较高,但是并未摆脱对光学系统的依赖;同时由于边合成边测序的基本技术原理,仪器的体积比较大,搬运和携带仍然比较困难。
1.2 纳米孔测序技术2012年,ONT公布了首个高通量测序平台MinION。该测序平台先将测序接头与马达蛋白连接于双链DNA分子上,当双链DNA分子进入嵌在合成生物膜上的纳米孔后,马达蛋白即解开DNA双链,带负电荷的DNA在电场力与马达蛋白作用下以可控的速率通过纳米孔。DNA分子在纳米孔中穿过时会造成电流的扰动,通过记录电流序列变化,根据电子信号产生的差异识别不同的碱基,从而实现实时分析DNA链中的序列;该技术建库起始量低至2 µg[4](图 1B)。
MinION和PromethION是ONT最常用的2种纳米孔测序仪,MinION是一种纳米孔型便携式测序仪,最初商业化设备的读取长度约为10 kb,单个流通池(flowcell)的吐量约为5 Gb[5]。PromethION增加了每个流通池的孔数,并支持运行多个流通池,最多可同时运行48个流通池,测序数据量可超过100 Gb[6]。基于纳米孔测序原理的测序仪具有设备体积小巧、便携性好、适用于条件受限的特殊场景等特点。
近年来,国产纳米孔测序仪也逐步走向市场。齐碳科技有限公司推出了国内首个具有完全自主知识产权的纳米孔测序仪QNome-3841,该产品实现了生物芯片、集成电路等核心组件100% 国产化。今是科技有限公司推出了国内首款中通量纳米孔测序仪Gseq500,单芯片测序单元数量达到50万个。此外,普译生物科技有限公司正在进行具有自主知识产权的纳米孔蛋白优化以及中高通量纳米孔基因测序仪的产品开发。
1.3 新型单分子LRS技术上述2种测序技术是目前较为成熟的LRS技术,随着研究人员对测序原理的改进,新的能够分析经过修饰的核酸分子的单分子LRS技术应运而生。
单管长片段测序(single-tube long fragment read sequencing,stLFR-seq)是由华大智造科技股份有限公司自主研发的基于DNBSEQ测序平台的一种长片段读取技术。stLFR-seq利用Tn5转座酶将带有分子标签的杂交序列插入DNA中,以对每个长DNA分子进行编码,实现读取长度达10 000~300 000 bp的序列;该技术建库起始量低至1 ng[7](图 2A)。

|
图 2 新型单分子LRS技术的工作原理 Fig 2 Workflow of novel single-molecule LRS technologies A: Workflow of stLFR-seq. The Tn5 transposase inserts a hybridized sequence with a molecular tag into the DNA to encode each long DNA molecule. Then PCR products are collected and quantified, followed by MGISEQ-2000 DNB-based library preparation and sequencing. B: Workflow of TELL-seq. TELL beads and barcodes are used to fragment DNA, and then Illumina sequencing is performed after PCR amplification and library construction. C: Workflow of scNanoATAC-seq. Unfragmented DNA is tagged by Tn5 transposase. Single nuclei are sorted into lysis buffer containing inner barcode. After performing PCR with outer barcode primers, long reads are sequenced by ONT PromethION 48. LRS: Long-read sequencing; stLFR-seq: Single-tube long fragment read sequencing; TELL-seq: Transposase enzyme-linked long-read sequencing; scNanoATAC-seq: Single-cell assay for transposase-accessible chromatin on nanopore sequencing platform; PCR: Polymerase chain reaction; DNB: DNA nanoball; ONT: Oxford Nanopore Technologies. |
转座酶关联长读长测序(transposase enzyme-linked long-read sequencing,TELL-seq)是一种使用NGS技术获取远程区域信息的方法[8],该方法使用转座酶来片段化DNA并添加条形码,帮助NGS技术重新组装DNA片段,让短读长数据产生超LRS效果,等价读长可达20 000~200 000 bp(图 2B)。与PacBio和ONT测序方法相比,TELL-seq的碱基准确度更高,所需的DNA样本量更少,人类基因组DNA仅需5 ng。但TELL-seq需要根据目标基因组进行复杂的生物信息学分析,以对获得的碎片信息进行精确注释。
通过NGS平台使用测序技术对转座酶可及性染色质进行单细胞测定的技术,即scATAC-seq(single-cell assay for transposase-accessible chromatin using sequencing),是一种成熟的对单个细胞内开放染色质区域进行检测的方法。然而,由于基于NGS技术,scATAC-seq对检测基因组结构变异存在困难。北京大学汤富酬团队开发了一种基于LRS技术的转座酶可及性染色质单细胞测定方法,即scNanoATAC-seq(single-cell assay for transposase-accessible chromatin on nanopore sequencing platform)[9],该方法可以实现同时检测单个细胞内的可及性染色质和结构变异(图 2C)。
由于测序方式和DNA样品制备的差异,每种LRS技术均可以产生不同类型的长读长数据,它们的长度和准确性都有差异。表 1总结了长读长数据集的特征,并就每种测序数据类型的相对长度和精度等与Illumina NGS数据进行了比较[10-13]。
2 LRS技术在肿瘤研究中的应用 2.1 基因组变异检测 2.1.1 结构变异检测
结构变异是肿瘤基因组不稳定性的重要标志,包括易位、大片段缺失、扩增和倒位。结构变异通常是通过驱动易位和扩增使癌基因激活,驱动缺失和倒位使抑癌基因失活。因此高灵敏检测肿瘤特异性突变,包括小的变异(如单碱基替换)及大的变异(如结构变异),对于指导肿瘤患者早期检测、复发监测和靶向治疗等至关重要。
(1)基于LRS检测癌细胞基因组的结构变异:NGS的诞生为研究基因组微小变异提供了便利工具,但对于较大结构变异,NGS技术的短片段(<300 bp)检测仍有很大的局限性。重复区域(包括着丝粒、端粒和其他重复元件)包含人类基因组一半以上的序列(56%),单端测序在不同位置重复出现的序列片段中识别出相同的信息会导致该序列比对时产生歧义[14],而LRS(测序长度达20 000 bp)可以跨越这些重复区域进行片段识别。
针对结构变异的LRS策略通常分为3种:全面覆盖测序、混合覆盖测序和混合测序[15]。全面覆盖测序方法指所有样本都以中到高覆盖率进行测序;混合覆盖测序方法指一部分样本以中到高覆盖率进行测序,其余样本使用低覆盖率进行测序;混合测序方法指一部分样本以中到高覆盖率进行测序,其余样本进行短读长测序。不同策略对群体中常见或罕见变异的检测能力会产生影响,具体见表 2。
|
|
表 2 不同LRS策略对结构变异检测能力的影响 Tab 2 Impact of different LRS strategies on ability to detect structure variation |
Hu等[16]和Aganezov等[17]利用ONT和PacBio平台对乳腺癌样本进行了全基因组测序。研究结果表明,即使在相对较低的覆盖率(25~30倍)下,LRS不仅可以检测到单核苷酸变异区域,也可以实现更准确且灵敏的结构变异检测,并且不同LRS方式产出结果的一致率达90%~95%。
利用LRS技术检测结构变异在临床应用中也有巨大潜力。研究人员利用MinION技术评估了胰腺导管腺癌细胞系的结构变异类型,包括单纯的缺失、易位、倒位及易位和倒位的组合,也对1∶100稀释度的PCR扩增产物进行检测,结果提示MinION技术对检测患者的特异性结构变异具有可行性,且每个样本的测序深度仅需要500读长即可快速得到检测结果[18]。
(2)基于LRS的基因组结构变异快速鉴定可用于癌症监测:循环肿瘤DNA(circulating tumor DNA,ctDNA)来源于肿瘤细胞,已被证明与肿瘤负荷呈线性正相关,且在癌症复发出现临床症状前几个月即可被检出[19]。液体活检可用于检测体液(如血液)中的肿瘤成分,是一种对患者损伤小、可接受度高的肿瘤负荷动态监测方式[20]。在液体活检中检测ctDNA中的代表性结构变异,可用于高灵敏度检测和量化ctDNA[21],因此,准确、及时地检测血液中包括结构变异在内的肿瘤相关改变对于患者从早期预警到监测复发均具有重要作用。鉴于测序速度快、体积小和成本低等优点,LRS可能成为早诊、复发或疗效监测过程中癌症相关结构变异检测的理想方式。
为了能够快速和高效地识别一组特异性体细胞结构变异,用于ctDNA监测,Valle-Inclan等[22]利用纳米孔测序的长读长和快速测序能力开发出一种能够准确选择纳米孔测序数据体细胞结构变异模型的算法,该算法仅用3 d时间即可检出卵巢癌和前列腺癌患者体细胞结构变异,同时,应用该算法的DNA样本需求量可以进一步减少到80 ng。
2.1.2 拷贝数变化(copy number alteration,CNA)检测CNA是人类基因组变异的一种常见形式,已被证实与许多病理变化相关,包括罕见的基因组疾病[23]、神经系统疾病[24]和癌症[25]。在癌症中,体细胞CNA促进了基因组的变异,具有诊断、预后和治疗意义[26]。CNA信息可通过染色体分析、核型分析及荧光原位杂交[27]等方法检测,然而,这些方法均存在灵敏度或准确度不高等局限性。NGS能够从短读长测序数据中显示出高准确度CNA信息来提高灵敏度和准确度[28],但此类方法成本较高,应用场所局限于大型、资源丰富的临床中心。
Martignano等[29]利用低覆盖率纳米孔测序检测来自癌症患者的血浆核酸分子的CNA,仅用200万读数就完成了对6例肺癌患者和4名健康受试者的全基因组分子核型检测,并发现了常见的与肺癌相关的CNA。这是纳米孔测序成功应用于血浆DNA拷贝数分析的案例,展现出LRS对患者高效、无创检测的潜力。
2.1.3 基因融合检测基因融合是由于染色体倒位、串联重复、区段缺失或易位事件造成不同的、独立的基因或基因片段发生合并的过程,在癌症中较为常见。例如,慢性髓细胞性白血病中可检出BCR-ABL(BCR activator of RhoGEF and GTPase-ABL tyrosine kinase)融合,导致费城染色体(Philadelphia chromosome,Ph)形成[30];肺腺癌患者的肿瘤组织中EML4-ALK(EMAP-like 4-ALK receptor tyrosine kinase)融合基因编码的融合蛋白具有致癌作用[31-32]。因此检测融合基因并阐明其形成的机制和演化过程对于肿瘤研究具有重要价值。免疫组织化学和荧光原位杂交是临床检测融合基因的2种主要方法[33]。这2种方法用于分析单个基因,且对于组织样本有一定的要求,在临床实践中可行性低且耗时。因此,LRS技术作为能够以最少的样本量测试多个基因的新技术,可助力融合基因的快速发现。
Jeck等[34]利用ONT MinION测序系统,通过修改锚定多重PCR方法进行文库构建,在测序15 min内获得>100读长,可实现BCR-ABL融合转录本的检测。在Romagnoli等[35]的研究中,LRS技术能在60 h内鉴定出嗜酸性粒细胞增多症患者循环血DNA样本中的血小板源性生长因子受体α/β、成纤维细胞生长因子受体1和Janus激酶2基因重排,该结果与诊断时独立进行的荧光原位杂交分析结果完全一致。同时,测序结果为识别这几例患者基因融合位点提供了理论依据,这是传统方法无法提供的信息。
2.2 全长RNA转录本检测及转录本异构体和lncRNA识别一些转录亚型的异常表达可能会激活癌基因或使抑癌基因失活,从而导致癌症发生[36]。在RNA水平上研究癌细胞的分子特征很大程度上依赖于参考转录组的精确度和完整性。然而,由于传统Illumina短读长(≤150 bp)的限制,通常无法捕捉到转录本中的所有外显子,导致一些新的转录亚型无法识别[37]。Fang等[38]对3例肝细胞癌患者的肿瘤组织、门静脉癌栓和癌旁组织进行了纳米孔转录组RNA测序。研究结果发现,短读长测序的转录本组装可能会出现许多错误,而LRS在识别结构复杂的长片段新转录本方面具有明显的优势。此外,其他研究者也使用LRS成功地发现了细胞中大量新的转录本[39-41]。以上研究结果表明,LRS是准确组装转录本的有效工具,并且相较于NGS技术能够识别更多未知的转录本。
lncRNA参与基因组修饰、转录激活、转录干扰、核内运输等过程[42],其在肿瘤发生与发展过程中的调控作用已被大量研究证实。lncRNA会因细胞类型、组织类型和物种类型的不同产生多种同工型。短读长测序可揭示lncRNA表达差异,但表达差异不足以代表其作用方式,因此LRS技术为探索lncRNA不同加工方式及生物学功能提供了有力工具[43]。为了加速lncRNA注释,GENCODE联盟开发了RNA捕获长测序(RNA capture long-read sequencing,CLS),它将靶向RNA捕获与LRS相结合,所生成的全长转录本模型能够明确描述lncRNA的基因组特征,包括启动子和基因结构及蛋白质编码潜能[44]。
2.3 表观遗传修饰识别表观遗传学在驱动癌症及其异质性方面具有重要作用,基因表达的表观遗传调控主要依赖于DNA甲基化和组蛋白修饰[45]。迄今为止,研究DNA甲基化通常先利用亚硫酸氢盐处理,将胞嘧啶转化为尿嘧啶进而将表观遗传信息转化为遗传信息,再进行大规模平行DNA测序。然而,基于亚硫酸氢盐处理的测序无法区分胞嘧啶、5-甲基胞嘧啶和5-羟甲基胞嘧啶,这增加了文库制备的复杂性及来自不完全化学转化等偏差的可能性[46]。此外,常用的NGS技术存在读取长度短的限制,使得一些区域难以定位,无法进行等位基因特异性甲基化的研究。Rand等[47]和Simpson等[48]首次使用纳米孔测序实现了全基因组DNA甲基化分析。Ewing等[49]利用ONT测序技术实现了转座元件表观基因组的全面分析,同时评估转座元件的甲基化水平和检测体细胞转座因子的动员情况。此后,研究者们开发及改进了基于LRS技术对表观遗传景观进行建模和分析的工具[50-51],将现有DNA甲基化检测技术向前推进了一步。
3 小结与展望LRS技术助力人类癌细胞基因组及全转录组图谱绘制,使肿瘤患者游离DNA检测、大片段结构变异和甲基化等快速检测成为可能,为临床快速筛选、疾病诊断及预后判断提供了有力依据。
LRS技术处于起步阶段,仍然存在一些尚待解决的问题,首要挑战是对最低样本量的需求。相对NGS,LRS技术对检测DNA或RNA的质量和数量要求更低,但仍需要微克级DNA来制备用于全基因组测序的文库,特别是临床血液及体液样本提取的DNA量通常很有限,因此,LRS应用于临床还需要进一步减少对样本量的需求。另一个挑战则是ONT和PacBio测序存在较高的原始错误率及长扩增子嵌合体形成率,有待于通过升级优化检测平台和下游分析流程规避可能的错误。例如,ONT发布的PromethION流通池10.3版本的测序准确度远高于之前的版本,在单读时测序准确度可达到99%,有助于改善当前长读测序碱基准确度低的不足,进一步帮助改进下游分析。此外,针对LRS数据的下游分析,如重复序列比对、基因组结构变异检测和甲基化分析等依赖人工智能算法、软件和数据库等生物信息学工具。目前已有的针对NGS数据开发的算法无法解决LRS数据高原始错误率的问题,因此开发更适合处理长序列片段数据的算法有助于LRS技术准确度的提升。
LRS技术在肿瘤研究中的应用思路和探索领域正在不断扩展,特别是针对一些具有高突变率的肿瘤,如血液肿瘤中的各类白血病及多发性骨髓瘤、恶性淋巴瘤等,LRS技术可通过以下应用方向发挥重要作用:(1)检测肿瘤早期的基因突变,对癌症进行早期预警;(2)根据分子标志物对疾病进行鉴别诊断及分子分型;(3)根据患者的基因变异选择合适的靶向药,做到精准治疗;(4)利用基因变异导致患者对化疗药物耐受性的不同进行化疗相关不良反应预测;(5)利用LRS技术检测微小残留灶进行疗效判断及复发监测,进一步提高分子诊断在肿瘤早期诊断、精准治疗、判断疗效、复发预测等全程管理中的重要应用价值。
LRS技术尽管仍然存在诸多亟待解决的问题,其仍是测序技术更新换代的重要里程碑。相信随着技术的革新,未来LRS技术将加速肿瘤基因组学及表观遗传组学研究,窥探既往难以发现的分子生物学信息,为新的肿瘤生物标志物识别及诊疗方式开发提供新的思路。
| [1] |
SENOL CALI D, KIM J S, GHOSE S, ALKAN C, MUTLU O. Nanopore sequencing technology and tools for genome assembly: computational analysis of the current state, bottlenecks and future directions[J]. Brief Bioinform, 2019, 20: 1542-1559. DOI:10.1093/bib/bby017 |
| [2] |
KORLACH J, BJORNSON K P, CHAUDHURI B P, CICERO R L, FLUSBERG B A, GRAY J J, et al. Real-time DNA sequencing from single polymerase molecules[J]. Methods Enzymol, 2010, 472: 431-455. |
| [3] |
WENGER A M, PELUSO P, ROWELL W J, CHANG P C, HALL R J, CONCEPCION G T, et al. Accurate circular consensus long-read sequencing improves variant detection and assembly of a human genome[J]. Nat Biotechnol, 2019, 37: 1155-1162. DOI:10.1038/s41587-019-0217-9 |
| [4] |
B O W D E N R , D AV I E S R W, H E G E R A ,
PAGNAMENTA A T, DE CESARE M, OIKKONEN L E,
et al. Sequencing of human genomes with nanopore
technology[J/OL]. Nat Commun, 2019, 10: 1869.
10.1038/s41467-019-09637-5..
|
| [5] |
JAIN M, FIDDES I T, MIGA K H, OLSEN H E, PATEN B, AKESON M. Improved data analysis for the MinION nanopore sequencer[J]. Nat Methods, 2015, 12: 351-356. DOI:10.1038/nmeth.3290 |
| [6] |
JAIN M, KOREN S, MIGA K H, QUICK J, RAND A C, SASANI T A, et al. Nanopore sequencing and assembly of a human genome with ultra-long reads[J]. Nat Biotechnol, 2018, 36: 338-345. DOI:10.1038/nbt.4060 |
| [7] |
WANG O, CHIN R, CHENG X F, WU M K Y, MAO Q, TANG J B, et al. Efficient and unique cobarcoding of second-generation sequencing reads from long DNA molecules enabling cost-effective and accurate sequencing, haplotyping, and de novo assembly[J]. Genome Res, 2019, 29: 798-808. DOI:10.1101/gr.245126.118 |
| [8] |
CHEN Z, PHAM L, WU T C, MO G, XIA Y, CHANG P L, et al. Ultralow-input single-tube linked-read library method enables short-read second-generation sequencing systems to routinely generate highly accurate and economical long-range sequencing information[J]. Genome Res, 2020, 30: 898-909. DOI:10.1101/gr.260380.119 |
| [9] |
HU Y Q, JIANG Z H, CHEN K X, ZHOU Z X, ZHOU X, WANG Y, et al. scNanoATAC-seq: a long-read single-cell ATAC sequencing method to detect chromatin accessibility and genetic variants simultaneously within an individual cell[J]. Cell Res, 2023, 33: 83-86. |
| [10] |
KARST S M, ZIELS R M, KIRKEGAARD R H, SØRENSEN E A, MCDONALD D, ZHU Q Y, et al. High-accuracy long-read amplicon sequences using unique molecular identifiers with Nanopore or PacBio sequencing[J]. Nat Methods, 2021, 18: 165-169. DOI:10.1038/s41592-020-01041-y |
| [11] |
TEDERSOO L, ALBERTSEN M, ANSLAN S, CALLAHAN
B. Perspectives and benefits of high-throughput long-read
sequencing in microbial ecology[J/OL] . Appl Environ
Microbiol, 2021, 87: e0062621.
|
| [12] |
WANG Y H, ZHAO Y, BOLLAS A, WANG Y R, AU K F. Nanopore sequencing technology, bioinformatics and applications[J]. Nat Biotechnol, 2021, 39: 1348-1365. DOI:10.1038/s41587-021-01108-x |
| [13] |
FOOX J, TIGHE S W, NICOLET C M, ZOOK J M, BYRSKA-BISHOP M, CLARKE W E, et al. Performance assessment of DNA sequencing platforms in the ABRF Next-Generation Sequencing Study[J]. Nat Biotechnol, 2021, 39: 1129-1140. DOI:10.1038/s41587-021-01049-5 |
| [14] |
SEDLAZECK F J, RESCHENEDER P, SMOLKA M, FANG H, NATTESTAD M, VON HAESELER A, et al. Accurate detection of complex structural variations using single-molecule sequencing[J]. Nat Methods, 2018, 15: 461-468. DOI:10.1038/s41592-018-0001-7 |
| [15] |
DE COSTER W, WEISSENSTEINER M H, SEDLAZECK F J. Towards population-scale long-read sequencing[J]. Nat Rev Genet, 2021, 22: 572-587. DOI:10.1038/s41576-021-00367-3 |
| [16] |
HU T B, LI J J, LONG M P, WU J B, ZHANG Z, XIE F,
et al. Detection of structural variations and fusion
genes in breast cancer samples using third-generation
sequencing[J/OL] . Front Cell Dev Biol, 2022, 10:
854640.
10.3389/fcell.2022.854640..
|
| [17] |
AGANEZOV S, GOODWIN S, SHERMAN R M, SEDLAZECK F J, ARUN G, BHATIA S, et al. Comprehensive analysis of structural variants in breast cancer genomes using single-molecule sequencing[J]. Genome Res, 2020, 30: 1258-1273. DOI:10.1101/gr.260497.119 |
| [18] |
NORRIS A L, WORKMAN R E, FAN Y F, ESHLEMAN J R, TIMP W. Nanopore sequencing detects structural variants in cancer[J]. Cancer Biol Ther, 2016, 17: 246-253. DOI:10.1080/15384047.2016.1139236 |
| [19] |
SCHWARZENBACH H, HOON D S B, PANTEL K. Cell-free nucleic acids as biomarkers in cancer patients[J]. Nat Rev Cancer, 2011, 11: 426-437. DOI:10.1038/nrc3066 |
| [20] |
HEITZER E, HAQUE I S, ROBERTS C E S, SPEICHER M R. Current and future perspectives ofliquid biopsies in genomics-driven oncology[J]. Nat Rev Genet, 2019, 20: 71-88. DOI:10.1038/s41576-018-0071-5 |
| [21] |
OLSSON E, WINTER C, GEORGE A, CHEN Y L, HOWLIN J, TANG M H E, et al. Serial monitoring of circulating tumor DNA in patients with primary breast cancer for detection of occult metastatic disease[J]. EMBO Mol Med, 2015, 7: 1034-1047. DOI:10.15252/emmm.201404913 |
| [22] |
VALLE-INCLAN J E, STANGL C, DE JONG A C, VAN
DESSEL L F, VAN ROOSMALEN M J, HELMIJR
J C A, et al. Optimizing nanopore sequencing-based
detection of structural variants enables individualized
circulating tumor DNA-based disease monitoring in
cancer patients[J/OL] . Genome Med, 2021, 13: 86.
DOI: .
10.1186/s13073-021-00899-7.
|
| [23] |
ANGULO M A, BUTLER M G, CATALETTO M E. Prader-Willi syndrome: a review of clinical, genetic, and endocrine findings[J]. J Endocrinol Invest, 2015, 38: 1249-1263. DOI:10.1007/s40618-015-0312-9 |
| [24] |
STEFANSSON H, RUJESCU D, CICHON S, PIETIL INEN O P H, INGASON A, STEINBERG S, et al. Large recurrent microdeletions associated with schizophrenia[J]. Nature, 2008, 455: 232-236. DOI:10.1038/nature07229 |
| [25] |
SHLIEN A, MALKIN D. Copy number variations and
cancer [J/OL]. Genome Med, 2009, 1: 62.
|
| [26] |
D HNER H, WEI A H, APPELBAUM F R, CRADDOCK C, DINARDO C D, DOMBRET H, et al. Diagnosis and management of AML in adults: 2022 recommendations from an international expert panel on behalf of the ELN[J]. Blood, 2022, 140: 1345-1377. DOI:10.1182/blood.2022016867 |
| [27] |
GONZALEZ GARCIA J R, MEZA-ESPINOZA J P. Use of the International System for Human Cytogenetic Nomenclature (ISCN)[J]. Blood, 2006, 108: 3952-3953. |
| [28] |
ZEHIR A, BENAYED R, SHAH R H, SYED A, MIDDHA S, KIM H R, et al. Mutational landscape of metastatic cancer revealed from prospective clinical sequencing of 10, 000 patients[J]. Nat Med, 2017, 23: 703-713. DOI:10.1038/nm.4333 |
| [29] |
MARTIGNANO F, MUNAGALA U, CRUCITTA S,
MINGRINO A, SEMERARO R, DEL RE M, et al.
Nanopore sequencing from liquid biopsy: analysis of
copy number variations from cell-free DNA of lung
cancer patients[J/OL]. Mol Cancer, 2021, 20: 32.
10.1186/s12943-021-01327-5.
|
| [30] |
MITELMAN F, JOHANSSON B, MERTENS F. The impact of translocations and gene fusions on cancer causation[J]. Nat Rev Cancer, 2007, 7: 233-245. DOI:10.1038/nrc2091 |
| [31] |
SODA M, CHOI Y L, ENOMOTO M, TAKADA S, YAMASHITA Y, ISHIKAWA S, et al. Identification of the transforming EML4-ALK fusion gene in non-small-cell lung cancer[J]. Nature, 2007, 448: 561-566. DOI:10.1038/nature05945 |
| [32] |
SODA M, TAKADA S, TAKEUCHI K, CHOI Y L, ENOMOTO M, UENO T, et al. A mouse model for EML4-ALK-positive lung cancer[J]. Proc Natl Acad Sci USA, 2008, 105: 19893-19897. DOI:10.1073/pnas.0805381105 |
| [33] |
PISAPIA P, PEPE F, SGARIGLIA R, NACCHIO M, RUSSO G, GRAGNANO G, et al. Methods for actionable gene fusion detection in lung cancer: now and in the future[J]. Pharmacogenomics, 2021, 22: 833-847. DOI:10.2217/pgs-2021-0048 |
| [34] |
JECK W R, LEE J, ROBINSON H, LE L P, IAFRATE A J, NARDI V. A nanopore sequencing-based assay for rapid detection of gene fusions[J]. J Mol Diagn, 2019, 21: 58-69. DOI:10.1016/j.jmoldx.2018.08.003 |
| [35] |
ROMAGNOLI S, BARTALUCCI N, GESULLO F,
BALLIU M, BONIFACIO S, FERNANDEZ A G L,
et al. Nanopore sequencing for the screening of
myeloid and lymphoid neoplasms with eosinophilia
and rearrangement of PDGFRα, PDGFRβ, FGFR1 or
PCM1-JAK2[J/OL]. Biomark Res, 2021, 9: 83.
10.1186/s40364-021-00337-1.
|
| [36] |
CHEN H, GAO F, HE M, DING X F, WONG A M, SZE S C, et al. Long-read RNA sequencing identifies alternative splice variants in hepatocellular carcinoma and tumor-specific isoforms[J]. Hepatology, 2019, 70: 1011-1025. DOI:10.1002/hep.30500 |
| [37] |
STEIJGER T, ABRIL J F, ENGSTR M P G, KOKOCINSKI F, RGASP Consortium, HUBBARD T J, et al. Assessment of transcript reconstruction methods for RNA-seq[J]. Nat Methods, 2013, 10: 1177-1184. DOI:10.1038/nmeth.2714 |
| [38] |
FANG Y C, CHEN G, CHEN F, HU E, DONG X Q, LI Z L, et al. Accurate transcriptome assembly by Nanopore RNA sequencing reveals novel functional transcripts in hepatocellular carcinoma[J]. Cancer Sci, 2021, 112: 3555-3568. DOI:10.1111/cas.15058 |
| [39] |
KUO R I, CHENG Y Y, ZHANG R X, BROWN J W S,
SMITH J, ARCHIBALD A L, et al. Illuminating the
dark side of the human transcriptome with long read
transcript sequencing[J/OL]. BMC Genomics, 2020, 21:751.
10.1186/s12864-020-07123-7.
|
| [40] |
BOTI M A, ADAMOPOULOS P G, TSIAKANIKAS P,
SCORILAS A. Nanopore sequencing unveils diverse
transcript variants of the epithelial cell-specific
transcription factor Elf-3 in human malignancies[J/OL].
Genes, 2021, 12: 839.
10.3390/genes12060839.
|
| [41] |
DE JONG L C, CREE S, LATTIMORE V, WIGGINS
G A R, SPURDLE A B, MILLER A, et al. Nanopore
sequencing of full-length BRCA1 mRNA transcripts reveals
co-occurrence of known exon skipping events[J/OL].
Breast Cancer Res, 2017, 19: 127.
|
| [42] |
CHEN L L. Towards higher-resolution and in vivo understanding of lncRNA biogenesis and function[J]. Nat Methods, 2022, 19: 1152-1155. DOI:10.1038/s41592-022-01626-9 |
| [43] |
STATELLO L, GUO C J, CHEN L L, HUARTE M. Gene regulation by long non-coding RNAs and its biological functions[J]. Nat Rev Mol Cell Biol, 2021, 22: 96-118. DOI:10.1038/s41580-020-00315-9 |
| [44] |
LAGARDE J, USZCZYNSKA-RATAJCZAK B, CARBONELL S, PÉREZ-LLUCH S, ABAD A, DAVIS C, et al. High-throughput annotation of full-length long noncoding RNAs with capture long-read sequencing[J]. Nat Genet, 2017, 49: 1731-1740. DOI:10.1038/ng.3988 |
| [45] |
GUO M Z, PENG Y J, GAO A A, DU C, HERMAN J G.
Epigenetic heterogeneity in cancer [J/OL]. Biomark Res,
2019, 7: 23.
10.1186/s40364-019-0174-y.
|
| [46] |
SCHATZ M C. Nanopore sequencing meets epigenetics[J]. Nat Methods, 2017, 14: 347-348. DOI:10.1038/nmeth.4240 |
| [47] |
RAND A C, JAIN M, EIZENGA J M, MUSSELMAN-BROWN A, OLSEN H E, AKESON M, et al. Mapping DNA methylation with high-throughput nanopore sequencing[J]. Nat Methods, 2017, 14: 411-413. DOI:10.1038/nmeth.4189 |
| [48] |
SIMPSON J T, WORKMAN R E, ZUZARTE P C, DAVID M, DURSI L J, TIMP W. Detecting DNA cytosine methylation using nanopore sequencing[J]. Nat Methods, 2017, 14: 407-410. DOI:10.1038/nmeth.4184 |
| [49] |
EWING A D, SMITS N, SANCHEZ-LUQUE F J,
FAIVRE J, BRENNAN P M, RICHARDSON S R,
et al. Nanopore sequencing enables comprehensive
transposable element epigenomic profiling[J/OL]. Mol
Cell, 2020, 80: 915-928.e5.
|
| [50] |
ABANTE J, KAMBHAMPATI S, FEINBERG A P,
GOUTSIAS J. Estimating DNA methylation potential
energy landscapes from nanopore sequencing data[J/OL].
Sci Rep, 2021, 11: 21619.
|
| [51] |
SAKAMOTO Y, ZAHA S, NAGASAWA S, MIYAKE S,
KOJIMA Y, SUZUKI A, et al. Long-read whole-genome
methylation patterning using enzymatic base conversion
and nanopore sequencing[J/OL]. Nucleic Acids Res,
2021, 49: e81.
10.1093/nar/gkab397.
|
 2023, Vol. 44
2023, Vol. 44


