在肿瘤细胞中,DNA复制、RNA合成和加工等重要的生物学过程在细胞核内完成,这些生物学过程依赖于诸如核散斑、Cajal小体、核仁及核旁斑等核内亚细胞器的调控。这些核内亚细胞器一般是由RNA和蛋白质组成的亚细胞结构。核旁斑因大多分布于核散斑附近的染色质间区域,故而得名。它是由结构性RNA核富集常染色体转录本1(nuclear enriched autosomal transcript 1,NEAT1)和多个RNA结合蛋白在空间上有序组装而成的大型核糖核蛋白复合物[1]。研究已证实,肿瘤细胞核旁斑可充当“分子海绵”以调节核质中活性分子水平[2]。从机制上说,肿瘤细胞核旁斑通过扣押RNA或蛋白以及与miRNA相互作用等方式影响肿瘤相关基因的转录、剪接、RNA稳定性和翻译等过程,进而调控肿瘤的发生、发展[3-4],因此研究核旁斑在肿瘤细胞中的生物学作用对肿瘤的诊疗具有十分重要的意义,而肿瘤细胞核旁斑的结构及功能目前并未完全明确。本文将从肿瘤细胞核旁斑的组成、结构、装配、功能、作用机制及临床意义等几个方面进行综述,旨在为肿瘤的诊断、治疗和预后提供新的研究方向和线索。
1 肿瘤细胞核旁斑的组成组成肿瘤细胞核旁斑的结构性RNA是NEAT1[5],属于lncRNA。NEAT1在人类基因组中定位于第11号染色体(11q13.1),它的2个转录本NEAT1_1(短转录本,人3.7 kb;鼠3.2 kb)和NEAT1_2(长转录本,人22.7 kb;鼠20 kb)均由RNA聚合酶Ⅱ转录而来,它们共用同一启动子和转录起始位点,并且5' 端序列完全一致[6]。长转录本NEAT1_2的3' 端经由核糖核酸酶P处理后,可形成一个独特的3' 端三重螺旋(3' triple helix,3' TH)结构,该结构是稳定NEAT1_2的关键[7]。短转录本NEAT1_1是由长转录本NEAT1_2经多聚腺苷酸化及加工修饰处理形成的,在其3' 端可形成典型的多腺苷酸化信号(polyadenylation signal,PAS)[8]。
NEAT1是形成核旁斑的骨架,一般来说,它的表达量越高肿瘤细胞内核旁斑的数量越多,相反,干扰NEAT1的表达能使得核旁斑的数量减少。NEAT1的2个转录本在核旁斑的形成中发挥着不同的功能[9]。长转录本NEAT1_2是组成核旁斑不可缺少的成分,敲除NEAT1_2后核旁斑将无法形成。NEAT1_2在空间上分为3个作用不同的功能域:功能域A(5' 端1 kb及3' TH区)、功能域B(2.1~2.8 kb,4.0~5.1 kb)、功能域C(中间8~16.6 kb),功能域C还可以进一步分为3个亚区:C1(9.8~12 kb)、C2(12~13 kb)、C3(15.4~16.6 kb)。功能域A、B、C分别发挥稳定自身结构、转录本剪切及核旁斑装配的功能[1, 10]。短转录本NEAT1_1尽管不是组成核旁斑必要的成分,但它在特定情况下可以调控核旁斑的形成速度。另外,在没有NEAT1_2时,NEAT1_1可形成靠近核散斑的“微斑点”(microspeckle),但关于微斑点的结构及功能目前尚不明确。
组成核旁斑的RNA结合蛋白大约有40多种,根据蛋白的缺失造成核旁斑的破坏程度可将组成核旁斑的RNA结合蛋白大致分为3类[11-12](表 1)。
|
|
表 1 肿瘤细胞核旁斑蛋白的分类 |
第Ⅰ类蛋白质对维持核旁斑的结构不可或缺,缺乏此类蛋白质核旁斑将无法形成,根据是否影响NEAT1_2的表达量分为Ⅰa和Ⅰb 2个亚类。Ⅰa类蛋白质是生成和稳定NEAT1_2的必需蛋白,包括如富脯氨酸和谷氨酰胺的剪接因子(splicing factor proline and glutamine rich,SFPQ)、含非POU域的八聚体结合蛋白(non-POU domain containing octamer binding,NONO)和RNA结合蛋白14(RNA-binding motif protein 14,RBM14)等。Ⅰb类蛋白质对NEAT1_2的表达量不产生影响,包括肉瘤融合/脂肪肉瘤转运蛋白(fused in sarcoma/translocation in liposarcoma,FUS/TLS)、转录激活因子SMARCA4/BRG1(SWI/SNF相关,基质关联,肌动蛋白依赖染色质调控因子,亚家族A,成员4)等[12]。第Ⅱ类蛋白质的缺失可造成核旁斑的大量减少,该类蛋白质的代表是反式激活应答DNA结合蛋白(transactive response DNA binding protein,TARDBP)。第Ⅲ类蛋白质影响核旁斑的数量,根据其是否影响NEAT1_1的表达量可分为Ⅲa和Ⅲb 2个亚类。Ⅲa蛋白质的缺失可造成NEAT1_1的表达量减少,包括剪切及多聚腺苷酸化特异性因子6(cleavage and polyadenylation specific factor 6,CPSF6)、泛素相关蛋白样因子2(ubiquitin associated protein 2 like,UBAP2L)等。Ⅲb类蛋白质不影响NEAT1_1的表达量,包括核旁斑元件1(paraspeckle component 1,PSPC1)、CCCH型锌指蛋白6(zinc finger CCCH-type containing 6,ZC3H6)等。
2 肿瘤细胞核旁斑的结构核旁斑在电子显微镜和超高分辨率结构图解显微镜下呈球形的壳核结构。单个肿瘤细胞中的核旁斑数量大约为5~20个,如HeLa细胞中核旁斑数量约为13~17个。电子显微镜图像显示,核旁斑直径为0.5~1.0 μm。在结构上,NEAT1_2的5'、3' 端以及一些RNA结合蛋白组成球形核旁斑外壳,NEAT1_2的中间区域和其他蛋白质组成球形核旁斑的内核[13]。NEAT1_2的5'、3' 端是亲水的,而中间区域是疏水的,所以形成了核旁斑的核壳结构[14]。核旁斑的球形结构也并非一成不变,只不过与大多数相分离的冷凝体不同,核旁斑并不能沿各个维度生长,它只能拉长变成圆柱状[15]。有研究显示,当NEAT1_2的表达量增多时,单个的核旁斑可串联在一起形成链状结构[8]。另外,在蛋白酶抑制剂的诱导下,核旁斑还可呈细长的香肠状结构[13]。
3 肿瘤细胞核旁斑的装配核旁斑是由结构性RNA和蛋白质通过蛋白质-RNA相互作用、蛋白质-蛋白质相互作用及RNA-RNA相互作用的方式装配而成的核糖核蛋白复合物[12](图 1)。蛋白质NONO和SFPQ与结构性RNA NEAT1_2的C1、C2亚功能区相互作用是核旁斑形成的关键。NONO和SFPQ聚合形成一种异质二聚体,此异质二聚体与NEAT1 _2发生相互作用启动核旁斑的装配,NONO中特有的结构域能与果蝇行为/人类剪切家族蛋白结合[16]。NONO和SFPQ的聚合由SFPQ的卷曲螺旋功能域(coiled-coil domain,CC)介导,该区域最后将完全覆盖于NEAT1_2上。研究显示,NEAT1_2的3个功能域包含多个与NONO和SFPQ的突出结合峰,透射电镜结果也显示SFPQ可通过CC域与NEAT1_2聚合形成高阶复合物[10]。总而言之,NEAT1_2的3个功能域与NONO/SFPQ二聚体的相互作用是后续蛋白聚合的基础,也是核旁斑形成的关键[17]。
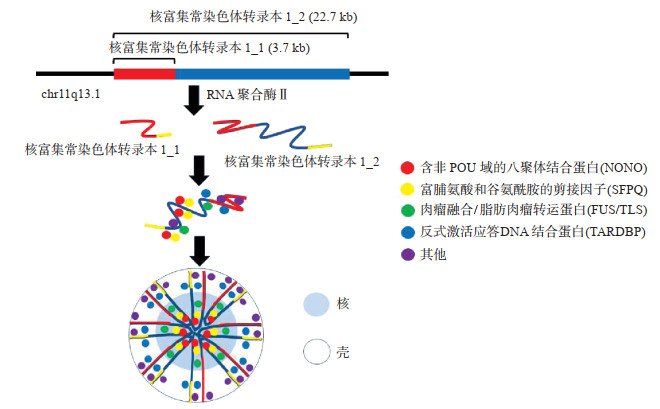
|
图 1 肿瘤细胞核旁斑的组装流程 |
液-液相分离(liquid-liquid phase separation,LLPS)介导的蛋白质-蛋白质相互作用是核旁斑形成的重要方式[17]。LLPS是由具有低复杂性结构域(low complexity domain,LCD)的蛋白质来完成的,LCD没有复杂的三维结构,为多价弱黏附分子间相互作用(如静电、Pi堆积和疏水相互作用)提供了条件[18]。在浓度超过一定的阈值时,蛋白之间发生相互作用,自我组装,经过LLPS之后形成大量无膜体[1]。核旁斑蛋白的LCD又称作朊样结构域,是有效诱导LLPS的重要结构[10, 19]。大多数核旁斑蛋白可以快速进出核旁斑,这也决定了核旁斑作为相分离体具备的一个重要特性——高度动态性。
综上所述,NEAT1_2 C2亚功能区与NONO/SFPQ二聚体的结合通过招募FUS和RBM14来触发LLPS,并最终形成一个大型核旁斑结构[20]。
研究表明,RNA-RNA相互作用也可能在核旁斑的形成中发挥作用[21]。在运用药物使得NONO从核旁斑解离之后,细胞内仍然可以检测到较弱的NEAT1聚合斑点,这说明核旁斑内存在非蛋白质依赖的RNA-RNA相互作用[10]。在生理条件下,足够高浓度的NEAT1可引起RNA的自我组装,这样组装而成的重复性RNA片段可形成独特的核体样结构,这种RNA-RNA相互作用有可能参与了核旁斑的装配[22]。RNA结构性图谱也证明了NEAT1_2分子内大量的RNA-RNA相互作用[23]。所以,非蛋白质依赖的RNA-RNA相互作用可能参与了核旁斑的装配。
4 肿瘤细胞核旁斑的作用核旁斑的很多功能都是通过NEAT1来实现的,很多研究已经证实了NEAT1的促癌作用[24-25]。在非小细胞肺癌中,NEAT1通过吸附miR-98-5p、miR-101-3p、miR-376b-3p分别上调MAPK、性别决定区Y框9(sex-determining region Y box 9,SOX9)、硫酸酯酶1(sulfatase 1,SULF1)的表达,促进癌症进展[26]。在胃癌中,NEAT1通过多种分子信号轴促进肿瘤细胞增殖、浸润和转移,如NEAT1/miR-497-5p/磷酸酰肌醇-3-激酶调节亚基1(phosphoinositide-3-kinase regulatory subunit 1,PIK3R1),NEAT1/miR-335-5p/Rho关联含卷曲螺旋蛋白激酶(Rho-associated coiled-coil containing protein kinase,ROCK),NEAT1/miR-103a/STAM结合蛋白样1(STAM-binding protein like 1,STAMBPL1),以及NEAT1/miR-365a-3p/ATP结合盒式蛋白C(ATP-binding cassette subfamily C,ABCC)[27-28]。NEAT1还靶向miR-296-5p、miR-139-5p和miR-485而分别上调钙调钙素2、TGF-β1、信号转导和转录激活因子3等分子的表达,促进肝癌进程[29]。研究发现,NEAT1分别作用于miR-34a、miR-196a-5p、miR-205-5p、miR-193a、miR-193a-3p、miR-195-5,促进去乙酰化酶1、胶质细胞源性神经营养因子、血管内皮生长因子A、白细胞介素17受体D、类肉瘤病毒癌基因、中心体蛋白55等分子的表达,进而促进直肠癌的进展[30]。在乳腺癌、宫颈癌、前列腺癌及血液系统肿瘤中也存在类似的促癌作用机制[31-34]。总而言之,NEAT1作为核旁斑的必需组分,通过作用于miRNA调控下游分子的表达发挥促癌作用。
也有研究发现NEAT1可能发挥抑癌效应,如在急性早幼粒细胞白血病中,NEAT1通过促进白细胞分化发挥肿瘤抑制作用[35]。在胰腺癌癌前病变的上皮内瘤变组织中,依赖于NEAT1/核旁斑的抑癌因子p53表达上调,对癌的发生和转化发挥抑制作用[36]。在高危神经母细胞瘤中,NEAT1通过驱动分化来抑制肿瘤细胞增殖产生抑癌效应[37]。
值得注意的是,尽管NEAT1是组成核旁斑必不可少的成分,NEAT1和核旁斑也存在确切的关联,但目前的绝大多数研究都独立于核旁斑之外探讨NEAT1作为lncRNA分子在肿瘤中的作用,并没有论证核旁斑对肿瘤的直接影响,还需要进行更加深入的研究来证明核旁斑对肿瘤的直接作用及效应。
5 肿瘤细胞核旁斑的作用机制研究已证实,肿瘤细胞核旁斑可以通过多种机制调控基因表达。如前所述,核旁斑组分NEAT1可作为竞争性内源RNA来吸附miRNA,以此调控癌症相关基因的表达,这是核旁斑发挥促癌作用的重要机制之一。另外,也有研究表明核旁斑的NONO/SFPQ异二聚体可与初级转录物(primary miRNA,pri-miRNA)结合,进而介导Drosha-DGCR8对pri-miRNA进行剪辑处理[38]。
核旁斑另一重要的调控基因表达的机制是对RNA和蛋白质的拘留作用,一方面,核旁斑蛋白NONO可识别并结合mRNA内含子以及3' 非编码区中的Alu元件,进而阻止RNA进入细胞质。另一方面,核旁斑组成蛋白SFPQ是多个凋亡基因的转录因子,当它被拘留在核旁斑时将对一些肿瘤相关基因的表达产生影响,如与B细胞慢性淋巴细胞白血病相关的2个凋亡基因Bcl-2绑定组件3和Bcl-2相关X蛋白的表达将被阻止,肿瘤细胞增殖[39]。
除上述机制之外,NONO/SFPQ异二聚体还可介导典型的非同源末端连接途径,促进DNA双链断裂修复[40]。研究显示,核旁斑各组分可直接结合到染色质上参与转录调控,比如在环磷酸腺苷信号转导通路中,NONO与雷帕霉素靶蛋白结合介导下游靶基因的激活[41]。另有研究发现SFPQ和NEAT1_2结合在活化基因的启动子附近,NONO和SFPQ还能直接结合到RNA聚合酶Ⅱ的羧基末端结构域上并参与基因表达调控[42]。
6 肿瘤细胞核旁斑的临床意义研究表明,NEAT1在肝癌[29]、肺癌[26]、胃癌[27]、直肠癌[30]等多种恶性肿瘤中异常高表达,且介导多种作用机制调节下游靶基因或信号通路,促进肿瘤细胞增殖、迁移、侵袭、耐药性和放射抵抗等恶性生物学行为。在非小细胞肺癌中,NEAT1的高表达与患者年龄、血管浸润、淋巴转移和肿瘤TNM分期呈正相关[43]。研究还发现,NEAT1的高表达与喉鳞状细胞癌的肿瘤大小、临床分期和淋巴结转移显著相关,且NEAT1高表达的喉鳞状细胞癌患者总体生存率偏低[44]。另外,在结直肠癌中,NEAT1高表达的患者无病生存期与总体生存期降低,且单因素COX回归分析表明NEAT1高表达是预后不良的独立标志物[45]。除此之外,NEAT1的高表达与肝癌TNM分期、转移、肿瘤结节数和肿瘤细胞浸润程度也显著相关[46]。在乳腺癌、卵巢癌、前列腺癌等肿瘤中,NEAT1高表达同样提示不良预后[47-49]。
核旁斑的必需组分NEAT1在诸多肿瘤中均可充当诊断及预后标志物,还是潜在的癌症治疗靶点。如Wang等[50]发现,敲除NEAT1可通过抑制细胞周期蛋白依赖激酶6(cyclin dependent kinase 6,CDK6)的表达抑制喉鳞状细胞癌的增殖,推测NEAT1是喉鳞状细胞癌的潜在治疗靶点。还有研究发现NEAT1的高表达和多种肿瘤的放化疗抵抗相关,而敲除NEAT1可提高肿瘤的放化疗敏感性[2]。综上所述,NEAT1及核旁斑可能具备早期诊断、靶向治疗和预测预后的临床应用价值。
7 小结与展望肿瘤细胞核旁斑是由结构性RNA NEAT1和多个RNA结合蛋白在空间上有序排列组装而成的大型核糖核蛋白复合物。其主要成分NEAT1_2以及SFPQ、NONO和RBM14等RNA结合蛋白通过蛋白质-RNA相互作用、蛋白质-蛋白质相互作用及RNA-RNA相互作用的方式装配成核旁斑。一般情况下,核旁斑呈球形壳核状结构,亲水的NEAT1_2的5'、3' 端以及一些RNA结合蛋白组成球形核旁斑外壳,疏水的NEAT1_2的中间区域和其他蛋白质组成球形核旁斑的内核。核旁斑可通过RNA或蛋白的拘留以及与miRNA相互作用等方式调控基因的表达,从而促进肿瘤细胞增殖、迁移、侵袭、耐药性和放射抵抗等恶性生物学行为,因此,NEAT1及核旁斑可能具备早期诊断、靶向治疗和预测预后的临床应用价值。不过,目前的很多研究都只是独立于核旁斑之外探究NEAT1在肿瘤中的作用。尽管NEAT1是核旁斑必不可少的组成成分,NEAT1和核旁斑存在确切的关联,但核旁斑还有其他成分,其功能要比NEAT1复杂的多。核旁斑直接影响肿瘤进展的机制尚待进一步探究。
| [1] |
HIROSE T, YAMAZAKI T, NAKAGAWA S. Molecular anatomy of the architectural NEAT1 noncoding RNA: the domains, interactors, and biogenesis pathway required to build phase-separated nuclear paraspeckles[J]. Wiley Interdiscip Rev RNA, 2019, 10(6): e1545. DOI:10.1002/wrna.1545 |
| [2] |
LI K, YAO T, ZHANG Y, et al. NEAT1 as a competing endogenous RNA in tumorigenesis of various cancers: role, mechanism and therapeutic potential[J]. Int J Biol Sci, 2021, 17(13): 3428-3440. DOI:10.7150/ijbs.62728 |
| [3] |
WANG Z, LI K, HUANG W. Long non-coding RNA NEAT1-centric gene regulation[J]. Cell Mol Life Sci, 2020, 77(19): 3769-3779. DOI:10.1007/s00018-020-03503-0 |
| [4] |
ZHANG M, GUO J, LIU L, et al. The role of long noncoding RNA, nuclear enriched abundant transcript 1(NEAT1) in cancer and other pathologies[J]. Biochem Genet, 2022, 60(3): 843-867. DOI:10.1007/s10528-021-10138-8 |
| [5] |
PISANI G, BARON B. NEAT1 and paraspeckles in cancer development and chemoresistance[J]. NcRNA, 2020, 6(4): 43. DOI:10.3390/ncrna6040043 |
| [6] |
LI R, HARVEY A R, HODGETTS S I, et al. Functional dissection of NEAT1 using genome editing reveals substantial localization of the NEAT1_1 isoform outside paraspeckles[J]. RNA, 2017, 23(6): 872-881. DOI:10.1261/rna.059477.116 |
| [7] |
NAGANUMA T, NAKAGAWA S, TANIGAWA A, et al. Alternative 3'-end processing of long noncoding RNA initiates construction of nuclear paraspeckles[J]. EMBO J, 2012, 31(20): 4020-4034. DOI:10.1038/emboj.2012.251 |
| [8] |
WANG Y, HU S B, WANG M R, et al. Genome-wide screening of NEAT1 regulators reveals cross-regulation between paraspeckles and mitochondria[J]. Nat Cell Biol, 2018, 20(10): 1145-1158. DOI:10.1038/s41556-018-0204-2 |
| [9] |
NAKAGAWA S, NAGANUMA T, SHIOI G, et al. Paraspeckles are subpopulation-specific nuclear bodies that are not essential in mice[J]. J Cell Biol, 2011, 193(1): 31-39. DOI:10.1083/jcb.201011110 |
| [10] |
YAMAZAKI T, SOUQUERE S, CHUJO T, et al. Functional domains of NEAT1 architectural lncRNA induce paraspeckle assembly through phase separation[J]. Mol Cell, 2018, 70(6): 1038-1053. DOI:10.1016/j.molcel.2018.05.019 |
| [11] |
FONG K W, LI Y, WANG W, et al. Whole-genome screening identifies proteins localized to distinct nuclear bodies[J]. J Cell Biol, 2013, 203(1): 149-164. DOI:10.1083/jcb.201303145 |
| [12] |
刘晨. 核体paraspeckle结构及形成研究进展[J]. 安徽农业科学, 2020, 48(19): 4-8. DOI:10.3969/j.issn.0517-6611.2020.19.002 |
| [13] |
WEST J A, MITO M, KUROSAKA S, et al. Structural, super-resolution microscopy analysis of paraspeckle nuclear body organization[J]. J Cell Biol, 2016, 214(7): 817-830. DOI:10.1083/jcb.201601071 |
| [14] |
YAMAZAKI T, YAMAMOTO T, YOSHINO H, et al. Paraspeckles are constructed as block copolymer micelles[J]. EMBO J, 2021, 40(12): e107270. DOI:10.15252/embj.2020107270 |
| [15] |
HIROSE T, VIRNICCHI G, TANIGAWA A, et al. NEAT1 long noncoding RNA regulates transcription via protein sequestration within subnuclear bodies[J]. Mol Biol Cell, 2014, 25(1): 169-183. DOI:10.1091/mbc.E13-09-0558 |
| [16] |
KNOTT G J, BOND C S, FOX A H. The DBHS proteins SFPQ, NONO and PSPC1: a multipurpose molecular scaffold[J]. Nucleic Acids Res, 2016, 44(9): 3989-4004. DOI:10.1093/nar/gkw271 |
| [17] |
SAWYER I A, STURGILL D, DUNDR M. Membraneless nuclear organelles and the search for phases within phases[J]. Wiley Interdiscip Rev RNA, 2019, 10(2): e1514. DOI:10.1002/wrna.1514 |
| [18] |
ALBERTI S, GLADFELTER A, MITTA G T. Considerations and challenges in studying liquid-liquid phase separation and biomolecular condensates[J]. Cell, 2019, 176(3): 419-434. DOI:10.1016/j.cell.2018.12.035 |
| [19] |
FOX A H, NAKAGAWA S, HIROSE T, et al. Paraspeckles: where long noncoding RNA meets phase separation[J]. Trends Biochem Sci, 2018, 43(2): 124-135. DOI:10.1016/j.tibs.2017.12.001 |
| [20] |
MAHARANA S, WANG J, PAPADOPOULOS D K, et al. RNA buffers the phase separation behavior of prion-like RNA binding proteins[J]. Science, 2018, 360(6391): 918-921. DOI:10.1126/science.aar7366 |
| [21] |
VAN TREECK B, PROTTER D S W, MATHENY T, et al. RNA self-assembly contributes to stress granule formation and defining the stress granule transcriptome[J]. Proc Natl Acad Sci U S A, 2018, 115(11): 2734-2739. DOI:10.1073/pnas.1800038115 |
| [22] |
VAN TREECK B, PARKER R. Emerging roles for intermolecular RNA-RNA interactions in RNP assemblies[J]. Cell, 2018, 174(4): 791-802. DOI:10.1016/j.cell.2018.07.023 |
| [23] |
LIN Y, SCHMIDT B F, BRUCHEZ M P, et al. Structural analyses of NEAT1 lncRNAs suggest long-range RNA interactions that may contribute to paraspeckle architecture[J]. Nucleic Acids Res, 2018, 46(7): 3742-3752. DOI:10.1093/nar/gky046 |
| [24] |
ZHANG Y, LUN L, LI H, et al. The value of lncRNA NEAT1 as a prognostic factor for survival of cancer outcome: a meta-analysis[J]. Sci Rep, 2017, 7(1): 13080. DOI:10.1038/s41598-017-10001-0 |
| [25] |
CHEN T, WANG H, YANG P, et al. Prognostic role of long noncoding RNA NEAT1 in various carcinomas: a meta-analysis[J]. OncoTargets Ther, 2017, 10: 993-1000. DOI:10.2147/ott.s128588 |
| [26] |
CHEN L M, NIU Y D, XIAO M, et al. LncRNA NEAT1 regulated cell proliferation, invasion, migration and apoptosis by targeting has-miR-376b-3p/SULF1 axis in non-small cell lung cancer[J]. Eur Rev Med Pharmacol Sci, 2020, 24(9): 4810-4821. DOI:10.26355/eurrev_202005_21170 |
| [27] |
GAO M, LIU L, ZHANG D, et al. Long non-coding RNA NEAT1 serves as sponge for miR-365a-3p to promote gastric cancer progression via regulating ABCC4[J]. OncoTargets Ther, 2020, 13: 3977-3985. DOI:10.2147/ott.s245557 |
| [28] |
YU D J, GUO C X, QIAN J, et al. The long non-coding RNA NEAT1 promotes gastric cancer cell proliferation and invasion by regulating miR-103a/ STAMBPL1 axis[J]. Technol Cancer Res Treat, 2020, 19: 1533033820964081. DOI:10.1177/1533033820964081 |
| [29] |
LI Y, DING X, XIU S, et al. LncRNA NEAT1 promotes proliferation, migration and invasion via regulating miR- 296-5p/CNN2 axis in hepatocellular carcinoma cells[J]. OncoTargets Ther, 2019, 12: 9887-9897. DOI:10.2147/ott.s228917 |
| [30] |
WANG S, DU H, SUN P. Long noncoding RNA NEAT1 contributes to the tumorigenesis of colorectal cancer through regulating SLC38A1 expression by sponging miR-138[J]. Cancer Biotherapy Radiopharm, 2021, 36(9): 793-802. DOI:10.1089/cbr.2020.3608 |
| [31] |
LIU X, YAO W, XIONG H, et al. LncRNA NEAT1 accelerates breast cancer progression through regulating miR-410-3p/CCND1 axis[J]. Cancer Biomark, 2020, 29(2): 277-290. DOI:10.3233/cbm-190721 |
| [32] |
XU H, SUN X, HUANG Y, et al. Long non coding RNA NEAT1 modifies cell proliferation, colony formation, apoptosis, migration and invasion via the miR 4500/ BZW1 axis in ovarian cancer[J]. Mol Med Rep, 2020, 22(4): 3347-3357. DOI:10.3892/mmr.2020.11408 |
| [33] |
GUO Z, HE C, YANG F, et al. Long non-coding RNANEAT1, a sponge for miR-98-5p, promotes expression of oncogene HMGA2 in prostate cancer[J]. Biosci Rep, 2019, 39(9): BSR20190635. DOI:10.1042/BSR20190635 |
| [34] |
ZHAO C, WANG S, ZHAO Y, et al. Long noncoding RNA NEAT1 modulates cell proliferation and apoptosis by regulating miR-23a-3p/SMC1A in acute myeloid leukemia[J]. J Cell Physiol, 2019, 234(5): 6161-6172. DOI:10.1002/jcp.27393 |
| [35] |
YU X, LI Z, ZHENG H, et al. NEAT1: a novel cancerrelated long non-coding RNA[J]. Cell Prolif, 2017, 50(2): e12329. DOI:10.1111/cpr.12329 |
| [36] |
MELLO S S, SINOW C, RAJ N, et al. Neat1 is a p53-inducible lincRNA essential for transformation suppression[J]. Genes Dev, 2017, 31(11): 1095-1108. DOI:10.1101/gad.284661.116 |
| [37] |
NAVEED A, COOPER J A, LI R, et al. NEAT1 polyAmodulating antisense oligonucleotides reveal opposing functions for both long non-coding RNA isoforms in neuroblastoma[J]. Cell Mol Life Sci, 2021, 78(5): 2213-2230. DOI:10.1007/s00018-020-03632-6 |
| [38] |
JIANG L, SHAO C, WU Q J, et al. NEAT1 scaffolds RNA-binding proteins and the microprocessor to globally enhance pri-miRNA processing[J]. Nat Struct Mol Biol, 2017, 24(10): 816-824. DOI:10.1038/nsmb.3455 |
| [39] |
ZENG C, LIU S, LU S, et al. The c-Myc-regulated lncRNA NEAT1 and paraspeckles modulate imatinibinduced apoptosis in CML cells[J]. Mol Cancer, 2018, 17(1): 130. DOI:10.1186/s12943-018-0884-z |
| [40] |
JAAFAR L, LI Z, LI S, et al. SFPQ·NONO and XLF function separately and together to promote DNA double-strand break repair via canonical nonhomologous end joining[J]. Nucleic Acids Res, 2017, 45(4): 1848-1859. DOI:10.1093/nar/gkw1209 |
| [41] |
LU J Y, SEWER M B. p54nrb/NONO regulates cyclic AMP-dependent glucocorticoid production by modulating phosphodiesterase mRNA splicing and degradation[J]. Mol Cell Biol, 2015, 35(7): 1223-1237. DOI:10.1128/MCB.00993-14 |
| [42] |
WEST J A, DAVIS C P, SUNWOO H, et al. The long noncoding RNAs NEAT1 and MALAT1 bind active chromatin sites[J]. Mol Cell, 2014, 55(5): 791-802. DOI:10.1016/j.molcel.2014.07.012 |
| [43] |
PAN L J, ZHONG T F, TANG R X, et al. Upregulation and clinicopathological significance of long non-coding NEAT1 RNA in NSCLC tissues[J]. Asian Pac J Cancer Prev, 2015, 16(7): 2851-2855. DOI:10.7314/apjcp.2015.16.7.2851 |
| [44] |
YUEQUAN J, SHIFENG C, BING Z. Prognostic factors and family history for survival of esophageal squamous cell carcinoma patients after surgery[J]. Ann Thorac Surg, 2010, 90(3): 908-913. DOI:10.1016/j.athoracsur.2010.05.060 |
| [45] |
LI Y, LI Y, CHEN W, et al. NEAT expression is associated with tumor recurrence and unfavorable prognosis in colorectal cancer[J]. Oncotarget, 2015, 6(29): 27641-27650. DOI:10.18632/oncotarget.4737 |
| [46] |
GUO S, CHEN W, LUO Y, et al. Clinical implication of long non-coding RNA NEAT1 expression in hepatocellular carcinoma patients[J]. Int J Clin Exp Pathol, 2015, 8(5): 5395-5402. |
| [47] |
CHEN Z, FENG R, KAHLERT U D, et al. Construction of ceRNA networks associated with CD8 T cells in breast cancer[J]. Front Oncol, 2022, 12: 883197. DOI:10.3389/fonc.2022.883197 |
| [48] |
JIA X, WEI L, ZHANG Z. NEAT1 Overexpression indicates a poor prognosis and induces chemotherapy resistance via the miR-491-5p/SOX3 signaling pathway in ovarian cancer[J]. Front Genet, 2021, 12: 616220. DOI:10.3389/fgene.2021.616220 |
| [49] |
LI W, XU W, SUN K, WANG F, WONG T, KONG A. Identification of novel biomarkers in prostate cancer diagnosis and prognosis[J]. Journal of biochemical and molecular toxicology, 2022, e23137. |
| [50] |
WANG P, WU T, ZHOU H, et al. Long noncoding RNA NEAT1 promotes laryngeal squamous cell cancer through regulating miR-107/CDK6 pathway[J]. J Exp Clin Cancer Res, 2016, 35: 22. DOI:10.1186/s13046-016-0297-z |
 2023, Vol. 44
2023, Vol. 44


