2. 中国人民解放军联勤保障部队第九〇〇医院骨科, 福州 350025;
3. 海军军医大学(第二军医大学)第三附属医院骨科, 上海 201805;
4. 同济大学附属上海市肺科医院检验科, 上海 200433
2. Department of Orthopaedics, The 900th Hospital of Joint Logistic Support Force of PLA, Fuzhou 350025, Fujian, China;
3. Department of Orthopaedics, The Third Affiliated Hosptial of Naval Medical University (Second Military Medical University), Shanghai 201805, China;
4. Department of Clinical Laboratory, Shanghai Pulmonary Hospital, Tongji University, Shanghai 200433, China
骨肉瘤是儿童和青少年中最常见的原发性恶性骨肿瘤[1],容易早期发生肺转移,致死、致残率高。虽然化学治疗的进步使初诊时未发生远处转移的骨肉瘤患者的5年生存率提高到60%~75%[2],但存在复发、转移的骨肉瘤患者生存率仍然很低[1]。分子靶向治疗和基因免疫治疗可能是恶性肿瘤治疗未来发展的方向。
长链非编码RNA(long non-coding RNA,lncRNA)是一类长度>200个核苷酸的RNA分子[3],不编码蛋白质,但在多种机制上调控基因的表达,并与多种肿瘤的发生、发展密切相关。肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,MALAT-1)属于lncRNA家族,最早在非小细胞肺癌中被发现,已被证实在多种肿瘤的增殖、侵袭、转移中发挥作用[4]。研究显示,MALAT-1与骨肉瘤患者的预后及骨肉瘤细胞的侵袭、转移密切相关[5-10]。目前有关MALAT-1表达改变的骨肉瘤细胞系在动物模型中的研究仅停留在对生长出的骨肉瘤的体积、质量等宏观层面的观察,没有进行微观层面的研究,MALAT-1表达改变对骨肉瘤内部产生的影响仍然未知。本研究通过建立裸鼠移植瘤模型,先观察MALAT-1稳定低表达的骨肉瘤细胞系植入裸鼠皮下后的生长情况,再通过H-E染色、免疫组织化学等方法在病理切片上探究抑制MALAT-1表达引起的骨肉瘤增殖性改变。
1 材料和方法 1.1 细胞培养与MALAT-1稳定低表达细胞系的构建人骨肉瘤U2OS细胞系购自上海赛新生物科技有限公司,采用含10% FBS的完全培养基(美国Invitrogen公司)在37 ℃、5% CO2培养箱中培养。细胞分为实验组、对照组、无处理组。慢病毒载体由上海赛新生物科技有限公司构建,实验组细胞使用携带MALAT-1 shRNA质粒的慢病毒感染,对照组细胞使用空载慢病毒感染,无处理组细胞不进行感染。感染24 h后,使用含嘌呤霉素的培养基筛选抗性细胞,传代并扩大培养,在荧光显微镜(日本Olympus公司)下观察、鉴定。
1.2 qPCR检测使用TRIzol试剂(北京艾德莱生物科技有限公司)提取细胞总RNA,然后用反转录试剂盒(美国GeneCopoeia公司)将RNA反转录为cDNA,进行qPCR扩增。引物序列:β-肌动蛋白正向引物为5'-CTGGAACGGT-GAAGGTGACA-3',反向引物为5'-AAGGGACT-TCCTGTAACAATGCA-3';MALAT-1正向引物为5'-GCAGCAGTTCGTGGTGAAGATAG-3',反向引物为5'-CGCCTCCTCCGTGTGGTTG-3'。以2-ΔΔCt法计算目的基因的相对表达量。
1.3 裸鼠皮下移植瘤模型的构建12只4~6周龄的雄性裸鼠由海军军医大学(第二军医大学)实验动物中心[实验动物使用许可证号:SYXK(沪)2015-0017]提供,随机分为实验组和对照组,每组6只。实验组裸鼠皮下植入MALAT-1稳定低表达的骨肉瘤U2OS细胞,对照组裸鼠皮下植入空载慢病毒处理的骨肉瘤U2OS细胞,注射细胞悬液体积均为0.1 mL。
1.4 肿瘤体积与质量的测定植瘤后第3、6、9、12、15、18、21天,分别用游标卡尺测量裸鼠皮下肿瘤的长度和宽度,计算肿瘤体积:肿瘤体积=(长度×宽度2)/2[11]。植瘤后第21天处死裸鼠,将裸鼠皮下肿瘤完整剥出,称取肿瘤质量。
1.5 H-E染色及评价肿瘤组织称重后用蜡块包埋,用病理切片机(德国Leica公司)切成厚度为4 μm的切片,经烤片、脱蜡、浸洗后,用Harris苏木精染液染色7 min,浸洗、分化、再浸洗,再用1%水溶性伊红染液染色2 min,浸洗、无水乙醇脱水、二甲苯透明、风干,中性树胶封片。在光学显微镜下观察肿瘤病理学形态。在放大100倍视野下,在每个肿瘤标本的切片上随机取5个视野,按以下标准进行评分:1分,视野中肿瘤细胞的范围<30%;2分,视野中肿瘤细胞的范围占30%~60%;3分,视野中肿瘤细胞的范围>60%。以5个视野得分的平均值作为最终得分。
1.6 免疫组织化学染色及评价对每个骨肉瘤标本分别进行增殖细胞核抗原(proliferating cell nuclear antigen,PCNA)和血管内皮生长因子(vascular endothelial growth factor,VEGF)免疫组织化学染色。将石蜡切片脱蜡及浸洗后,用EDTA抗原修复缓冲液(武汉谷歌生物科技有限公司)修复抗原。室温下遮光孵育25 min。用3%牛血清白蛋白(bovine serum albumin,BSA;北京索莱宝科技有限公司)均匀覆盖组织,室温下封闭30 min。使用丹麦DAKO公司免疫组织化学染色试剂盒,按照说明书在切片上先后滴加一抗和二抗并孵育,DAB显色液显色。用Harris苏木精染液复染,经分化、氨水返蓝、冲洗、脱水透明、晾干,中性树胶封片。在光学显微镜下观察PCNA和VEGF的表达情况。在每个肿瘤标本的切片上随机选择3个放大200倍的视野进行拍照,拍照时尽量让组织充满整个视野,保证每张照片的背景光一致。应用Image-Pro Plus 6.0软件对每张照片进行分析,选取相同的棕黄色作为判断所有照片阳性的统一标准,获得阳性染色的积分光密度值(integral optical density,IOD)。在放大400倍的视野下,在每个肿瘤标本的切片上随机选择不重叠的10个视野,共计数1 000个细胞。分别记录每个肿瘤标本中未着色(阴性)、淡着色(弱阳性)、中等以上程度着色(阳性)细胞的数量,计算各类细胞所占比例。
1.7 统计学处理应用SPSS 19.0软件进行统计学分析。符合正态分布的计量资料以x±s表示,两组间比较采用独立样本t检验或校正t检验,多组间比较采用单因素方差分析(多重比较采用最小显著性差异法);不符合正态分布的计量资料以中位数(范围)表示,组间比较采用Mann-Whitney U检验;等级资料以细胞个数和百分数表示,组间比较采用秩和检验。检验水准(α)为0.05。
2 结果 2.1 MALAT-1稳定低表达的骨肉瘤细胞系成功构建qPCR检测结果显示,对照组、无处理组、实验组骨肉瘤U2OS细胞中MALAT-1表达水平分别为1.000±0.000、0.916±0.150、0.207±0.034,3组间差异有统计学意义(P<0.01),实验组细胞中MALAT-1表达相比于对照组受到抑制(P<0.01),而对照组和未经处理组之间差异无统计学意义(P=0.290)。
2.2 MALAT-1低表达减缓裸鼠皮下骨肉瘤的生长肿瘤体积测量结果显示,实验组和对照组裸鼠皮下的骨肉瘤都随着时间有不同程度的生长。植瘤后第9天到第21天,两组裸鼠骨肉瘤的体积差异均有统计学意义(P均<0.01);与对照组相比,实验组裸鼠皮下的骨肉瘤生长更慢、体积更小(表 1)。在植瘤后第21天,实验组裸鼠皮下骨肉瘤的质量低于对照组,差异有统计学意义[(1.052±0.087)g vs(1.298±0.144)g,P=0.007]。
|
|
表 1 皮下植瘤后两组裸鼠骨肉瘤体积随时间的变化 Tab 1 Changes of osteosarcoma volume after subcutaneous tumor implantation in nude mice of 2 groups |
2.3 MALAT-1低表达抑制骨肉瘤细胞增殖并促进坏死
H-E染色结果显示,两组裸鼠皮下骨肉瘤组织均可见梭形肿瘤细胞及骨样组织,细胞内核分裂象多见,骨样组织呈花边样、条索样及片状分布在肿瘤细胞周围。在放大100倍视野下,相比于对照组(图 1A),实验组(图 1B、1C)裸鼠骨肉瘤中的基质成分明显增多,视野中肿瘤细胞范围减少;实验组染色得分低于对照组,差异有统计学意义(1.867±0.301 vs 2.600±0.358,P=0.003)。在放大200倍视野下,与对照组(图 1D)相比,实验组(图 1E、1F)的细胞核数量减少,核分裂象相对少见,肿瘤细胞密集程度降低,骨样组织成分增多,这在坏死区域周围更为明显。结果表明,抑制骨肉瘤细胞MALAT-1的表达可以抑制骨肉瘤细胞的增殖、促进骨肉瘤细胞的坏死并增加成骨。
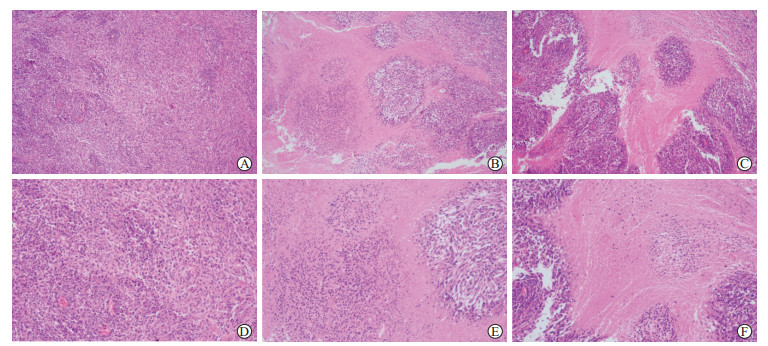
|
图 1 抑制MALAT-1对裸鼠皮下移植骨肉瘤病理形态学的影响 Fig 1 Effect of MALAT-1 inhibition on pathomorphology of subcutaneously implanted osteosarcoma in nude mice A, D: Control group; B, C, E, F: Experimental group. In osteosarcoma of the experimental group, the matrix components increased and the tumor cells decreased, and the density of tumor cells decreased and the composition of osteoid tissue increased. Hematoxylin-eosin staining, 100× (A-C), 200× (D-F). Osteosarcoma U2OS cells with stable low expression of MALAT-1 were implanted subcutaneously in the experimental group, while osteosarcoma U2OS cells infected by empty lentivirus were implanted subcutaneously in the control group. MALAT-1: Metastasis-associated lung adenocarcinoma transcript 1. |
2.4 MALAT-1低表达使骨肉瘤中PCNA的表达降低
PCNA免疫组织化学染色结果显示,骨肉瘤细胞核被不同程度地染色。在放大200倍视野下,与对照组(图 2A)相比,实验组(图 2B、2C)裸鼠皮下生长的骨肉瘤染色程度较弱,IOD差异有统计学意义[8 531.03(491.42~12 889.70)vs 27 548.23(5 135.39~72 720.68),P=0.025]。在放大400倍视野下进行阳性着色细胞计数,与对照组(图 2D)相比,实验组(图 2E、2F)阳性细胞占比较低,差异有统计学意义(P<0.001,表 2)。结果表明,抑制骨肉瘤细胞MALAT-1的表达可以降低肿瘤细胞中PCNA的表达、降低肿瘤的侵袭性。
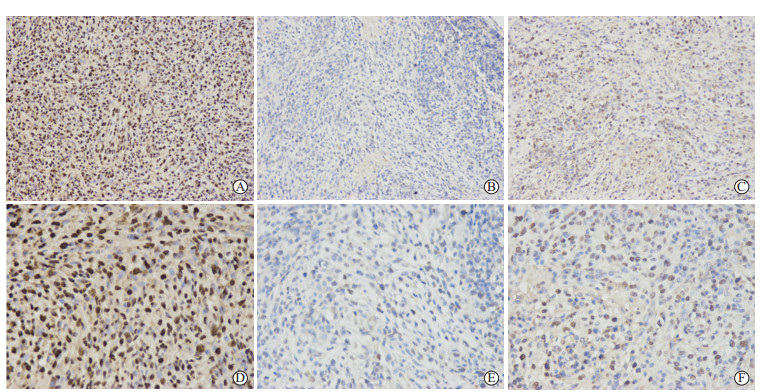
|
图 2 抑制MALAT-1对裸鼠皮下移植骨肉瘤中PCNA表达的影响 Fig 2 Effect of MALAT-1 inhibition on PCNA expression in subcutaneously implanted osteosarcoma in nude mice A, D: Control group; B, C, E, F: Experimental group. In osteosarcoma of the experimental group, the degree of PCNA staining decreased, and there were few PCNA positive cells. Immunohistochemical straining, 200× (A-C), 400× (D-F). Osteosarcoma U2OS cells with stable low expression of MALAT-1 were implanted subcutaneously in the experimental group, while osteosarcoma U2OS cells infected by empty lentivirus were implanted subcutaneously in the control group. MALAT-1: Metastasis-associated lung adenocarcinoma transcript 1; PCNA: Proliferating cell nuclear antigen. |
|
|
表 2 两组裸鼠皮下移植骨肉瘤免疫组织化学染色阳性着色细胞计数结果 Tab 2 Immunohistochemical staining positive results of subcutaneously implanted osteosarcoma in nude mice of 2 groups |
2.5 MALAT-1低表达不改变骨肉瘤中VEGF的表达
VEGF免疫组织化学染色结果显示,骨肉瘤细胞质被染色。在放大200倍视野下,对照组(图 3A)和实验组(图 3B)裸鼠皮下生长的骨肉瘤染色程度均较深,IOD差异无统计学意义(15 850.632±9 341.063 vs 18 179.490±18 299.433,P=0.787)。在放大400倍视野下,阳性着色细胞计数显示,对照组(图 3C)与实验组(图 3D)阳性细胞占比差异无统计学意义(P=0.229,表 2)。
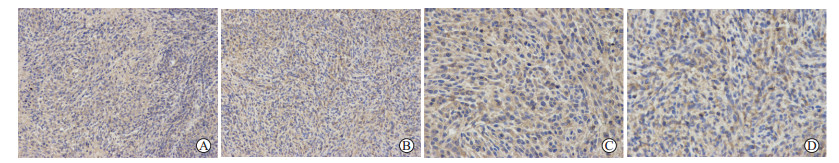
|
图 3 抑制MALAT-1对裸鼠皮下移植骨肉瘤中VEGF表达的影响 Fig 3 Effect of MALAT-1 inhibition on VEGF expression in subcutaneously implanted osteosarcoma in nude mice A, C: Control group; B, D: Experimental group. There was no difference in VEGF staining degree or positive cell proportion between control group and experimental group. Immunohistochemical straining, 200× (A, B), 400× (C, D). Osteosarcoma U2OS cells with stable low expression of MALAT-1 were implanted subcutaneously in the experimental group, while osteosarcoma U2OS cells infected by empty lentivirus were implanted subcutaneously in the control group. MALAT-1: Metastasis-associated lung adenocarcinoma transcript 1; VEGF: Vascular endothelial growth factor. |
3 讨论
MALAT-1最早是作为与肺腺癌相关的预后标志物而被广泛研究,此后发现它在多种常见实体肿瘤的发生、发展中有重要作用。研究发现,骨肉瘤中MALAT-1高表达与骨肉瘤患者的预后及肿瘤细胞的侵袭、转移密切相关[5-10]。相关的机制包括上调miRNA-9表达[10]、通过miRNA-34a/c-5p和miRNA-449a/b靶向肝细胞生长因子受体和性别决定区相关高迁移率族盒蛋白4[9]、调节miRNA34a/细胞周期蛋白1轴[8]、调节RhoA/Rho激酶通路[7]、激活PI3K-Akt通路[5]等。但是,既往的动物实验仅测量了肿瘤的体积和质量,而对实体骨肉瘤的病理学改变没有做进一步探究。
本研究根据既往报道以在骨肉瘤中MALAT-1表达较高的U2OS细胞系[7]进行实验,用慢病毒感染培养出MALAT-1稳定低表达的骨肉瘤U2OS细胞系,以空载慢病毒处理的U2OS细胞系作为对照,探索MALAT-1表达对骨肉瘤增殖的影响。本研究结果显示,与MALAT-1表达基本正常的对照组细胞相比,MALAT-1稳定低表达的实验组细胞在植入裸鼠皮下后生长更慢,植瘤第21天的骨肉瘤体积小、质量低;且病理学实验证实,MALAT-1低表达的骨肉瘤细胞生长出的实体骨肉瘤内基质成分增多,骨样组织成分增多,肿瘤细胞密集程度降低,PCNA表达下降。结果提示,抑制MALAT-1的表达可以抑制骨肉瘤的增殖、促进肿瘤的坏死、降低肿瘤的侵袭性。
PCNA主要表达于细胞核中,是反映细胞增殖状态的良好指标[12]。VEGF主要表达于细胞质中,是一种促血管生成因子,也是影响恶性肿瘤新生血管形成的重要因素之一[13]。在本实验中,MALAT-1低表达的骨肉瘤细胞系生长出的肿瘤PCNA的表达降低,验证了抑制MALAT-1能够降低骨肉瘤的增殖能力。此前有研究表明MALAT-1参与调控血管内皮细胞的功能,并且促进血管生长[14],在本研究中,实验组和对照组的骨肉瘤VEGF表达差异没有统计学意义,原因可能包括:(1)MALAT-1对血管内皮细胞功能的影响不能直接反映在VEGF的表达程度上;(2)本研究中两组的切片中VEGF阳性的细胞比例和染色强度都较高,可能需要更多的样本量或更精确的方法来分析两组间VEGF表达的差别。
总之,本研究结果显示,MALAT-1表达在骨肉瘤的增殖中发挥重要作用。MALAT-1低表达可以延缓骨肉瘤的生长,降低骨肉瘤的侵袭性,增加肿瘤内的骨样组织成分,促进实体骨肉瘤中肿瘤细胞的坏死。
| [1] |
HARRISON D J, GELLER D S, GILL J D, LEWIS V O, GORLICK R. Current and future therapeutic approaches for osteosarcoma[J]. Expert Rev Anticancer Ther, 2017, 18: 39-50. |
| [2] |
GRINBERG S Z, POSTA A, WEBER K L, WILSON R J. Limb salvage and reconstruction options in osteosarcoma[J]. Adv Exp Med Biol, 2020, 1257: 13-29. |
| [3] |
ZHANG X, HAMBLIN M H, YIN K J. The long noncoding RNA Malat1:its physiological and pathophysiological functions[J]. RNA Biol, 2017, 14: 1705-1714. DOI:10.1080/15476286.2017.1358347 |
| [4] |
LI Z X, ZHU Q N, ZHANG H B, HU Y, WANG G, ZHU Y S. MALAT1:a potential biomarker in cancer[J]. Cancer Manag Res, 2018, 10: 6757-6768. DOI:10.2147/CMAR.S169406 |
| [5] |
CHEN Y, HUANG W, SUN W, ZHENG B, WANG C, LUO Z, et al. LncRNA MALAT1 promotes cancer metastasis in osteosarcoma via activation of the PI3K-Akt signaling pathway[J]. Cell Physiol Biochem, 2018, 51: 1313-1326. DOI:10.1159/000495550 |
| [6] |
LIU M, YANG P, MAO G, DENG J, PENG G, NING X, et al. Long non-coding RNA MALAT1 as a valuable biomarker for prognosis in osteosarcoma: a systematic review and meta-analysis[J]. Int J Surg, 2019, 72: 206-213. DOI:10.1016/j.ijsu.2019.11.004 |
| [7] |
CAI X, LIU Y, YANG W, XIA Y, YANG C, YANG S, et al. Long noncoding RNA MALAT1 as a potential therapeutic target in osteosarcoma[J]. J Orthop Res, 2016, 34: 932-941. DOI:10.1002/jor.23105 |
| [8] |
DUAN G, ZHANG C, XU C, XU C, ZHANG L, ZHANG Y. Knockdown of MALAT1 inhibits osteosarcoma progression via regulating the miR34a/cyclin D1 axis[J]. Int J Oncol, 2019, 54: 17-28. |
| [9] |
SUN Z, ZHANG T, CHEN B. Long non-coding RNA metastasis-associated lung adenocarcinoma transcript 1(MALAT1) promotes proliferation and metastasis of osteosarcoma cells by targeting c-Met and SOX4 via miR-34a/c-5p and miR-449a/b[J]. Med Sci Monit, 2019, 25: 1410-1422. DOI:10.12659/MSM.912703 |
| [10] |
FANG D, YANG H, LIN J, TENG Y, JIANG Y, CHEN J, et al. 17β-estradiol regulates cell proliferation, colony formation, migration, invasion and promotes apoptosis by upregulating miR-9 and thus degrades MALAT-1 in osteosarcoma cell MG-63 in an estrogen receptor-independent manner[J]. Biochem Biophys Res Commun, 2015, 457: 500-506. DOI:10.1016/j.bbrc.2014.12.114 |
| [11] |
TOMAYKO M M, REYNOLDS C P. Determination of subcutaneous tumor size in athymic (nude) mice[J]. Cancer Chemother Pharmacol, 1989, 24: 148-154. DOI:10.1007/BF00300234 |
| [12] |
李永昊, 肖玉周, 汪万英. 骨肉瘤中hMLH1、hMSH2、PCNA的表达及其与骨肉瘤临床特征的关系[J]. 中国组织化学与细胞化学杂志, 2016, 25: 30-36. |
| [13] |
黄世超, 王丙武, 陈向阳. 血管内皮生长因子、基质金属蛋白酶在骨肉瘤中的表达及临床意义[J]. 癌症进展, 2019, 17: 1212-1214, 1218. |
| [14] |
MICHALIK K M, YOU X, MANAVSKI Y, DODDABALLAPUR A, ZORNIG M, BRAUN T, et al. Long noncoding RNA MALAT1 regulates endothelial cell function and vessel growth[J]. Circ Res, 2014, 114: 1389-1397. DOI:10.1161/CIRCRESAHA.114.303265 |
 2023, Vol. 44
2023, Vol. 44


