2. 上海中医药大学附属龙华医院肿瘤科,上海 200032;
3. 上海中医药大学附属龙华医院脾胃病研究所,上海 200032;
4. 浙江数字内容研究院数字医疗与人工智能实验室,绍兴 312366
2. Department of Oncology, Longhua Hospital Affiliated to Shanghai University of Traditional Chinese Medicine, Shanghai 200032, China;
3. Institute of Spleen and Stomach Diseases, Longhua Hospital Affiliated to Shanghai University of Traditional Chinese Medicine, Shanghai 200032, China;
4. Laboratory of Digital Health and Artificial Intelligence, Zhejiang Digital Content Research Institute, Shaoxing 312366, Zhejiang, China
肝脏作为人体最重要的器官之一,承担着人体大部分的新陈代谢功能。肝脏病变通常有2种临床类别,一是弥漫性肝脏病变,包括肝硬化、酒精性脂肪肝、非酒精性脂肪性肝病(non-alcoholic fatty liver disease,NAFLD)等;二是肝占位性病变,分为良性病变和恶性病变,其中良性病变包括肝细胞腺瘤、肝海绵状血管瘤、肝脏局灶性结节增生和肝囊肿等,恶性病变包括肝细胞癌、肝内胆管细胞癌、肝转移癌、肝血管肉瘤和肝母细胞瘤等[1]。在NAFLD病变过程中通常会出现3种特征:脂肪变性、炎症和纤维化[2]。病理组织学检查是衡量炎症活动度、脂肪变性和纤维化程度的重要手段[3]。对肝穿刺标本采用H-E染色、嗜银染色和Masson染色三色染色技术,能够准确判断肝组织炎症活动度、脂肪变性及纤维化程度,还可根据实际需要增加天狼猩红染色或免疫组织化学染色等以进一步进行临床研究[4]。
近年来,许多基于CT、MRI等医学影像学技术的研究探索了NAFLD的定量分析特征,这些研究通过传统图像算法或机器学习算法识别医学影像中的病变特征并进行分析[5-8],而病理切片同样能用于NAFLD病理特征的分析研究,但多数研究存在单一、片面等不足。利用数字病理切片开发出一种全面的、准确的自动化NAFLD分析系统,能够为该病的分期和评估提供参考。本研究通过人工智能算法和数字病理切片对NAFLD的3种病理特征(脂肪变性细胞、炎症细胞和纤维化)进行识别,提出基于人工智能算法的NAFLD病理特征识别模型,以帮助病理医师提高识别NAFLD病理特征的效率与准确率。
1 材料和方法 1.1 材料65只NAFLD小鼠的肝组织病理切片由上海中医药大学附属龙华医院宋海燕教授惠赠。所有病理切片均使用匈牙利3D HIETECH公司Pannoramic 250数字化扫描仪进行扫描,获得数字病理切片。数字病理切片纳入标准:(1)病理切片具有脂肪变性细胞或炎症细胞等NAFLD的病理特征;(2)病理切片的评估数据完整、明确;(3)病理切片染色均匀、清晰完整。选择符合纳入标准的H-E染色和天狼猩红染色数字病理切片各65张。
1.2 数据集制作取H-E染色数字病理切片,使用CaseViewer 2.3软件在放大200、300、400倍后截取病变部位图像各2张,每张切片共截取包含脂肪变性细胞的图像6张、包含炎症细胞的图像6张,分辨率均为1 920像素×1 000像素。获得的390张脂肪变性细胞病理图像和390张炎症细胞病理图像构成数据集,用于脂肪变性细胞和炎症细胞的识别研究。
取天狼猩红染色数字病理切片,使用CaseViewer 2.3软件在放大50倍后进行全视野截取,每张切片可截取4~6张有组织纤维化的图像,最终共获得370张纤维化图像用于肝纤维化的识别研究。
1.3 图像标注将数据集中的脂肪变性细胞图像和炎症细胞图像上传至Horizope标注平台,由多名住院医师进行手动标注,标注完成后由1名富有经验的主治医师进行纠错及补充。纤维化的识别采用颜色阈值分割图像算法,无须标注。将Horizope标注平台生成的标注文件(带有标注坐标点信息的JSON文件)通过Python代码转化为可用于深度学习训练的标签图像,格式为PNG。原始图像、Horizope标注平台标注图像及标签图像示例见图 1。

|
图 1 原始图像(A)、Horizope标注平台标注图像(B)及标签图像(C)示例 Fig 1 Examples of original images (A), Horizope platform annotations (B) and label images (C) The upper 3 pictures are pathological images of steatosis cells with hematoxylin-eosin staining (300×) and the lower 3 pictures are pathological images of inflammatory cells with hematoxylin-eosin staining (200×). |
1.4 数据增强
在深度学习训练中,390张图像的数据量用于深度学习模型训练远远不够,容易造成模型欠拟合,因此采用翻转(水平翻转和垂直翻转)、旋转(90°、180°和270°旋转)和高斯噪声等处理方法对原始图像及对应的标签图像进行增强,共得到2 340张脂肪变性细胞病理图像和对应的标签图像,以及2 340张炎症细胞病理图像和对应的标签图像。传统的颜色特征提取算法不需要很大的数据量,因此未对纤维化图像进行增强。
1.5 训练集、验证集与测试集的划分将脂肪变性细胞图像和炎症细胞图像数据集分别按照4∶1∶1划分为训练集、验证集和测试集。训练集包括1 560张脂肪变性细胞图像和1 560张炎症细胞图像,验证集包括390张脂肪变性细胞图像和390张炎症细胞图像,训练集和验证集用于深度学习模型的训练和参数迭代。测试集包括390张脂肪变性细胞图像和390张炎症细胞图像,用于对训练好的深度学习模型进行测试,得到识别结果,并计算模型性能指标。纤维化图像不需要划分为训练集、验证集和测试集。
1.6 人工智能算法对于脂肪变性细胞图像和炎症细胞图像的识别,使用U-Net深度学习模型对训练集和验证集中人工标注的图像进行训练学习和参数迭代。U-Net采用编码-解码结构,是一种全卷积神经网络[9],示意图见图 2。编码器为特征提取网络,由下采样卷积层和池化层组成,每次下采样都会使通道数翻倍;解码器为特征融合网络,由上采样卷积层和下采样卷积层构成。将上采样生成的特征图与编码器中下采样生成的特征图进行融合操作,最后经过2次卷积操作生成特征图。U-Net特殊的结构保证了模型最终得到的特征图融合了更多的低级特征,也使得不同尺度的特征得到了融合,从而可以进行多尺度识别和预测。多次的上采样也使模型能够更加精细、准确地提取分割图像的边缘特征和细节信息[10]。所用的U-Net深度学习模型使用交叉熵作为损失函数,初始学习率设置为0.000 1,最大迭代步长为100,批量大小设置为16,下采样率为0.5,使用Adam优化器更新模型参数。使用Pytorch 1.6框架和Python 3.6环境训练深度学习模型,图形处理器为NVIDIA Gtx 2080ti。
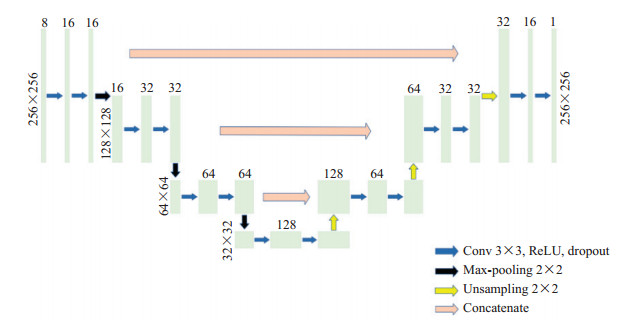
|
图 2 U-Net网络结构示意图 Fig 2 Schematic diagram of U-Net network structure Conv: Convolution; ReLU: Rectified linear unit. |
肝纤维化作为NAFLD的一种病理特征,其形态学特征与炎症细胞和脂肪变性细胞差异显著[11],且通过对小鼠肝组织进行天狼猩红染色发现,经天狼猩红染色后肝纤维化特征尤为明显。因此,为了节约计算机资源,对纤维化特征的识别并未采用深度学习算法,而是采用颜色特征提取这一传统人工智能图像算法。
1.7 基于人工智能算法的NAFLD病理特征识别模型的性能评估为了验证人工智能算法在NAFLD病理特征识别中的有效性,以Horizope标注平台人工标注图像为标准,在测试集脂肪变性细胞图像和炎症细胞图像上,采用4个常用于评价医学图像分割算法精准度的指标对模型性能进行评估,分别是Dice相似系数(Dice’s similarity coefficient,DSC)[12]、平均交互比(mean intersection over union,MIoU)[13]、平均准确度(mean accuracy,MA)和灵敏度:DSC=2TP/(2TP+FP+FN),MIoU=TP/(TP+FP+FN),MA=(TP+TN)/(TP+TN+FP+FN),灵敏度=TP/(TP+FN),其中TP为真阳性样本数,FP为假阳性样本数,TN为真阴性样本数,FN为假阴性样本数。由于纤维化识别采用的是传统图像算法,没有相应的标注图像,因此未进行性能评估。
1.8 病理特征参数的计算通过人工智能算法对130张数字病理切片进行病理特征的识别和参数计算。(1)脂肪变性细胞面积占比(proportion of fatty degeneration cell area,PFA):对65张H-E染色切片放大400倍后进行分析,每张H-E染色切片随机选取10个视野进行脂肪变性细胞识别,并计算脂肪变性细胞面积占视野面积的百分比,取平均值得到局部PFA;再根据视野面积与整张切片面积的比例关系计算得到全局PFA[14]。(2)炎症细胞密度(density of inflammatory cell,DIC):对65张H-E染色切片放大400倍后进行分析,随机选取20个视野进行炎症细胞识别,计算炎症细胞个数与视野面积的比值,取平均值得到局部DIC;再根据视野面积与整张切片面积的比例关系,计算得到全局DIC[14]。(3)纤维化面积占比(ratio of fibrotic area,RFA):对65张天狼猩红染色切片放大50倍后进行全视野截取及纤维化识别,计算纤维化面积占全视野面积的百分比,即为RFA。
由多名住院医师根据NAFLD活动度积分(NAFLD activity score,NAS)半定量评分系统的标准[15]对65张H-E染色数字病理切片进行人工脂肪变性细胞和炎症细胞评分,以及对65张天狼猩红染色数字病理切片进行人工纤维化评分。基于NAS半定量评分系统制定一套适用于人工智能算法识别的评分规则,即机器评分:对65张H-E染色切片根据PFA和DIC分别进行脂肪变性细胞、炎症细胞的评分,PFA<0.06计0分,0.06~0.33计1分,>0.33~0.66计2分,>0.66计3分;DIC<100/mm2计0分,100~400/mm2计1分,>400~700/mm2计2分,>700/mm2计3分。基于NAS半定量评分系统,根据RFA对65张天狼猩红染色切片进行纤维化机器评分,RFA<0.01计0分,0.01~0.07计1分(细分为1A、1B、1C级),>0.07~0.10计2分,>0.10计3分。
1.9 统计学处理应用SPSS 26.0软件进行统计学分析。通过计算偏度和峰度的Z评分对计量资料进行正态性检验,若为正态分布以x±s表示,若为偏态分布以中位数(范围)表示。相关性分析采用Spearman秩相关分析。检验水准(α)为0.05。
2 结果 2.1 基于人工智能算法的NAFLD病理特征识别模型的识别效果在测试集图像上对脂肪变性细胞和炎症细胞进行识别,通过对比标签图像与人工智能算法识别图像可见,人工智能算法对脂肪变性细胞和炎症细胞的识别结果与住院医师手动标注的结果高度一致(图 3)。人工智能算法识别病理特征的性能评估结果显示,在测试集脂肪变性细胞图像上DSC为0.87,MIoU为0.80,MA为0.88,灵敏度为0.84;在测试集炎症细胞图像上DSC为0.84,MIoU为0.78,MA为0.85,灵敏度为0.80。在纤维化病理图像上进行肝纤维化识别,人工智能算法对于肝纤维化的识别准确性较高(图 4)。

|
图 3 人工智能算法对脂肪变性细胞和炎症细胞的识别结果 Fig 3 Recognition results of artificial intelligence algorithm on steatosis and inflammatory cells A: The original pathological images; B: The recognition results of the U-Net deep learning model; C: The annotation images of the resident physicians. The upper 3 pictures are pathological images of steatosis cells with hematoxylin-eosin staining (300×) and the lower 3 pictures are pathological images of inflammatory cells with hematoxylin-eosin staining (200×). |

|
图 4 人工智能算法对肝纤维化的识别结果(50×) Fig 4 Recognition results of artificial intelligence algorithm for liver fibrosis (50×) A: The pathological image with picrosirius red staining; B: The recognition results of the color feature extraction algorithm. |
2.2 病理特征参数分析结果
65张H-E染色数字病理切片中55张切片的脂肪变性细胞机器评分与人工NAS评分相同,吻合率为84.6%。其中机器评分为0分的切片7张,PFA为0.019 4±0.005 6;机器评分为1分的切片22张,PFA为0.181 3±0.085 0;机器评分为2分的切片23张,PFA为0.466 7±0.088 6;机器评分为3分的切片13张,PFA为0.704 5±0.022 2。65张病理切片的PFA为0.371(0.013~0.743)。Spearman秩相关分析显示,PFA与脂肪变性细胞机器评分和人工NAS评分均存在正相关关系(rs=0.953、0.928,P均<0.001)。
65张H-E染色数字病理切片中49张切片的炎症细胞机器评分与人工NAS评分相同,吻合率为75.4%。其中机器评分为0分的切片12张,DIC为(30.25±8.65)/mm2;机器评分为1分的切片32张,DIC为(261.81±80.66)/mm2;机器评分为2分的切片19张,DIC为(519.42±154.67)/mm2;机器评分为3分的切片2张,平均DIC为870.00/mm2。65张病理切片的DIC为288(19~894)/mm2。Spearman秩相关分析显示,DIC与炎症细胞机器评分和人工NAS评分均存在正相关关系(rs=0.883、0.869,P均<0.001)。
65张天狼猩红染色数字病理切片中52张切片的纤维化机器评分与人工NAS评分相同,吻合率为80.0%。其中机器评分为0分的切片5张,RFA为0.002 4±0.000 9;机器评分为1A的切片9张,RFA为0.018 6±0.005 4;机器评分为1B的切片17张,RFA为0.040 7±0.004 7;机器评分为1C的切片26张,RFA为0.059 6±0.005 8;机器评分为2分的切片6张,RFA为0.083 2±0.004 6;机器评分为3分的切片2张,平均RFA为0.118 0。65张病理切片的RFA为0.048 5±0.025 4。Spearman秩相关分析显示,RFA与纤维化机器评分和人工NAS评分均呈正相关(rs=0.887、0.749,P均<0.001)。
3 讨论病理组织学检查是明确诊断、衡量炎症活动度、脂肪变性和纤维化程度,以及判定药物疗效的重要依据[3]。病理医师对病理图像分析与结果判断是否准确,在很大程度上取决于自身的临床经验,这不仅是一个费时、费力的过程,而且易出现漏诊,甚至对于同一张病理图像,不同的医师由于经验、精神状态等主、客观因素的影响可能会做出不同的诊断结论。因此,急需深度学习等新技术使高水平医疗资源惠及大众,在减轻医师负担的同时辅助诊断,提高医疗服务水平。
NAFLD的病理诊断是其分期、分型的关键环节之一,诊断结果影响着临床诊疗方案的制定。近年来,人工智能被越来越多地应用于NAFLD的研究[16],随着全玻片数字化扫描仪的使用,基于全视野数字切片(whole slide image,WSI)的NAFLD病理特征分析的研究逐渐增多。2019年,荷兰拉德堡德大学的研究人员在肝脏组织WSI上开发了脂肪变性的数字化自动定量方法,进一步验证发现该方法能够自动计算PFA[17]。2021年,Taylor-Weiner等[18]提出了一种基于机器学习的肝脏组织学评估方法,该方法通过机器学习算法测量脂肪变性、炎症、气球样变和纤维化程度,以准确评估NAFLD的严重程度和异质性。但是,当前的病理人工智能算法系统面临着临床应用困难的问题。本研究亦探索了深度学习模型能否对NAFLD的病理图像进行分析,并可视化和定量其病理特征,结果显示深度学习模型能够准确评估NAFLD的严重程度和异质性,这将为NAFLD的药物研发、生物标志物识别及基础医学机制研究提供思路和帮助。
利用人工智能算法识别NAFLD能够减轻医师的工作量、提高诊断精准度和杜绝造假的可能性。此外,定量和可视化病理特征也会对NAFLD的评估产生积极影响。根据肉眼观察到的特征进行评分极具主观性,因此利用人工智能算法定量数据进行分级是未来研究的重点。值得一提的是,在基于人工智能算法识别脂肪变性细胞、炎症细胞和纤维化,并根据得到的PFA、DIC、RFA对所有病理切片参照NAS评分系统进行机器评分后发现,PFA、DIC、RFA与通过人工智能算法得到的机器评分和人工NAS评分之间均呈正相关,验证了相关算法的有效性,然而3个病理特征的机器评分与人工NAS评分之间存在不一致的现象,吻合率均在80%左右,原因可能是传统的人工NAS评分不够精准和本研究纳入数据量较小,具体原因有待进一步研究。
模拟人类NAFLD的小鼠模型不仅为研究NAFLD的发病机制提供了有力的研究工具,也为相关药物的开发提供了良好的实验模型。然而任何动物模型的应用都无法很好地对人类疾病进行表征,本研究仅使用了模型小鼠病理切片,研究结果还有待进一步验证,并且只有在动物模型上实现有效性和安全性并获得足够的数据支持后才能进一步申请开展临床试验。
基于人工智能算法的NAFLD病理特征识别模型在NAFLD病理检查中可准确识别病理特征,能够帮助病理医师提高识别NAFLD病理特征的效率与准确率,准确评估疾病严重程度和异质性,以及进行正确的疾病分级与分期和疗效评估,从而提高诊断准确性和客观性。
| [1] |
LAING R W, MIRZA D F. Liver lesions: types, risk factors, investigations and treatment[J]. Trend Urol Mens Heal, 2020, 11: 20-25. |
| [2] |
MURPHY P, HOOKER J, ANG B, WOLFSON T, GAMST A, BYDDER M, et al. Associations between histologic features of nonalcoholic fatty liver disease (NAFLD) and quantitative diffusion-weighted MRI measurements in adults[J]. J Magn Reson Imaging, 2015, 41: 1629-1638. DOI:10.1002/jmri.24755 |
| [3] |
Working Group, Association of Pathologists for Guidebook of NASH and NAFLD, 2015: The Japan Society of Hepatology. Pathological findings of NASH and NAFLD: for guidebook of NASH and NAFLD, 2015: the Japan Society of Hepatology[J]. Hepatol Res, 2017, 47: 3-10. DOI:10.1111/hepr.12847 |
| [4] |
BRUNT E M. Pathology of nonalcoholic fatty liver disease[J]. Nat Rev Gastroenterol Hepatol, 2010, 7: 195-203. DOI:10.1038/nrgastro.2010.21 |
| [5] |
FENG G, ZHENG K I, LI Y Y, RIOS R S, ZHU P W, PAN X Y, et al. Machine learning algorithm outperforms fibrosis markers in predicting significant fibrosis in biopsy-confirmed NAFLD[J]. J Hepatobiliary Pancreat Sci, 2021, 28: 593-603. DOI:10.1002/jhbp.972 |
| [6] |
DINANI A M, KOWDLEY K V, NOUREDDIN M. Application of artificial intelligence for diagnosis and risk stratification in NAFLD and NASH: the state of the art[J]. Hepatology, 2021, 74: 2233-2240. DOI:10.1002/hep.31869 |
| [7] |
KEZER C A, SHAH V H, SIMONETTO D A. Advances in predictive modeling using machine learning in the field of hepatology[J]. Clin Liver Dis (Hoboken), 2021, 18: 288-291. DOI:10.1002/cld.1148 |
| [8] |
LEE H W, SUNG J J Y, AHN S H. Artificial intelligence in liver disease[J]. J Gastroenterol Hepatol, 2021, 36: 539-542. DOI:10.1111/jgh.15409 |
| [9] |
RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]//Lecture Notes in Computer Science book series (LNIP, volume 9351). Cham: Springer, 2015: 234-241.
|
| [10] |
LI X M, CHEN H, QI X J, DOU Q, FU C W, HENG P A. H-DenseUNet: hybrid densely connected UNet for liver and tumor segmentation from CT volumes[J]. IEEE Trans Med Imaging, 2018, 37: 2663-2674. DOI:10.1109/TMI.2018.2845918 |
| [11] |
MACK M. Inflammation and fibrosis[J]. Matrix Biol, 2018, 68/69: 106-121. DOI:10.1016/j.matbio.2017.11.010 |
| [12] |
SHAMIR R R, DUCHIN Y, KIM J, SAPIRO G, HAREL N. Continuous Dice coefficient: a method for evaluating probabilistic segmentations[J/OL]. arXiv: 1906.11031 (2019-06-26)[2022-04-13]. https://doi.org/10.48550/arXiv.1906.11031.
|
| [13] |
BERMAN M, TRIKI A R, BLASCHKO M B. The Lovász-Softmax loss: a tractable surrogate for the optimization of the intersection-over-union measure in neural networks[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Salt Lake City: IEEE, 2018: 4413-4421.
|
| [14] |
董伟. 肝细胞癌患者新型肝硬化病理分级评分系统的建立及临床病理学意义的研究[D]. 上海: 第二军医大学, 2017.
|
| [15] |
MICHELOTTI G A, MACHADO M V, DIEHL A M. NAFLD, NASH and liver cancer[J]. Nat Rev Gastroenterol Hepatol, 2013, 10: 656-665. DOI:10.1038/nrgastro.2013.183 |
| [16] |
HEINEMANN F, BIRK G, STIERSTORFER B. Deep learning enables pathologist-like scoring of NASH models[J/OL]. Sci Rep, 2019, 9: 18454. DOI: 10.1038/s41598-019-54904-6.
|
| [17] |
MUNSTERMAN I D, VAN ERP M, WEIJERS G, BRONKHORST C, DE KORTE C L, DRENTH J P H, et al. A novel automatic digital algorithm that accurately quantifies steatosis in NAFLD on histopathological whole-slide images[J]. Cytometry B Clin Cytom, 2019, 96: 521-528. DOI:10.1002/cyto.b.21790 |
| [18] |
TAYLOR-WEINER A, POKKALLA H, HAN L, JIA C, HUSS R, CHUNG C, et al. A machine learning approach enables quantitative measurement of liver histology and disease monitoring in NASH[J]. Hepatology, 2021, 74: 133-147. DOI:10.1002/hep.31750 |
 2022, Vol. 43
2022, Vol. 43


