原发性肝癌(主要为肝细胞癌,以下简称肝癌)是我国常见的恶性肿瘤,其死亡率逐年升高,发病率在恶性肿瘤中居第4位,死亡率位居肿瘤相关死亡的第3位,是60岁以下男性最常见且死亡率最高的肿瘤,是肝硬化相关死亡的首因[1]。肝癌的发生、发展受一系列遗传因素和环境因素的共同影响,发病机制十分复杂。目前肝癌临床治疗的方法除放射治疗、化疗、手术治疗及生物治疗外,还有介入疗法、局部消融疗法、分子靶向治疗等[2]。但肝癌的治愈率很低,肝癌患者手术后复发率很高,术后5年生存率多不超过40%,复发率高达80%~90%[3-4],且复发多发生在术后1~2年[5]。深入研究肝癌复发的具体分子机制对于肝癌和肝癌复发的治疗与预后有重要意义。本研究采用生物信息学方法分析肝癌复发患者的基因表达数据,筛选出肝癌复发患者的差异表达基因,并对差异基因进行功能注释与通路富集分析、核心基因鉴别、预后分析,旨在发现新的肝癌复发生物标志物。
1 材料和方法 1.1 数据下载通过癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库(https://portal.gdc.cancer.gov/)下载374例肝癌患者(复发性肝癌患者202例、原发性肝癌患者172例)和50例健康对照的基因数据与临床信息。通过基因表达汇编(Gene Expression Omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)GSE76427数据集中下载115例肝癌组织样本和52例癌旁正常组织样本的基因表达数据与临床信息。
1.2 差异表达基因筛选利用edgeR算法计算102个正常肝组织样本与489个肝癌组织样本、172个原发性肝癌样本与202个复发性肝癌样本的差异表达基因{以校正P值[采用错误发现率(false discovery rate,FDR)校正]<0.05且log2|FC|>1为筛选标准,其中FC为差异倍数(fold change)},将此两组差异表达基因取交集,筛选出交集基因。
1.3 基因本体(Gene Ontology,GO)与京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析采用R 3.6.1软件clusterProfiler和enrichplot工具对差异表达基因进行GO功能分析与KEGG通路分析,得出交集基因的生物学功能,设定FDR P<0.05。
1.4 核心基因筛选及其预后分析利用STRING数据库(http://string.embl.de)对交集基因构建蛋白质-蛋白质相互作用网络图,设最低关联度为0.4,采用Cytoscape软件筛选核心基因。通过Kaplan-Meier Plotter在线分析工具(http://kmplot.com/analysis/)分析核心基因与肝癌预后的关系。
1.5 免疫细胞浸润分析利用单样本基因集富集分析(single sample gene set enrichment analysis,ssGSEA)[6]、使用表达数据估计恶性肿瘤组织中的基质细胞和免疫细胞(estimation of stromal and immune cells in malignant tumor tissues using expression data,ESTIMATE)[7]、微环境细胞种群计数器(microenvironment cell populations-counter,MCP)[8]方法分别通过R 3.6.1软件GSVA、estimate和MCPcounter工具对筛选出的与肝癌复发相关的基因进行免疫细胞浸润分析。采用ProteinAtlas数据库(https://www.proteinatlas.org/)进行免疫组织化学评测,以发掘肝癌复发的潜在机制。
1.6 最小绝对收缩和选择算子(least absolute shrinkage and selection operator,LASSO)-logistic回归预测模型构建使用R 3.6.1软件glmnet工具构建LASSO-logistic回归模型,筛选核心基因中影响肝癌复发的关键基因,并对每例肝癌患者复发风险进行评分。评分系统构建完成后,用ROC曲线的AUC值评估预测模型的可靠性。使用R 3.6.1软件中的rms工具构建列线图并可视化预测模型。
2 结果 2.1 差异表达基因分析结果通过筛选正常肝组织与肝癌组织的差异表达基因得到4 576个差异基因,其中上调基因3 866个、下调基因710个。将原发性肝癌患者与复发性肝癌患者基因表达谱进行比较,筛选得到455个差异表达基因,其中上调基因235个、下调基因220个。将这两组差异表达基因取交集得到343个差异表达基因,其中上调基因321个、下调基因22个。
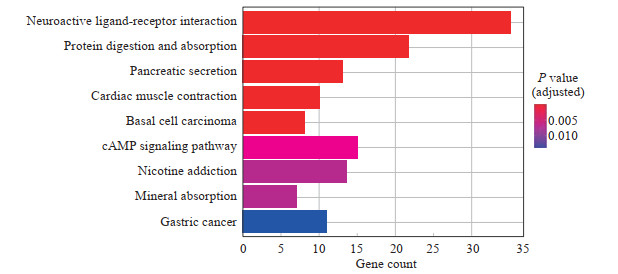
2.2 差异表达基因GO功能及KEGG通路富集分析对筛选的343个差异基因进行GO功能分析,结果显示差异表达基因主要涉及前脑发育、化学突触传递的调节、反式突触信号传导的调节等生物过程,突触膜、神经元细胞体、突触后膜等细胞组分,受体配体活性、信号受体激活活性、门控通道活性等分子功能(图 1)。KEGG通路分析结果显示,差异表达基因参与的主要信号通路富集于神经激活配体-受体相互作用、蛋白质的消化与吸收、cAMP信号通路等(图 2)。

|
图 1 基因本体分析揭示肝癌复发相关差异表达基因的潜在功能 Fig 1 Gene Ontology analysis reveals potential functions of differentially expressed genes related to liver cancer recurrence |
|
图 2 KEGG分析明确肝癌复发相关差异表达基因的富集通路 Fig 2 KEGG analysis identifies enrichment pathways of differentially expressed genes related to liver cancer recurrence KEGG: Kyoto Encyclopedia of Genes and Genomes; cAMP: Cyclic adenosine monophosphate. |
2.3 复发相关核心差异基因筛选
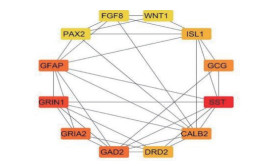
通过STRING数据库对343个差异表达基因进行蛋白质-蛋白质相互作用网络分析,构建复发性肝癌的核心基因网络,筛选出生长抑素(somatostatin,SST)、谷氨酸脱羧酶2(glutamate decarboxylase 2,GAD2)、离子型谷氨酸受体AMPA亚基2(glutamate ionotropic receptor AMPA type subunit 2,GRIA2)、胰高血糖素(glucagon,GCG)、多巴胺D2受体(dopamine receptor D2,DRD2)、胰岛素基因增强子结合蛋白1(insulin gene enhancer binding protein 1,ISL1)、无翅型MMTV整合位点家族成员1(wingless-type MMTV integration site family,member 1,WNT1)、成纤维细胞生长因子8(fibroblast growth factor 8,FGF8)、配对基因盒2(paired box 2,PAX2)、离子型谷氨酸受体NMDA亚基1 (glutamate ionotropic receptor NMDA type subunit 1,GRIN1)、胶质原纤维酸性蛋白(glial fibrillary acidic protein,GFAP)、钙结合蛋白2(calbindin 2,CALB2)等12个核心基因(图 3)。
|
图 3 肝癌复发相关核心基因蛋白质-蛋白质相互作用网络图 Fig 3 Protein-protein interaction network diagram of hub genes related to liver cancer recurrence WNT1: Wingless-type MMTV integration site family, member 1; ISL1: Insulin gene enhancer binding protein 1; GCG: Glucagon; SST: Somatostatin; CALB2: Calbindin 2; DRD2: Dopamine receptor D2; GAD2: Glutamate decarboxylase 2; GRIA2: Glutamate ionotropic receptor AMPA type subunit 2; GRIN1: Glutamate ionotropic receptor NMDA type subunit 1; GFAP: Glial fibrillary acidic protein; PAX2: Paired box 2; FGF8: Fibroblast growth factor 8. |
2.4 复发性肝癌患者的核心基因表达与预后分析
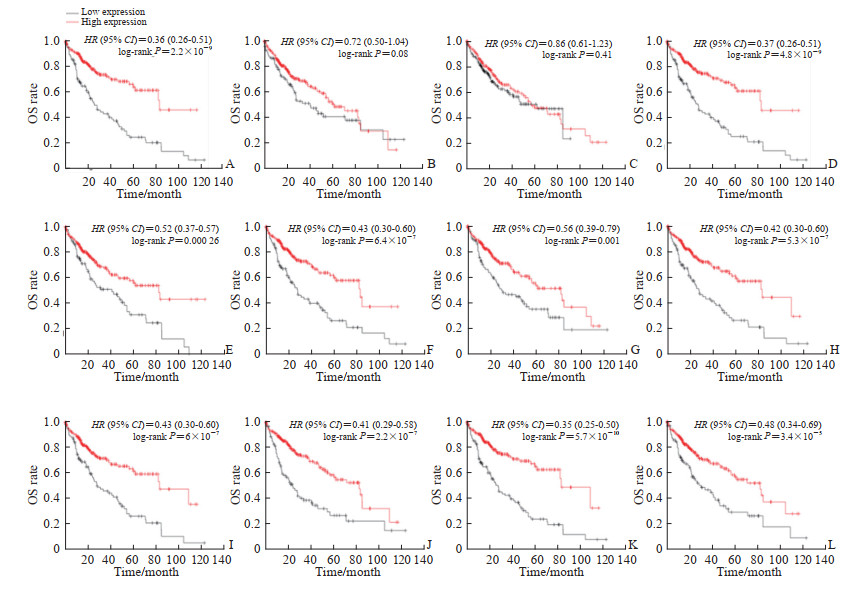
通过Kaplan-Meier Plotter在线分析工具对12个肝癌复发相关核心基因进行预后分析,结果显示SST、GAD2、GRIA2、GCG、DRD2、ISL1、WNT1、FGF8、PAX2、CALB2高表达组患者的预后均优于低表达组(按基因的中位值分组,P<0.01),GRIN1、GFAP的高、低表达组之间总生存时间差异无统计学意义(图 4)。
|
图 4 Kaplan-Meier Plotter构建12个肝癌复发相关核心基因预后分析图 Fig 4 Prognostic analysis of 12 hub genes of liver cancer recurrence was generated by Kaplan-Meier Plotter A: Somatostatin; B: Glutamate ionotropic receptor NMDA type subunit 1; C: Glial fibrillary acidic protein; D: Glutamate decarboxylase 2; E: Glutamate ionotropic receptor AMPA type subunit 2; F: Glucagon; G: Calbindin 2; H: Dopamine receptor D2; I: Insulin gene enhancer binding protein 1; J: Wingless-type MMTV integration site family, member 1; K: Fibroblast growth factor 8; L: Paired box 2. HR: Hazard ratio; CI: Confidence interval; OS: Overall survival. |
2.5 肝癌复发相关核心基因的LASSO-logistic预后模型构建
通过LASSO-logistic回归分析对核心基因进行降维,确定了最佳λ为0.090 9(图 5A)。筛选高度相关基因后,在GEO数据集中选择了3个肝癌复发相关基因(SST、DRD2、CALB2)进行建模:肝癌复发风险评分=22.1-SST表达值×0.21-DRD2表达值×1.94-CALB2表达值×1.153。对患者的临床病理特征及复发风险评分进行Cox回归分析,结果显示该复发风险评分可作为肝癌患者复发的独立评价指标(图 5B)。该评分模型ROC曲线AUC值为0.683(图 5C)。结合复发风险评分和临床相关变量构建的列线图(图 5D)显示,总评分能准确预测肝癌患者的复发风险。校准曲线(图 5E)与实际观测结果拟合性较好,平均误差为0.041。

|
图 5 LASSO-logistic肝癌复发预测模型构建 Fig 5 Construction of LASSO-logistic liver cancer recurrence prediction model A: Construction of LASSO-logistic model; B: Risk score could serve as an independent risk factor in predicting patient recurrence. C: ROC curve showed prediction efficiency of scoring system GEO dataset; D-E: Development of nomograms (D) and calibration curves (E) for prognosis in total dataset. The number above in figure 5A represents the number of genes that build the model. The dotted line on the left indicates λ.min, which refers to the one that obtains the mean value of the minimum target parameter among all the λ values. The right dotted line represents λ.1se, which refers to the λ value of the simplest model within a variance range of λ.min. The λ between these 2 values is considered appropriate. LASSO: Least absolute shrinkage and selection operator; ROC: Receiver operating characteristic; GEO: Gene Expression Omnibus; AUC: Area under curve; BCLC: Barcelona clinic liver cancer; HR: Hazard ratio; CI: Confidence interval. |
2.6 CALB2与肝癌复发患者肿瘤微环境免疫细胞浸润密切相关
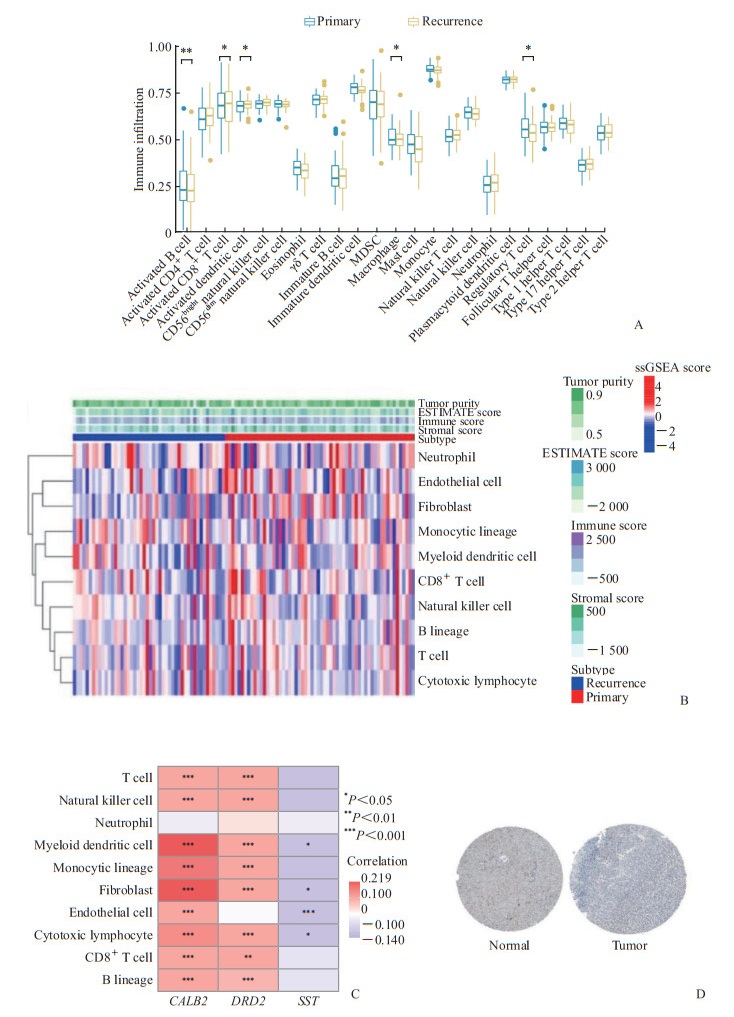
通过ssGSEA、MCP和ESTIMATE方法对比原发性与复发性肝癌患者肿瘤微环境中免疫细胞,原发性肝癌患者肿瘤微环境的活化B细胞、活化CD8+ T细胞、活化树突状细胞、巨噬细胞、调节性T细胞等有富集浸润(图 6A、6B)。对复发风险评分模型中的3个核心基因的分析结果显示,CALB2与复发性肝癌患者的免疫细胞浸润呈正相关(图 6C)。免疫组化结果显示,与正常肝组织样本相比,肿瘤组织样本中CALB2表达降低(图 6D)。以上结果表明,CALB2相对高表达的患者肿瘤微环境中免疫细胞浸润增多,CALB2的表达增高使肝癌患者的预后更佳、复发概率更低。
|
图 6 CALB2在肝癌免疫微环境中影响免疫细胞浸润的作用 Fig 6 Effect of CALB2 on immune cell infiltration in immune microenvironment of liver cancer A: ssGSEA indicated the immune cell infiltration between 2 different groups; B: MCP indicated the correlation of immune cell infiltration and hub genes; C: Correlation between 3 hub genes and immune cell infiltration; D: Immunohistochemical staining of CALB2. CALB2: Calbindin 2; MDSC: Myeloid-derived suppressor cell; DRD2: Dopamine receptor D2; SST: Somatostatin; ESTIMATE: Estimation of stromal and immune cells in malignant tumour tissues using expression data; ssGSEA: Single sample gene set enrichment analysis; MCP: Microenvironment cell populations-counter. |
3 讨论
为进一步研究与肝癌复发相关基因,基于STRING数据库对筛选出的肝癌复发相关差异表达基因进行蛋白质-蛋白质相互作用分析,筛选出12个最可能引起肝癌复发的核心差异表达基因:SST、GRIN1、GFAP、GAD2、GRIA2、GCG、CALB2、DRD2、ISL1、WNT1、FGF8、PAX2。
FGF8是成纤维细胞生长因子(fibroblast growth factors,FGFs)家族的成员之一[9],其在人胚胎时期多种组织内进行表达,在各种器官的形成中起着重要的作用。研究表明,在正常成人体内FGF8的表达水平受到严格的限制,但在某些癌细胞或炎症部位中大量表达,对癌症的发生和发展中起着重要作用[9]。
裴哲等[10]报道Wnt信号的异常持续激活可导致肝癌的发生、发展。由于WNT1-Wnt信号通路富集靶基因最显著,闫瑞承等[11]通过蛋白质印迹实验中WNT3A和磷酸化β-连环蛋白低表达,证实了经典Wnt信号通路参与了维生素K2联合磷脂酰胆碱抑制人肝癌细胞的过程。
ISL1被认为是一个胚胎性基因。成年后ISL1只在特定的组织或器官中表达,如胰腺和大脑。近年来在多种肿瘤中检测到ISL1的异常表达,不仅在胰腺内分泌肿瘤、神经肿瘤中高表达(与之相应的正常组织也表达ISL1),还在淋巴瘤、胃癌和膀胱癌等肿瘤组织中表达(相对应的正常组织不表达ISL1),表明ISL1的异常表达可能与肿瘤的发生、发展及预后相关[12]。以上3个基因都已有文献报道,与肿瘤的发生、发展有着密切联系,其余的基因还需进一步验证。
通过对12个核心基因构建LASSO-logistic复发预测模型,构建了基于3个肝癌复发相关核心基因的评分系统。该评分系统的风险评分显示了较好的预测效能,并可以作为复发的独立预测因子。
近年来,肿瘤微环境已成为当前肿瘤研究的热点,微环境中的免疫细胞浸润与肝癌复发及转移密切相关[13]。肿瘤微环境指肿瘤发生、发展过程中所处的环境,由多种细胞成分、细胞外基质和细胞因子等组成[14]。其中细胞成分包括各种类型免疫细胞,如T淋巴细胞、B淋巴细胞、肿瘤相关巨噬细胞、树突状细胞和自然杀伤细胞等[15]。肿瘤组织中免疫细胞浸润与肿瘤的预后、转移及治疗反应密切相关。研究表明CD8+T细胞及调节性T细胞在肿瘤内的浸润程度可作为肝癌患者的独立预后指标,且与免疫检查点抑制剂的治疗效果密切相关[16-18]。此外,肿瘤内浸润较多的树突状细胞也成为预测肝癌患者无病生存期的标志物[19]。因此,探索影响复发性肝癌组织中微环境免疫细胞浸润的机制将为深入理解肝癌复发提供新的方向。通过ESTIMATE、ssGSEA及MCP等算法对原发性与复发性患者免疫细胞浸润情况进行比较,发现原发性肝癌患者肿瘤微环境的活化B细胞、活化CD8+T细胞、活化树突状细胞、巨噬细胞、调节性T细胞等有富集浸润,这提示了肝癌复发与缺乏多种类型的免疫细胞浸润相关。
CALB2是Ca2+结合蛋白的EF-hand蛋白家族的成员,在多种肿瘤中具有重要作用[20-21]。Vonlanthen等[22]研究表明,CALB2在抑制结肠癌发生、发展中发挥着重要作用。CALB2 是结肠癌细胞中丁酸盐负调控的重要基因,可以作为结肠癌的预后标志物。对3个肝癌复发相关核心基因深入分析发现,CALB2与肿瘤微环境中的免疫细胞浸润密切相关,CALB2高表达与T细胞、CD8+T细胞、B细胞、自然杀伤细胞等免疫细胞浸润呈正相关。同时,免疫组织化学染色结果显示,CALB2在癌组织中的表达低于癌旁正常组织。以上结果表明,肝癌复发患者中存在较低的免疫细胞浸润,其可能与CALB2的表达密切相关。
综上所述,本文通过选取正常组织样本与肝癌组织样本、原发性肝癌样本与复发性肝癌样本的两组基因数据,筛选出343个肝癌复发相关基因,初探了它们的生物学过程与分子通路,再通过蛋白质-蛋白质相互作用最终筛选出12个可能与肝癌复发相关的核心基因,通过LASSO-logistic模型筛选出3个与肝癌复发密切相关的基因。最后探索了原发性肝癌与复发性肝癌患者中肿瘤微环境免疫细胞浸润的差异及核心基因与免疫细胞浸润的关系,为研究肝癌复发提供了新的靶点。然而,本研究主要基于生物信息学分析,关于CALB2对肝癌复发的分子机制还需要进一步研究与探索。
| [1] |
DUTTA R, MAHATO R I. Recent advances in hepatocellular carcinoma therapy[J]. Pharmacol Ther, 2017, 173: 106-117. DOI:10.1016/j.pharmthera.2017.02.010 |
| [2] |
胡琦, 朱克俭. 中药有效成分抗肝癌复发转移的实验研究进展[J]. 湖南中医药大学学报, 2015, 35: 60-63. |
| [3] |
CHUMA M, TERASHITA K, SAKAMOTO N. New molecularly targeted therapies against advanced hepatocellular carcinoma: from molecular pathogenesis to clinical trials and future directions[J/OL]. Hepatol Res, 2015, 45: E1-E11. DOI: 10.1111/hepr.12459.
|
| [4] |
LLOVET J M, VILLANUEVA A, LACHENMAYER A, FINN R S. Advances in targeted therapies for hepatocellular carcinoma in the genomic era[J]. Nat Rev Clin Oncol, 2015, 12: 408-424. DOI:10.1038/nrclinonc.2015.103 |
| [5] |
王波, 钱叶本, 罗雪莲, 金雯, 朱治文. 肝癌复发的诊断与治疗进展[J]. 肝胆外科杂志, 2016, 24: 307-312. |
| [6] |
XIAO B, LIU L Y, LI A Y, XIANG C, WANG P X, LI H, et al. Identification and verification of immune-related gene prognostic signature based on ssGSEA for osteosarcoma[J/OL]. Front Oncol, 2020, 10: 607622. DOI: 10.3389/fonc.2020.607622.
|
| [7] |
XU M, LI Y, LI W H, ZHAO Q Y, ZHANG Q L, LE K H, et al. Immune and stroma related genes in breast cancer: a comprehensive analysis of tumor microenvironment based on The Cancer Genome Atlas (TCGA) database[J/OL]. Front Med (Lausanne), 2020, 7: 64. DOI: 10.3389/fmed.2020.00064.
|
| [8] |
ZHANG X Q, REN L H, YAN X Y, SHAN Y, LIU L, ZHOU J, et al. Identification of immune-related lncRNAs in periodontitis reveals regulation network of gene-lncRNA-pathway-immunocyte[J/OL]. Int Immunopharmacol, 2020, 84: 106600. DOI: 10.1016/j.intimp.2020.106600.
|
| [9] |
王一, 田海山, 李校堃. 成纤维细胞生长因子8(FGF8)研究进展[J]. 中国生物工程杂志, 2011, 31: 75-80. |
| [10] |
裴哲, 曾中秋, 陈雨文, 李文华, TANG Y J, 唐亚雄. 肝癌Wnt通路可溶性跨膜受体Frizzled-7稳定表达细胞株的构建与鉴定[J]. 应用与环境生物学报, 2015, 21: 1076-1079. |
| [11] |
闫瑞承, 罗建飞, 沈世强. 维生素K2联合磷脂酰胆碱抑制人肝癌细胞作用机制的生物信息学分析[J]. 中华实验外科杂志, 2019, 36: 1738-1740. |
| [12] |
石琼, 王卫平. 胰岛素增强子结合蛋白-1(ISL1)与肿瘤的发生与发展[J]. 中国生物化学与分子生物学报, 2017, 33: 242-246. |
| [13] |
SUN Y F, WU L, ZHONG Y, ZHOU K Q, HOU Y, WANG Z F, et al. Single-cell landscape of the ecosystem in early-relapse hepatocellular carcinoma[J/OL]. Cell, 2021, 184: 404-421. e16. DOI: 10.1016/j.cell.2020.11.041.
|
| [14] |
YANG J D, NAKAMURA I, ROBERTS L R. The tumor microenvironment in hepatocellular carcinoma: current status and therapeutic targets[J]. Semin Cancer Biol, 2011, 21: 35-43. DOI:10.1016/j.semcancer.2010.10.007 |
| [15] |
TAHMASEBI BIRGANI M, CARLONI V. Tumor microenvironment, a paradigm in hepatocellular carcinoma progression and therapy[J/OL]. Int J Mol Sci, 2017, 18: 405. DOI: 10.3390/ijms18020405
|
| [16] |
HUANG C Y, WANG Y, LUO G Y, HAN F, LI Y Q, ZHOU Z G, et al. Relationship between PD-L1 expression and CD8+ T-cell immune responses in hepatocellular carcinoma[J]. J Immunother, 2017, 40: 323-333. DOI:10.1097/CJI.0000000000000187 |
| [17] |
FLECKEN T, SCHMIDT N, HILD S, GOSTICK E, DROGNITZ O, ZEISER R, et al. Immunodominance and functional alterations of tumor-associated antigen-specific CD8+ T-cell responses in hepatocellular carcinoma[J]. Hepatology, 2014, 59: 1415-1426. DOI:10.1002/hep.26731 |
| [18] |
LANGHANS B, NISCHALKE H D, KRÄMER B, DOLD L, LUTZ P, MOHR R, et al. Role of regulatory T cells and checkpoint inhibition in hepatocellular carcinoma[J]. Cancer Immunol Immunother, 2019, 68: 2055-2066. DOI:10.1007/s00262-019-02427-4 |
| [19] |
CAI X Y, GAO Q, QIU S J, YE S L, WU Z Q, FAN J, et al. Dendritic cell infiltration and prognosis of human hepatocellular carcinoma[J]. J Cancer Res Clin Oncol, 2006, 132: 293-301. DOI:10.1007/s00432-006-0075-y |
| [20] |
BLUM W, PECZE L, RODRIGUEZ J W, STEINAUER M, SCHWALLER B. Regulation of calretinin in malignant mesothelioma is mediated by septin 7 binding to the CALB2 promoter[J/OL]. BMC Cancer, 2018, 18: 475. DOI: 10.1186/s12885-018-4385-7.
|
| [21] |
TU S Q, WU J Y, CHEN L L, TIAN Y G, QIN W, HUANG S H, et al. LncRNA CALB2 sponges miR-30b-3p to promote odontoblast differentiation of human dental pulp stem cells via up-regulating RUNX2[J/OL]. Cell Signal, 2020, 73: 109695. DOI: 10.1016/j.cellsig.2020.109695.
|
| [22] |
VONLANTHEN S, KAWECKI T J, BETTICHER D C, PFEFFERLI M, SCHWALLER B. Heterozygosity of SNP513 in intron 9 of the human calretinin gene (CALB2) is a risk factor for colon cancer[J]. Anticancer Res, 2007, 27(6C): 4279-4288. |
 2022, Vol. 43
2022, Vol. 43


