2. 海军军医大学(第二军医大学)第一附属医院放疗科, 上海 200433
2. Department of Radiotherapy, The First Affiliated Hospital of Naval Medical University (Second Military Medical University), Shanghai 200433, China
胰腺癌恶性程度极高,5年总体生存率仅约10%[1]。胰腺癌的预后与肿块的大小密切相关,小胰腺癌(最大径≤2 cm的胰腺癌)预后较好,当肿瘤大小超过2 cm时,胰腺癌患者的预后明显变差[2]。在所有被检出的胰腺癌中小胰腺癌占比为3%~18%[2-4]。及时、准确诊断为患者争取手术治疗的机会,可显著改善胰腺癌患者的预后,但准确、及时检出小胰腺癌极其困难。首先,大多数小胰腺癌患者缺乏临床症状或仅有腹痛、背痛等非特异性症状[3, 5],常错过最佳检出时机。其次,让所有人接受胰腺CT增强检查进行胰腺癌筛查不切实际,会造成大量医疗资源浪费。再者,即便患者接受了腹部CT检查,小胰腺癌的漏诊率仍很高,约为40%[6]。
影像组学在分析影像、识别病变方面具有得天独厚的优势,相关技术已被用于胰腺肿瘤的诊断、鉴别诊断、预后预测、疗效评估及小胰腺癌的筛查[7]。但目前研究所用CT图像多为增强图像、放射剂量大、不适用因过敏或肾功能不全等无法行增强CT检查的人群,或所纳入的肿瘤体积较大不易漏诊,筛查小胰腺癌的临床价值有限。因此,亟须改进现有的检测手段来提高小胰腺癌的检出率。本研究拟探讨基于腹部CT平扫图像的影像组学能否准确检出小胰腺癌。
1 资料和方法 1.1 研究对象在海军军医大学(第二军医大学)第一附属医院病理登记系统检索、收集2014年1月至2021年12月经手术病理证实的小胰腺癌患者资料,共297例。排除标准:(1)术前1个月内未行胰腺多层螺旋CT检查的患者(12例);(2)胰管或胆管支架植入术后患者(8例);(3)术前接受过新辅助治疗的患者(20例);(4)伴胰腺导管内乳头状黏液性肿瘤的患者(2例);(5)图像部分缺失者(49例)。最终206例小胰腺癌患者纳入研究。
在海军军医大学(第二军医大学)第一附属医院影像存储与传输系统中检索胰腺正常者的资料。检索条件:(1)检查时间为2014年1月至2021年12月;(2)检查项目为上腹部CT增强、胰腺CT平扫+增强扫描和胰腺动脉CT增强造影;(3)报告结论为未见明显异常;(4)年龄>40岁。共检索到2 194条记录。由2名有10年以上腹部放射学诊断经验的主治医师重新评估患者的影像报告或图像,必要时查阅患者的电子病历,排除有胰腺或其他部位占位性病变、恶性肿瘤病史或因其他疾病需住院治疗的患者。最终纳入胰腺正常者268例。
将研究对象按时间顺序分成训练集(2014年1月至2019年12月收治的143例小胰腺癌患者和188例胰腺正常者)和验证集(2020年1月至2021年12月收治的63例小胰腺癌患者和80例胰腺正常者)。
本研究通过海军军医大学(第二军医大学)第一附属医院伦理委员会审批。
1.2 CT检查方法采用640层CT(Aquilion ONE,佳能医疗,东京,日本)和256层CT[Brilliance iCT,美国飞利浦医疗系统(克利夫兰)公司]行螺旋扫描。扫描参数:管电压120 kV,管电流150 mA,准直100×0.5 mm,矩阵350×350,旋转时间0.5 s。平扫结束后进行动态增强扫描,造影剂为碘普罗胺注射液(优维显370,德国拜耳先灵葆雅制药公司)90~95 mL(含碘355 mg/mL),注射速率5.5 mL/s。3期增强分别为动脉期(20~25 s)、门静脉期(60~70 s)和延迟期(110~130 s),注射结束给予98 mL生理盐水冲洗。扫描层厚为0.8 mm,层间距为1.0 mm,扫描范围为膈顶到盆腔。
1.3 影像组学分析流程 1.3.1 胰腺自动分割选用腹部CT原始平扫图像进行影像组学分析。首先以层厚为3 mm的平扫图像作为配准基准,使用3D变形配准方法配准图像[8]。数据增强方法包括平移、重采样、线性插值、白噪声。本研究采用nnU-Net[9]对胰腺整体进行自动分割,预处理流程中,根据整体数据集灰度分布并基于每张图像的灰度中位数进行归一;图像使用紧邻方法进行插值,标签使用线性重采样进行插值,训练图像间距以最低10分位间距为准,训练主干模型网络使用3D U-Net模型,学习模型使用Poly学习率策略,损失函数使用Dice和交叉熵,优化器使用牛顿动量法和随机梯度下降法,批大小为2,训练过程包括1 000个循环,每个循环包含250个小批。随机选择50例患者(25例小胰腺癌患者和25例胰腺正常者)由2位影像医学科主治医师共同在横断面图像对胰腺进行手动勾画,以勾画的结果作为金标准。将模型生成的结果与医师勾画的结果进行比对,计算Dice系数。调整训练模型,使胰腺自动分割模型的Dice系数>0.8。后续所有的胰腺分割均由自动分割模型来完成。
1.3.2 提取并筛选影像组学特征使用Python 3.8软件提取影像组学特征,提取的影像组学特征包括一阶统计量、形状特征、灰阶共生矩阵特征、灰阶依赖矩阵特征、灰阶游程矩阵特征、灰阶大小区域矩阵特征和邻域灰阶差矩阵特征7组,共提取到影像组学特征849个。首先利用方差分析、Spearman相关分析排除组间差异无统计学意义或与胰腺癌无相关性的特征,然后分别绘制单个组学特征的ROC曲线,并以AUC值作为评判标准,选取AUC值排在前8位的组学特征建立分类模型。
1.3.3 预测模型的构建和评估选择极端梯度提升(extreme gradient boosting,XGBoost)分类器,利用R 3.3.3软件的XGBoost包进行XGBoost预测模型的构建。具体参数设置如下:总迭代次数为100,学习率为0.3,叶节点继续分裂所需的最小损失函数下降值为5,每棵树的最大深度为6。XGBoost在损失函数里加入了正则项,在控制模型的复杂度的同时防止模型过拟合。绘制XGBoost预测模型的ROC曲线,计算AUC值以评价预测模型的诊断效能。采用决策曲线分析法(decision curve analysis,DCA)评估预测模型的临床应用性。
1.4 统计学处理应用SPSS 22软件进行统计学分析。呈正态分布的计量资料以x ±s表示,采用t检验进行比较;计数资料以例数和百分数表示,采用χ2检验进行比较。检验水准(α)为0.05。
2 结果 2.1 研究人群的临床和病理特征训练集和验证集中小胰腺癌患者和胰腺正常者的人口学特征见表 1。训练集和验证集中小胰腺癌患者与胰腺正常者的年龄、BMI差异均无统计学意义(P均>0.05);训练集中小胰腺癌患者的男性占比高于胰腺正常者(P=0.03),验证集中小胰腺癌患者和胰腺正常者性别差异无统计学意义(P=0.44)。206例小胰腺癌的最大径为(1.69±0.77)cm,其中训练集小胰腺癌的最大径为(1.61±0.34)cm,验证集小胰腺癌的最大径为(1.81±1.32)cm。依据美国癌症联合委员会(American Joint Committee on Cancer,AJCC)胰腺癌分期第8版[10],206例小胰腺癌中T1b期5例、T1c期201例,N0、N1、N2期分别有122、67和17例,M1期2例;ⅠA期(T1N0M0)121例。
|
|
表 1 训练集和验证集患者的人口学特征 Tab 1 Demographic characteristics of patients in training set and validation set |
2.2 影像组学特征
胰腺自动分割模型的Dice系数为0.81~0.92,平均值为0.85,能很好地完成胰腺自动分割任务。从整体胰腺的3D容积中共提取了849个影像组学特征,组间差异无统计学意义或与胰腺癌无相关性的779个特征首先被排除,剩余70个影像组学特征。以单个影像组学特征的AUC值作为评判标准,选取AUC值排在前8位的组学特征(一阶_偏度、一阶_中位数、一阶_平均值、原始图像_最大值、原始图像_最小值、一阶_四分位数间距、一阶_第90百分位数、一阶_第10百分位数)作为分类模型特征。平均值的误差棒图显示上述8个组学特征在组间均有显著差异(图 1)。
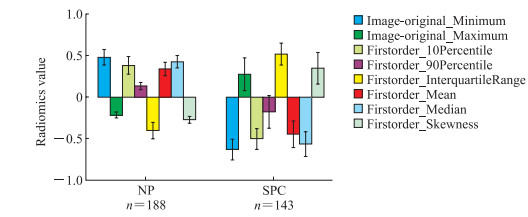
|
图 1 选中的8个影像组学特征平均值的误差棒图 Fig 1 Error-bar chart of seclected 8 radiomics features x ±sx. NP: Normal pancreas; SPC: Small pancreatic cancer. |
2.3 XGBoost预测模型的评价
利用XGBoost分类器构建了一个分类模型,纳入模型的影像组学特征的相对重要性见图 2。XGBoost预测模型的鉴别表现见图 3,XGBoost预测模型在训练集和验证集的AUC值分别为0.99(95% CI 0.97~0.99)和0.99(95% CI 0.96~0.99)。在训练集中XGBoost预测模型诊断小胰腺癌的灵敏度、特异度、准确度、阳性预测值和阴性预测值分别为0.92、0.97、0.95、0.91和0.98,在验证集中其灵敏度、特异度、准确度、阳性预测值和阴性预测值分别为0.94、0.96、0.95、0.93和0.97。XGBoost预测模型鉴别小胰腺癌的典型病例见图 4。

|
图 2 影像组学特征在XGBoost预测模型中的相对重要性 Fig 2 Relative importance of the selected 8 radiomics features in XGBoost prediction model XGBoost: Extreme gradient boosting. |

|
图 3 XGBoost预测模型的受试者操作特征曲线 Fig 3 Receiver operating characteristic curves of XGBoost prediction model XGBoost: Extreme gradient boosting; AUC: Area under curve. |

|
图 4 XGBoost预测模型准确识别出小胰腺癌 Fig 4 XGBoost prediction model accurately identifying small pancreatic cancer Male, 45-year-old. A: Axial non-enhanced computed tomography (CT) image showed that no obvious abnormality was observed in the head of pancreas; B: Axial contrast-enhanced CT arterial phase image demonstrated a hypoattenuation mass located at the pancreatic head; C: The 3D volume of the whole pancreas was automatically segmented from the non-enhanced CT images by nnU-Net; D: According to the XGBoost model, the patient had a 94.72% probability of small pancreatic cancer. XGBoost: Extreme gradient boosting. |
在训练集86例早期(T1N0M0)小胰腺癌中,XGBoost预测模型准确识别出79例,灵敏度为0.92;在验证集35例早期小胰腺癌中,XGBoost预测模型准确识别出33例,灵敏度为0.94。
2.4 XGBoost预测模型的临床适用性DCA分析结果(图 5)表明,无论在验证集还是训练集中,当XGBoost预测模型诊断小胰腺癌的概率>0.02时,使用XGBoost预测模型诊断小胰腺癌比将所有患者视为小胰腺癌患者或将所有患者视为正常胰腺者更能使患者在临床中获益。
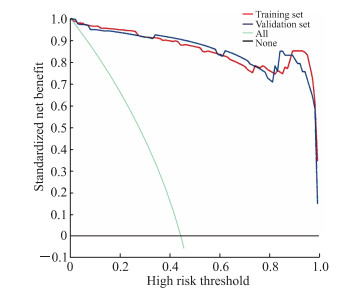
|
图 5 XGBoost预测模型的决策曲线分析 Fig 5 Decision curve analysis for XGBoost prediction model With a threshold probability greater than 0.02, using the XGBoost prediction model to predict small pancreatic cancer is more beneficial than the "treat-all-patients as small pancreatic cancer" scheme or the "treat-all-patients as normal pancreas" scheme in both training set and validation set. XGBoost: Extreme gradient boosting. |
3 讨论
本研究利用nnU-Net模型在腹部CT平扫图像上完成对整体胰腺的自动分割,提取影像组学特征,构建的XGBoost预测模型在训练集和验证集预测小胰腺癌和正常胰腺的AUC值均为0.99,在训练集和验证集预测早期胰腺癌(T1N0M0)的灵敏度分别为0.92和0.94,初步证实基于腹部CT平扫图像的影像组学分析有可能成为小胰腺癌筛查的一个辅助工具,有助于胰腺癌的早期诊断。
影像学检查诊断胰腺癌的灵敏度随肿瘤体积的减小而降低[6]。当前的诊断技术在小胰腺癌的诊断方面存在较大的局限性,且缺乏针对小胰腺癌的新的检测手段,因此有必要改进现有的检测手段以提高小胰腺癌的检出率。虽然腹部CT诊断胰腺癌的灵敏度和特异度不及MRI和超声内镜[3],但因其方便、快捷、经济和可及性高等,仍然被视为初次评估可疑胰腺癌最常用的影像学检查方式。在腹部CT图像上小胰腺癌的灰度与周围正常胰腺组织极为接近,导致小胰腺癌常被漏诊。CT增强虽然有助于小胰癌的检出,但其应用范围不如CT平扫。若能在腹部CT平扫图像上识别出小胰腺癌,势必可提高小胰腺癌的检出率。基于这些原因,本研究利用常规的腹部CT平扫图像、采用影像组学分析方法探索筛查小胰腺癌的策略。
有学者积极探索新的影像学征象和新的技术手段以改善腹部CT在小胰腺癌诊断中的应用价值,如部分胰腺实质萎缩是诊断≤10 mm小胰腺癌的一种新的特异性征象[11],局灶性胰腺实质萎缩是原位癌的重要标志[12],孤立的高级别上皮内瘤变可表现为胰管狭窄[13],“ K征”可作为胰腺癌早期诊断的指标[14],这些征象的发现有助于提高医务工作者对早期胰腺癌影像学征象的认识,可能对发现小胰腺癌、提高胰腺癌早期诊断率有积极意义。但这些研究样本量均较小(20~52例),对于胰腺癌早期诊断的实际价值仍有待通过大样本、前瞻性研究进一步验证。
目前传统的影像学检查在提高胰腺癌早期诊断方面价值有限,需要突破现有的基础。人工智能是一种基于人脑神经结构的强大分析工具,可以识别人眼无法识别的征象,已被用于胰腺肿瘤的鉴别诊断、预后及治疗反应评估[15-18]。Liu等[7]报道基于卷积神经网络的人工智能模型可以在门静脉期CT图像中准确地识别出胰腺癌,准确度接近99%;该模型诊断小胰腺癌的灵敏度在内部测试集中亦高达92.1%,在外部测试集中为63.1%。但是,该研究有两点局限,一是采用人工标注胰腺图像费时、费力,二是标注的是增强图像的胰腺肿瘤区域而不是整体胰腺。因小胰腺癌和周围正常胰腺密度差异小,常常肉眼不可见,无法在平扫CT图像上完成标记,在小胰腺癌筛查方面价值有限。Chu等[19]探索了基于CT的影像组学特征在鉴别胰腺导管腺癌和正常胰腺方面的应用效果,构建的随机森林二元分类模型总体准确度为99.2%。但该研究纳入的胰腺癌平均大小为4.1 cm,普通的放射科医师很容易观察到,不易漏诊。此外,该研究纳入的健康对照受试者比导管腺癌患者年轻,年龄相关的胰腺脂肪化和萎缩可能有助于影像组学特征对这些病例进行分类。与上述研究不同,本研究纳入的胰腺癌最大径均≤2 cm,这部分患者是胰腺癌筛查的难点;采用腹部CT平扫图像,常规医疗易于实现;采用基于卷积神经网络的自动分割模型,省时、省力;对胰腺进行整体分割,不进行肿块标记,更符合胰腺癌筛查的真实世界研究。
影像组学特征和疾病状态之间相关性的生物学基础一直是影像组学研究的关注点之一。有研究尝试探索部分影像组学特征潜在的生物学基础,如与接受免疫治疗的非小细胞肺癌患者的生存率显著相关的灰度共生矩阵(gray level co-occurrence matrix,GLCM)反向差异与碳酸酐酶Ⅸ有关[20];Grossmann等[21]研究发现圆球度和紧密性(sphericity and compactness)可以预测TNF受体相关因子6介导的NF-κB激活;Koay等[22]通过定量分析胰腺癌与实质交界区CT平均值分布的差异,发现没有明显界面的胰腺癌比有明显界面的胰腺癌更易发生转移、生存时间更短,并证实胰腺癌与实质交界区平均CT值分布的差异与肿瘤基质和间质的比例相关。偏度(skewness)是纳入本研究XGBoost预测模型的8个组学参数中权重最大的一个参数,偏度描述的是灰度强度分布的形状,反映了灰度分布的不对称性,理论上讲小胰腺癌的存在会增大胰腺整体的灰度偏度,本研究结果也显示小胰腺癌的灰度偏度大于正常胰腺,但胰腺整体的灰度偏度与小胰腺癌的直接关系仍有待进一步研究。
本研究存在以下不足。一是本研究样本量相对较小。胰腺癌的发病率较低,2018年全球癌症统计结果显示,东亚地区每10万人仅有7人发病[23],而大多数胰腺癌发病时已处于晚期,文献报道仅约25%的早期胰腺癌有症状[24]。然而为了使研究结果稳定、可靠,组学研究往往需要较大的样本量。二是本研究为单中心、回顾性研究。在临床实践中,不同医疗机构的医师会不可避免地遇到源自不同影像设备、扫描技术等所带来的变异,而且回顾性研究所固有的选择性偏倚难以避免,因此本研究结果是否具有广泛代表性尚需外部数据集的验证。三是本研究以整体胰腺作为靶区进行影像组学分析,能准确识别出小胰腺癌患者,但不能预测小胰腺癌在胰腺的具体位置,需要在横断面增强图像或行超声内镜检查以进一步评估病灶位置。
本研究结果初步证实基于腹部CT平扫图像的影像组学分析能准确鉴别小胰腺癌患者和胰腺正常者,可能有助于小胰腺癌的筛查。
| [1] |
SIEGEL R L, MILLER K D, FUCHS H E, JEMAL A. Cancer statistics, 2021[J]. CA Cancer J Clin, 2021, 71: 7-33. DOI:10.3322/caac.21654 |
| [2] |
AGARWAL B, CORREA A M, HO L. Survival in pancreatic carcinoma based on tumor size[J/OL]. Pancreas, 2008, 36: e15-e20. DOI: 10.1097/mpa.0b013e31814de421.
|
| [3] |
KANNO A, MASAMUNE A, HANADA K, MAGUCHI H, SHIMIZU Y, UEKI T, et al. Multicenter study of early pancreatic cancer in Japan[J]. Pancreatology, 2018, 18: 61-67. DOI:10.1016/j.pan.2017.11.007 |
| [4] |
EGAWA S, TOMA H, OHIGASHI H, OKUSAKA T, NAKAO A, HATORI T, et al. Japan pancreatic cancer registry; 30th year anniversary: Japan pancreas society[J]. Pancreas, 2012, 41: 985-992. DOI:10.1097/MPA.0b013e318258055c |
| [5] |
KAWADA N, UEHARA H, KATAYAMA K, TAKAKURA R, IOKA T, TANAKA S, et al. Diagnostic clues and subsequent examinations that detected small pancreatic cancer[J]. Hepatogastroenterology, 2012, 59: 1665-1669. |
| [6] |
DEWITT J, DEVEREAUX B M, LEHMAN G A, SHERMAN S, IMPERIALE T F. Comparison of endoscopic ultrasound and computed tomography for the preoperative evaluation of pancreatic cancer: a systematic review[J]. Clin Gastroenterol Hepatol, 2006, 4: 717-725. DOI:10.1016/j.cgh.2006.02.020 |
| [7] |
LIU K L, WU T H, CHEN P T, TSAI Y M, ROTH H, WU M S, et al. Deep learning to distinguish pancreatic cancer tissue from non-cancerous pancreatic tissue: a retrospective study with cross-racial external validation[J/OL]. Lancet Digit Health, 2020, 2: e303-e313. DOI: 10.1016/S2589-7500(20)30078-9.
|
| [8] |
HEINRICH M P, JENKINSON M, BRADY M, SCHNABEL J A. MRF-based deformable registration and ventilation estimation of lung CT[J]. IEEE Trans Med Imaging, 2013, 32: 1239-1248. DOI:10.1109/TMI.2013.2246577 |
| [9] |
ISENSEE F, JAEGER P F, KOHL S A A, PETERSEN J, MAIER-HEIN K H. nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation[J]. Nat Methods, 2021, 18: 203-211. DOI:10.1038/s41592-020-01008-z |
| [10] |
CHUN Y S, PAWLIK T M, VAUTHEY J N. 8th edition of the AJCC cancer staging manual: pancreas and hepatobiliary cancers[J]. Ann Surg Oncol, 2018, 25: 845-847. DOI:10.1245/s10434-017-6025-x |
| [11] |
YAMAO K, TAKENAKA M, ISHIKAWA R, OKAMOTO A, YAMAZAKI T, NAKAI A, et al. Partial pancreatic parenchymal atrophy is a new specific finding to diagnose small pancreatic cancer (≤ 10 mm) including carcinoma in situ: comparison with localized benign main pancreatic duct stenosis patients[J/OL]. Diagnostics (Basel), 2020, 10: E445. DOI: 10.3390/diagnostics10070445.
|
| [12] |
NAKAHODO J, KIKUYAMA M, NOJIRI S, CHIBA K, YOSHIMOTO K, KAMISAWA T, et al. Focal parenchymal atrophy of pancreas: an important sign of underlying high-grade pancreatic intraepithelial neoplasia without invasive carcinoma, i.e., carcinoma in situ[J]. Pancreatology, 2020, 20: 1689-1697. DOI:10.1016/j.pan.2020.09.020 |
| [13] |
YOKODE M, AKITA M, FUJIKURA K, KIM M J, MORINAGA Y, YOSHIKAWA S, et al. High-grade PanIN presenting with localised stricture of the main pancreatic duct: a clinicopathological and molecular study of 10 cases suggests a clue for the early detection of pancreatic cancer[J]. Histopathology, 2018, 73: 247-258. DOI:10.1111/his.13629 |
| [14] |
KOBASHI Y, UCHIYAMA M, MATSUI J. The "K-sign"-a novel CT finding suggestive before the appearance of pancreatic cancer[J/OL]. Cancers (Basel), 2021, 13: 4222. DOI: 10.3390/cancers13164222.
|
| [15] |
SANDRASEGARAN K, LIN Y N, ASARE-SAWIRI M, TAIYINI T, TANN M. CT texture analysis of pancreatic cancer[J]. Eur Radiol, 2019, 29: 1067-1073. DOI:10.1007/s00330-018-5662-1 |
| [16] |
BIAN Y, GUO S W, JIANG H, GAO S Z, SHAO C W, CAO K, et al. Relationship between radiomics and risk of lymph node metastasis in pancreatic ductal adenocarcinoma[J]. Pancreas, 2019, 48: 1195-1203. DOI:10.1097/MPA.0000000000001404 |
| [17] |
PARK S, CHU L C, HRUBAN R H, VOGELSTEIN B, KINZLER K W, YUILLE A L, et al. Differentiating autoimmune pancreatitis from pancreatic ductal adenocarcinoma with CT radiomics features[J]. Diagn Interv Imaging, 2020, 101: 555-564. DOI:10.1016/j.diii.2020.03.002 |
| [18] |
REN S, ZHAO R, CUI W J, QIU W L, GUO K, CAO Y Y, et al. Computed tomography-based radiomics signature for the preoperative differentiation of pancreatic adenosquamous carcinoma from pancreatic ductal adenocarcinoma[J/OL]. Front Oncol, 2020, 10: 1618. DOI: 10.3389/fonc.2020.01618.
|
| [19] |
CHU L C, PARK S, KAWAMOTO S, FOULADI D F, SHAYESTEH S, ZINREICH E S, et al. Utility of CT radiomics features in differentiation of pancreatic ductal adenocarcinoma from normal pancreatic tissue[J]. AJR Am J Roentgenol, 2019, 213: 349-357. DOI:10.2214/AJR.18.20901 |
| [20] |
CHETAN M R, GLEESON F V. Radiomics in predicting treatment response in non-small-cell lung cancer: current status, challenges and future perspectives[J]. Eur Radiol, 2021, 31: 1049-1058. DOI:10.1007/s00330-020-07141-9 |
| [21] |
GROSSMANN P, STRINGFIELD O, EL-HACHEM N, BUI M M, RIOS VELAZQUEZ E, PARMAR C, et al. Defining the biological basis of radiomic phenotypes in lung cancer[J/OL]. Elife, 2017, 6: e23421. DOI: 10.7554/eLife.23421.
|
| [22] |
KOAY E J, LEE Y, CRISTINI V, LOWENGRUB J S, KANG Y A, LUCAS F A S, et al. A visually apparent and quantifiable CT imaging feature identifies biophysical subtypes of pancreatic ductal adenocarcinoma[J]. Clin Cancer Res, 2018, 24: 5883-5894. DOI:10.1158/1078-0432.CCR-17-3668 |
| [23] |
BRAY F, FERLAY J, SOERJOMATARAM I, SIEGEL R L, TORRE L A, JEMAL A. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68: 394-424. DOI:10.3322/caac.21492 |
| [24] |
KANNO A, MASAMUNE A, HANADA K, KIKUYAMA M, KITANO M. Advances in early detection of pancreatic cancer[J/OL]. Diagnostics (Basel), 2019, 9: E18. DOI: 10.3390/diagnostics9010018.
|
 2022, Vol. 43
2022, Vol. 43


