蛋白质是细胞功能的最终执行者之一,大部分蛋白质与其他分子组成复合体后才能发挥正常的生理功能[1]。研究生物体内蛋白质相互作用的方法目前主要有2种:高通量筛选技术和蛋白质相互作用验证技术。高通量筛选技术包括酵母双杂交技术、免疫共沉淀-质谱联用技术、亲和纯化-质谱联用技术和BioID-质谱联用技术等[2];蛋白质相互作用验证技术包括免疫共沉淀-蛋白质印迹、双分子荧光互补技术、荧光共振能量转移技术等[3]。其中BioID-质谱联用技术是一种新的蛋白质相互作用筛选方法[4]。
BirA是一个分子量为35 000的蛋白质,在大肠杆菌中能够对BirA接受肽序列进行生物素标记[5]。这一过程包括2个步骤:(1)生物素与ATP结合形成生物素-单磷酸腺苷(adenosine monophosphate,AMP);(2)BirA蛋白将生物素从生物素-AMP转移到生物素接受肽标签(biotin acceptor tag,BAT)序列的特定赖氨酸残基上[5]。通过对BirA突变体的筛选,研究者发现BirA R118G突变体(BirA*)对BAT序列的选择性明显减弱,从而使BirA*具备了对几乎所有邻近蛋白进行生物素标记的能力[6]。由于蛋白质生物素化在细胞内较为罕见,并且易于选择性隔离,因此如果构建BirA*与目的蛋白质的融合表达载体并使其在细胞内表达,那么细胞内与目的蛋白质相互作用的蛋白质即能够被BirA*进行生物素标记,从而可以被链霉素磁珠纯化,这一技术被称为BioID技术。
本研究通过建立BioID-质谱联用平台筛选与转录因子Yes相关蛋白1(Yes-associated protein 1,YAP1)相互作用的蛋白质,并采用免疫共沉淀-蛋白质印迹法对部分蛋白质进行验证。
1 材料和方法 1.1 YAP1-BirA*融合表达载体的构建选择本实验室保存的pcDNA3.1作为质粒骨架,构建YAP1-BirA*融合表达载体。首先将pcDNA3.1-BirA*质粒用NheⅠ和EcoRⅠ进行双酶切,获得质粒骨架。通过带有NheⅠ和EcoRⅠ酶切位点的引物PCR扩增YAP1的表达序列,然后再用NheⅠ和EcoRⅠ对纯化的PCR产物进行酶切。通过T4连接酶将YAP1片段与质粒骨架进行连接,连接产物转化大肠杆菌后接种到LB(Luria-Bertani)平板培养基。次日,挑选单克隆菌落,PCR验证后进行Sanger法测序,获得YAP1-BirA*融合表达载体。
1.2 细胞培养293T细胞培养在含10% FBS、1%青链霉素的DMEM培养基中。细胞接种至10 cm培养皿后,用Lipofectamine 2000将pcDNA3.0、pcDNA3.0-BirA*或pcDNA3.0-YAP1-BirA*质粒转染进293T细胞中。转染后给予细胞50 μmol/L生物素处理。24 h后收取细胞总蛋白质进行后续分析。
1.3 生物素化蛋白质的亲和力捕获用PBS洗涤细胞后,室温下加入1 mL裂解液(50 mmol/L Tris、500 mmol/L NaCl、0.4% SDS、5 mmol/L EDTA、1 mmol/L DTT和蛋白酶抑制剂,pH 7.4)裂解细胞。超声处理后,加入Triton X-100至终浓度为2%,再次超声处理后加入等体积预冷的50 mmol/L Tris,再次进行超声处理。12 000×g 4 ℃离心15 min,取上清,与300 μL Dynabead磁珠孵育过夜。用1 mL洗涤缓冲液1(2% SDS)洗2次,每次8 min;用1 mL洗涤缓冲液2(0.1%脱氧胆酸钠、1% Triton X-100、500 mmol/L NaCl、1 mmol/L EDTA和50 mmol/L Hepes,pH 7.5)洗2次,洗涤缓冲液3(250 mmol/L LiCl、0.5% NP-40、0.5% 脱氧胆酸钠、1 mmol/L EDTA和10 mmol/L Tris,pH 8.1)洗1次,洗涤缓冲液4(50 mmol/L Tris、50 mmol/L NaCl,pH 7.4)洗1次。从获得的蛋白质样品中取10%用于蛋白质印迹法检测,余90%用于质谱分析。所有溶液的溶剂均为去离子水。
1.4 蛋白质印迹法检测蛋白质样品经BCA法定量后加入SDS-还原性裂解液,依次进行超声处理、沸水浴煮5 min、冰上静置2 min,用于后续蛋白质印迹法检测。蛋白样品按每孔40 μg上样,140 V电泳1 h,100 V冰上转膜2 h。5%脱脂牛奶封闭2 h后,4 ℃孵育一抗过夜。次日,室温下用TBST洗膜4次,每次5 min。然后室温孵育二抗2 h,用TBST洗膜4次,每次5 min。室温下采用ECL法显影。所用抗体包括YAP1抗体(货号:12395,美国CST公司)、GAPDH抗体(货号:Ab0037,上海泊湾生物科技有限公司)和HRP标记山羊抗兔IgG二抗(货号:Ab0101,上海泊湾生物科技有限公司)。一抗稀释比例均为1∶1 000,二抗为1∶5 000。
1.5 质谱鉴定蛋白质样品中加入磁珠,用胰酶消化过夜,离心去除磁珠。通过高分辨率、高精度液相色谱-串联质谱法(liquid chromatography-tandem mass spectrometry,LC-MS/MS)分析肽的组成,比对人类蛋白质数据库UniProt(www.uniprot.org)进行蛋白质识别。最后对比对结果进行排序、过滤和统计分析。
2 结果 2.1 BioID-质谱联用技术筛选细胞内邻近蛋白质的原理BioID的基本原理和操作流程如图 1所示。首先通过分子生物学方法构建目的蛋白质-BirA*的融合表达载体。将蛋白质-BirA*、BirA*、对照质粒转染293T细胞,同时给予细胞50 μmol/L生物素处理。在生物素存在的情况下,细胞中的融合蛋白质可以对目的蛋白质的邻近蛋白质进行生物素标记。生物素化的蛋白质在细胞裂解后,可以被链霉素磁珠纯化,进一步通过质谱分析,可以鉴定出能与目的蛋白质相互作用的蛋白质。
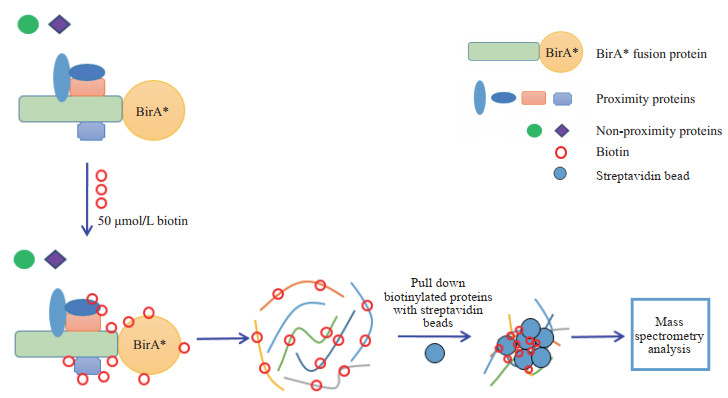
|
图 1 BioID-质谱联用技术筛选目的蛋白质相互作用蛋白质的原理 Fig 1 Mechanism of screening interacting proteins of target protein by BioID combined with mass spectrometry |
2.2 YAP1-BirA*融合表达载体在细胞中对邻近蛋白质进行生物素标记
选择转录因子YAP1作为目的蛋白质进行技术体系验证。首先将YAP1克隆到BirA*的氨基末端,构建YAP1-BirA*融合表达载体。在培养的293T细胞中转染YAP1-BirA*融合表达载体、BirA*表达载体、空载体,同时在培养基中加入50 μmol/L生物素。24 h后裂解细胞,提取蛋白质进行SDS-PAGE;常规转膜后,5%脱脂牛奶封闭2 h,用HRP偶联的链霉亲和素与PVDF膜孵育过夜,洗膜后,ECL显影,检测细胞内的蛋白质是否被生物素化修饰。如图 2所示,在不加生物素处理的细胞中,转染YAP1-BirA*融合表达载体、BirA*表达载体、空载体都不能引起细胞内蛋白质被生物素化修饰。在培养基中添加生物素的细胞中,空载体和BirA*表达载体均不能引起细胞内蛋白质的生物素化修饰,而YAP1-BirA*融合蛋白能够明显引起细胞内蛋白质的生物素化修饰。这一结果提示,细胞内的多种蛋白质可与YAP1-BirA*融合蛋白相互作用,而不与BirA*蛋白质相互作用,表明BioID技术可以用来筛选能与YAP1相互作用的蛋白质。
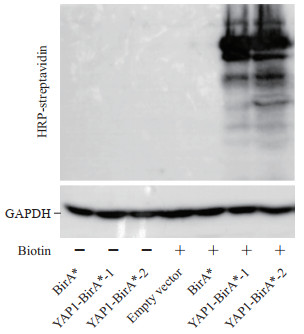
|
图 2 YAP1-BirA*融合蛋白能够对邻近蛋白质进行生物素标记 Fig 2 YAP1-BirA* fusion protein successfully biotin-labeled proximity proteins YAP1: Yes-associated protein 1; HRP: Horseradish peroxidase; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. |
2.3 YAP1-BirA*融合表达载体能够成功表达目的蛋白质
通过蛋白质印迹法检测YAP1-BirA*融合表达载体能否成功表达YAP1-BirA*目的蛋白质。如图 3所示,通过YAP1特异抗体检测发现,无论细胞培养基中是否加入生物素,转染YAP1-BirA*融合表达载体的细胞中均能检测到YAP1-BirA*融合蛋白,而在BirA*表达载体转染的细胞中未检测到YAP1-BirA*融合蛋白。
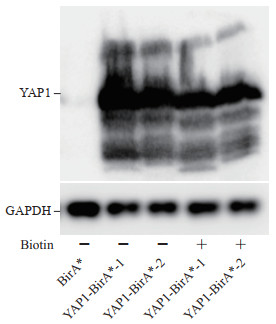
|
图 3 YAP1-BirA*融合表达载体成功表达目的蛋白质 Fig 3 YAP1-BirA* fusion expression plasmid successfully expressed target protein YAP1: Yes-associated protein 1; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. |
2.4 链霉亲和素磁珠纯化生物素标记的蛋白质
细胞裂解后,超声处理,采用链霉亲和素磁珠对细胞内生物素标记的蛋白质进行亲和纯化。磁珠经多次洗涤后,用SDS样本缓冲液处理,PAGE分离蛋白质后采用银染试剂盒对PAGE凝胶中的蛋白质进行显色。如图 4所示,目的蛋白质相互的作用蛋白质被亲和素磁珠纯化。
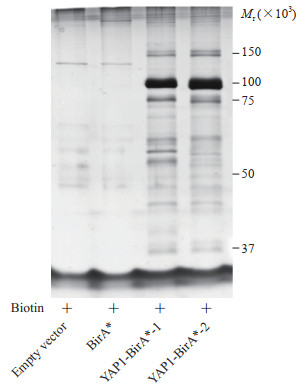
|
图 4 蛋白质银染显示亲和素磁珠能够纯化目的蛋白质的相互作用蛋白质 Fig 4 Protein silver staining showed affinity magnetic beads could purify interacting proteins of target protein YAP1: Yes-associated protein 1. |
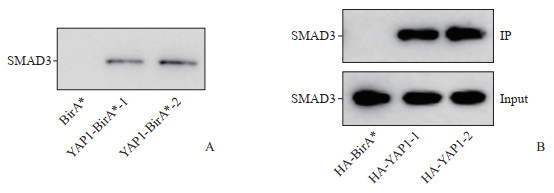
2.5 SMAD家族成员3(SMAD family member 3,SMAD3)与YAP1存在相互作用
对亲和素纯化的蛋白质进行质谱分析发现SMAD3、TEAD(TEA domain transcription factor)1、TEAD3等均可以被检测到。选择SMAD3蛋白进行进一步验证。首先利用SMAD3的特异性抗体,采用蛋白质印迹法检测链霉亲和素磁珠纯化的蛋白质中是否有SMAD3蛋白,结果显示BirA*表达载体表达的细胞样品中链霉亲和素磁珠不能富集SMAD3蛋白,而YAP1-BirA*融合表达载体表达的细胞样品中SMAD3蛋白被链霉亲和素磁珠富集(图 5A)。这一结果表明YAP1-BirA*可以对SMAD3蛋白进行生物素标记,而BirA*不能对SMAD3蛋白进行生物素标记,提示SMAD3蛋白能特异性地与YAP1结合。
|
图 5 SMAD3蛋白能在细胞中与YAP1蛋白相互作用 Fig 5 Intracellular protein-protein interaction between SMAD3 and YAP1 A: SMAD3 can be purified by streptavidin beads in YAP1-BirA* expressing cells; B: Co-IP and Western blotting shows that SMAD3 can be pulled down by YAP1. SMAD3: SMAD family member 3; YAP1: Yes-associated protein 1; IP: Immunoprecipitation. |
进一步采用免疫共沉淀-蛋白质印迹法对YAP1与SMAD3的相互作用进行验证。首先在细胞中过表达HA-BirA*和HA-YAP1,然后通过HA抗体(货号:3724,美国CST公司)进行免疫共沉淀,并通过蛋白质印迹法检测沉淀前(Input)和沉淀后的样品中SMAD3蛋白的含量,结果显示SMAD3蛋白在所有质粒转染的细胞中具有相似的表达水平;在HA抗体沉淀的样品中,在HA-BirA*过表达的细胞中SMAD3蛋白不能被HA抗体富集,然而在HA-YAP1过表达的细胞中SMAD3蛋白可以被HA抗体富集(图 5B)。这一结果表明SMAD3蛋白与YAP1蛋白在细胞中存在相互作用。
3 讨论酵母双杂交系统[7]和免疫共沉淀-质谱技术[8]是蛋白质相互作用研究的2种主要策略,它们各有优缺点。酵母双杂交技术基于cDNA文库进行筛选,可以检测到弱相互作用的蛋白质;它要求蛋白质保持正确折叠的能力,这些相互作用必须发生在酵母内。免疫共沉淀是基于抗原-抗体反应研究蛋白质相互作用的经典方法,常被用于鉴定特定蛋白复合物中未知的蛋白组分;缺点是这2种蛋白质的结合可能不是直接结合,且实验过程比较烦琐,对实验者的操作技术要求较高。在对目的蛋白质的相互作用蛋白质进行高通量筛选中,倾向首先通过BioID-质谱联用技术确定候选分子,然后通过免疫共沉淀-蛋白质印迹法对候选分子进行验证。膜镶嵌蛋白质的相互作用蛋白质筛选研究中不适于用酵母双杂交或免疫共沉淀-质谱技术。BioID可以对膜镶嵌蛋白质周围的蛋白质进行生物素标记,在严格的裂解条件和亲和纯化条件下,将生物素化的蛋白质进行富集,因此应用BioID技术筛选膜镶嵌蛋白质的相互作用蛋白质可能具有明显优势。
本实验验证了BioID技术在筛选核内转录因子的相互作用蛋白质中具有较好的价值。在具体实施过程中,首先需要考虑表达载体的选用,对于一些容易转染的细胞类型,可以选择本实验所用的pcDNA3.1骨架质粒,瞬转后进行BioID研究。对于心肌细胞、内皮细胞、平滑肌细胞等转染难度大的细胞,我们构建了BioID的腺病毒系统,可以进行相互作用蛋白质筛选。其次,需要考虑融合蛋白的正确表达和准确定位,这是整个实验成功的关键步骤。重组质粒需要通过测序验证,保证目的基因与BirA*在同一个阅读框内。在本实验过程中,一方面需要用特异抗体对目的蛋白质在细胞中的表达进行检测,另一方面需要用HRP标记的亲和素对细胞内蛋白质的生物素标记状态进行检测。此外,还需要考虑将目的蛋白质融合在BirA*的氨基末端还是羧基末端,本实验结果表明将YAP1融合在BirA*的氨基末端可以发挥生物素标记的功能。对于一些在细胞中切割后才能发挥功能的蛋白,如跨膜蛋白早老蛋白1(presenilin 1,PSEN1)等,需要根据具体的实验目的、蛋白质在细胞中的定位、蛋白质行使功能时的空间构象选择目的蛋白质的与BirA*进行融合的末端。
在实验操作过程中,需要对每个步骤进行质控。在本实验中,我们通过HRP偶联的链霉亲和素进行细胞内生物素标记蛋白质的检测时发现,在不加外源性生物素的情况下,YAP1-BirA*融合蛋白不能对细胞内蛋白质进行生物素标记;在生物素存在的情况下,只有YAP1-BirA*融合蛋白可以对细胞内的蛋白质进行生物素标记;表明这一方法具有很高的特异性。在利用BioID系统筛选细胞中目的蛋白质的相互作用蛋白质时,应设立阴性对照,以检测整个实验体系的特异性。对于阴性对照的选择,我们对亲和纯化后的蛋白质进行银染时发现空载体和BirA*表达载体的亲和纯化样品很少有蛋白被纯化富集,表明单一BirA*蛋白在细胞内作用的底物较少,特异性较高;然而我们仍然可以看到细胞内存在一些生物素化蛋白质,这可能导致实验结果出现假阳性。因此质谱鉴定的阴性对照最好选择BirA*样品进行比较分析。在这一技术体系的建立过程中,我们比较了免疫共沉淀样品与BioID亲和纯化样品的银染结果,BioID亲和纯化的方法具有非常低的假阳性率。这与生物素和亲和素的强作用力有关,在BioID亲和纯化过程中,我们通过不同的洗涤液洗涤纯化样品9次,去除了一些非特异信号。
蛋白质相互作用的BioID-质谱筛选技术有一定的局限性,包括如下几个方面:(1)BioID系统依赖于细胞培养,一些组织样本可能无法被用作研究对象。(2)BioID系统依赖于BirA*-目的蛋白质的正确表达和定位,一些分子量较小的目的蛋白质的定位和功能属性可能会对BirA*造成影响。我们在构建YAP1-BirA*融合表达载体时,采用荧光素酶报告基因法检测了YAP1-BirA*融合蛋白,对YAP1下游靶基因结缔组织生长因子启动子的转录活性进行了验证。(3)BioID系统可能会出现假阴性。BirA*主要是对邻近蛋白质的赖氨酸进行生物素化[6],其作用强弱一方面取决于BirA*融合蛋白对邻近蛋白质的生物素标记能力,另一方面取决于邻近蛋白质中可被生物素化的赖氨酸的数量和可用性,因此邻近蛋白质生物素标记的程度仅能在一定水平上反映2种蛋白质相互作用的关联强弱。同样,没有被生物素标记的蛋白质也不能排除它们与目的蛋白质可能存在的相互作用。鉴于BioID的分子机制,邻近蛋白质生物素标记技术不能替代免疫共沉淀用于验证蛋白质的相互作用,但可以作为一种策略对目的蛋白质的相互作用蛋白质进行筛选,具有很高的实用价值。
| [1] |
SHIN W H, KUMAZAWA K, IMAI K, HIROKAWA T, KIHARA D. Current challenges and opportunities in designing protein-protein interaction targeted drugs[J]. Adv Appl Bioinform Chem, 2020, 13: 11-25. |
| [2] |
IACOBUCCI I, MONACO V, COZZOLINO F, MONTI M. From classical to new generation approaches: an excursus of -omics methods for investigation of protein-protein interaction networks[J/OL]. J Proteom, 2021, 230: 103990. DOI: 10.1016/j.jprot.2020.103990.
|
| [3] |
ENGLAND C G, EHLERDING E B, CAI W B. NanoLuc: a small luciferase is brightening up the field of bioluminescence[J]. Bioconjug Chem, 2016, 27: 1175-1187. DOI:10.1021/acs.bioconjchem.6b00112 |
| [4] |
XU Y F, FAN X Q, HU Y. In vivo interactome profiling by enzyme-catalyzed proximity labeling[J/OL]. Cell Biosci, 2021, 11: 27. DOI: 10.1186/s13578-021-00542-3.
|
| [5] |
BECKETT D, KOVALEVA E, SCHATZ P J. A minimal peptide substrate in biotin holoenzyme synthetase-catalyzed biotinylation[J]. Protein Sci, 1999, 8: 921-929. |
| [6] |
ROUX K J, KIM D I, RAIDA M, BURKE B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells[J]. J Cell Biol, 2012, 196: 801-810. DOI:10.1083/jcb.201112098 |
| [7] |
PAIANO A, MARGIOTTA A, DE LUCA M, BUCCI C. Yeast two-hybrid assay to identify interacting proteins[J/OL]. Curr Protoc Protein Sci, 2019, 95: e70. DOI: 10.1002/cpps.70.
|
| [8] |
TANG Z Y, TAKAHASHI Y. Analysis of protein-protein interaction by Co-IP in human cells[J]. Methods Mol Biol, 2018, 1794: 289-296. |
 2021, Vol. 42
2021, Vol. 42


