2. 海军军医大学(第二军医大学)东方肝胆外科医院外科, 上海 200438;
3. 海军军医大学(第二军医大学)长海医院消化科, 上海 200433;
4. 海军军医大学(第二军医大学)东方肝胆外科医院病理科, 上海 200438
2. Department of Surgery, Eastern Hepatobiliary Surgery Hospital, Naval Medical University (Second Military Medical University), Shanghai 200438, China;
3. Department of Gastroenterology, Changhai Hospital, Naval Medical University (Second Military Medical University), Shanghai 200433, China;
4. Department of Pathology, Eastern Hepatobiliary Surgery Hospital, Naval Medical University (Second Military Medical University), Shanghai 200438, China
病理诊断是肿瘤诊断的金标准,是临床医师为恶性肿瘤患者制订治疗方案和预测预后的决定性因素。然而,一名合格的病理医师需要长时间的训练,通过阅读数百万张病理切片来积累经验。如果病理医师没有阅读足够的切片,就无法持续提高病理诊断能力。即使经验丰富的高年资病理医师,在每个工作日评估数百或数千张病理切片的巨大压力下,由于疲劳和重复性劳动,也难免出现漏诊或误诊。因此,发展计算病理学,为病理诊断建立标准化程序十分必要。
利用人工智能(artificial intelligence, AI)技术建立的专家医疗系统有助于降低人工相关成本,提高医疗效率[1-2]。基于深度学习的AI算法已开始用于医疗各个环节,包括恶性肿瘤的分类、筛查、风险分层和医疗决策等[3]。AI在乳腺癌前哨淋巴结诊断中的应用开创了AI在病理领域的先河[4]。Campanella等[5]建立了一个无须标注肿瘤细胞即能识别病理切片中肿瘤细胞的深度学习模型,在前列腺癌、基底细胞癌和淋巴结转移乳腺癌诊断中的AUC分别达到0.991、0.989、0.965。在另一项研究中,利用AI算法模型自动评估71例乳腺癌切除样本的人类表皮生长因子受体2(human epidermal growth factor receptor 2, HER-2)表达状态,评估结果与病理学家判断结果的一致性为83%;不一致的原因是HER-2染色的异质性高,通过对12例不一致的病例重新评估后,修正了8例诊断[6]。上述研究结果表明病理AI具有广阔的应用前景。
肝门部胆管癌(hilar cholangiocarcinoma, HC)是一种罕见的肿瘤类型,具有独特的病理特征,即广泛的纤维结缔组织增生和周围神经侵犯(perineural invasion, PNI)[7]。PNI是胆管癌和胰腺癌等多种肿瘤的特殊转移方式,也是预测肿瘤复发和患者预后的重要决定因素[8]。HC独特的病理特征使其成为AI深度学习的理想模型。有研究利用深度学习算法区分H-E染色全切片图像上原发性肝癌的2种亚型——肝细胞癌和胆管癌,准确度超过80%[9]。本研究建立了一种AI算法,其除了能够识别肿瘤细胞外,还能识别神经组织和侵入神经组织的肿瘤细胞。
1 材料和方法 1.1 组织标本来源收集2004-2008年在海军军医大学(第二军医大学)东方肝胆外科医院肝胆外科就诊的HC患者的组织样本。取90例手术切除和10例HC肝脏转移活检样本图像(每个病例的组织切片取5张大小为2 720像素×1 536像素的图片,共计500张)及100例来自慢性胆管炎或慢性胆囊炎的非癌变胆管组织图像(每个病例取1张大小为2 720像素×1 536像素的图片,共计100张)作为训练集。另取40例切除HC标本的250张图像和5例非癌胆管组织切片的50张图像作为测试集(共300张)。比较数据集含有100张图像,包括75例HC组织标本、23例正常胆管组织标本和2例高级别上皮内瘤变组织标本的图像。
1.2 数据标注标注工具为自主研发的标注软件Horizope 1.0,该软件使用Python语言的Qt开发,支持矩形、多边形、圆、曲线、直线和点的标记,标注结果导出格式为JSON。采用曲线标记,将腺体或癌栓的外边缘标记为蓝色,腺体的内边缘标记为绿色,神经标记为红色,分泌物标记为橙红色。正常组织中,胆管上皮标记为砖红色。训练集中的每张图像由2名经验丰富的病理科主治医师独立描述和标注,出现分歧时与1名病理科副主任医师讨论决定。如仍有混淆,用免疫组织化学细胞角蛋白19(cytokeratin 19, CK19)染色检测可疑肿瘤细胞。
1.3 深度学习本研究对每个32×32块(block)是否可能属于不同类型的细胞给出推论。选择32×32块基于以下原因:(1)32×32块足够精确,可以帮助医师在分辨率为50 000像素×30 000像素的图片中定位所有的肿瘤细胞;(2)由于训练集数据的稀疏性和不准确性,很难训练出能够对小于32×32块的区域进行准确判断的算法。此外,训练图片还需要大量的劳动力对其中的小区域进行逐像素标记。鉴于肿瘤形状的多样性,训练的网络不用于预测肿瘤细胞在32×32块内的位置,而是判断该块是否属于某种分类。这似乎很粗糙,但从整体来看,它的准确性足以帮助医师做出快速判断。我们搭建了一种结合GoogLeNet和DenseNet的神经网络模型用于特征提取。整个图片用N×M矩阵(由32×32块生成)表示,有1 024个通道。训练通道表达以32×32块为中心的局部特征和周围块特征,这是通过神经网络自适应学习得到的。训练图片中的肿瘤和其他特征由NxW工具用多边形标记出来,多边形的标记将尽可能准确,多边形内的区域不包含任何其他分类。根据块与多边形的重叠面积计算块的精度。为了对HC细胞和PNI进行深度学习,设计了将不同参数的GoogLeNet和DenseNet相结合的森林块。将分离卷积、重叠池化、跨层连接、局部层归一化、矫正线性单元整合到该森林块中。为了减少过拟合的影响,在每个森林块和全连接层中使用Dropout方法。根据既往经验,在训练中使用了性能优于ADAM、SGD等优化器的RMSProp优化器,并且将网络初始学习率设置为0.01,衰减率设置为0.9。对分辨率为50 000像素×30 000像素的图像进行数据增强,首先从中随机提取大小为1 720像素×1 536像素的图像块,然后对提取的图像块进行水平反射、随机旋转和扭曲后再输入到训练网络中。本研究使用的操作系统为Ubuntu 14.04 64bit,所有代码均是在Tensoflow(Google深度学习框架)中运行,且配置深度学习库CUDA6.5以提升运算速度。
1.4 比较测试专家组选择了一组来自75例HC组织、23例正常胆管组织和2例高级别上皮内瘤变组织标本的病理图像(共100张),以比较AI算法在真实世界的表现及与病理科医师诊断结果的差异。75例HC有2层分类(容易识别或难以识别)。每个病例都需要选择诊断(癌症与非癌症)、神经侵犯(有或没有)及肿瘤细胞在整个图像中的百分比。3名不同工作经验的病理科医师(副主任医师、主治医师和住院医师各1名)在没有任何额外信息的情况下,被要求独立评估同一队列。副主任医师的资历要求为在病理科有超过15年的工作经验,主治医师有5年以上的病理诊断经验,住院医师需要完成临床和病理学专业的培训。这些病例的诊断金标准由1名病理科主任医师和病理科出具的报告确定。
2 结果 2.1 癌细胞检测框架和精确标记AI深度学习网络检测HC细胞的流程如图 1所示。首先对训练集进行分类并精确标注(图 1A)。然后,使用深度卷积神经网络进行训练和分类。在建立AI算法时,利用测试集验证AI算法的效率和准确性。对于未被AI算法识别成功的肿瘤细胞,重新标注并进行深度学习(图 1B)。异质性是人类实体瘤最明显的特征。即使在同一幅图像中,也存在多种类型的肿瘤细胞,包括分化良好的肿瘤细胞、分化不良的肿瘤细胞及分散侵袭的单个肿瘤细胞。那些形成导管的肿瘤细胞很容易被AI算法标记和识别(图 2A)。然而,分散侵袭的肿瘤细胞很难从H-E染色切片图像中被正确识别和标记,易被肉眼忽略。为了避免遗漏,采用免疫组织化学方法在不能100%识别肿瘤细胞的病例中帮助显示这些细胞(图 2B和2C)。如此在对整个图像进行标记时将不会遗漏任何肿瘤细胞。
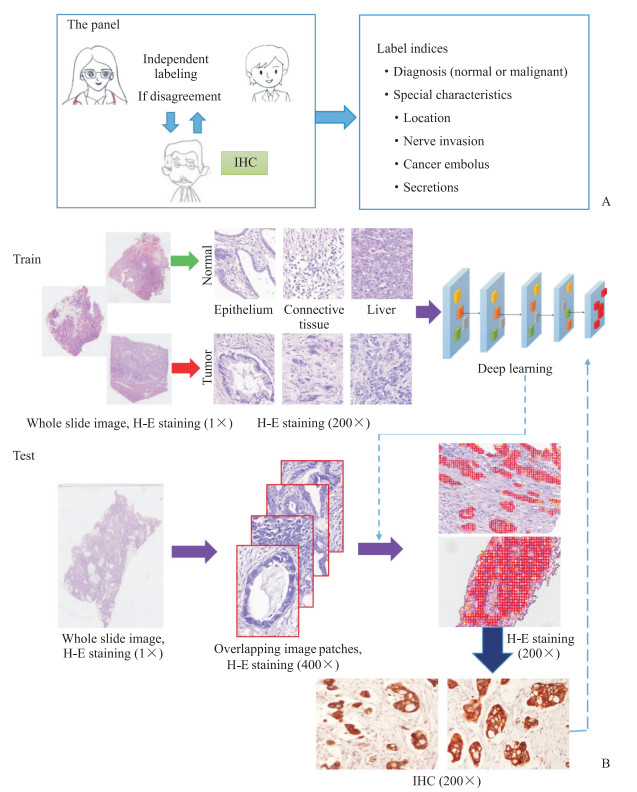
|
图 1 AI深度学习网络检测肿瘤细胞的流程 Fig 1 Process of AI deep learning network detecting tumor cells A: Labeling team and labeling content; B: Deep learning process. AI: Artificial intelligence; IHC: Immunohistochemistry; H-E: Hematoxylin-eosin. |
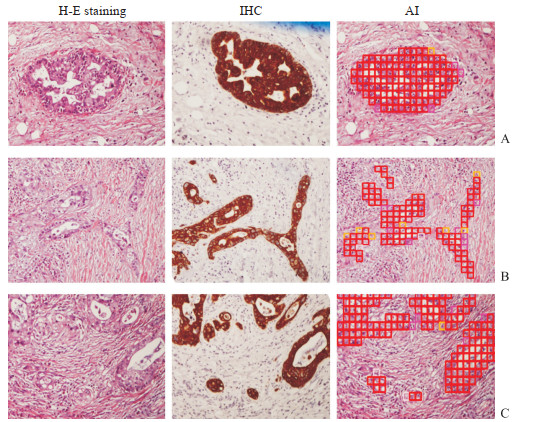
|
图 2 IHC染色助力AI识别病理切片中的低分化肿瘤细胞(400×) Fig 2 IHC staining helps AI to identify poorly differentiated tumor cells in pathological slides (400×) A: Well differentiated bile duct carcinoma cells; B, C: Poorly differentiated tumor cells. IHC: Immunohistochemistry; AI: Artificial intelligence; H-E: Hematoxylin-eosin. |
2.2 AI算法模型
AI模型鉴别肿瘤细胞和非肿瘤细胞的准确度为92.7%(765/825),识别神经病变的准确度为98.3%(177/180)。在训练集中,灵敏度和特异度分别为0.87和0.99。在测试集中,灵敏度为0.87,特异度为0.98,算法模型中假阳性率与漏报率之和低于10%。在HC组织标本切片中,分化良好的肿瘤细胞检测灵敏度极高(0.96),而分化较低的肿瘤细胞检出率稍有下降(0.79)。
2.3 PNI的检测结果PNI是HC转移的独立影响因素,HC细胞首先聚集在神经组织周围,逐渐侵入内膜,然后进入神经纤维,最后随着神经纤维转移。本研究对病理切片中所有接受AI学习的神经进行了标记。在测试集中,评估了AI算法是否能够识别PNI,并学习了肿瘤细胞在神经组织内或周围的位置。如图 3A~3C所示,AI算法很容易识别出HC组织标本H-E染色图像中所有的神经组织,并成功显示了肿瘤细胞与神经之间的空间分布模式。在大切片中,AI算法展示了所有HC细胞侵犯神经的部位(图 3D)。
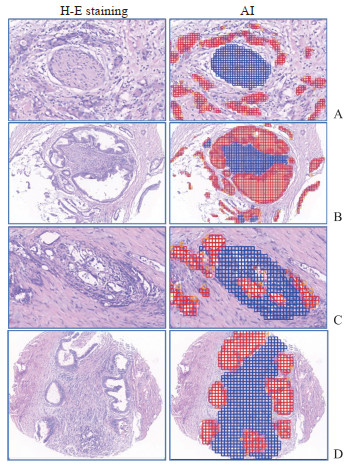
|
图 3 AI算法识别HC组织H-E染色病理切片中的PNI Fig 3 PNI in H-E staining pathological sections of HC tissues recognized by AI algorithm A-C: AI algorithm identifying the neural tissues in HC tissues (200×); D: AI algorithm showing the site of HC cell invasion in the large section (40×). Blue indicates PNI, and red indicates tumor cells. AI: Artificial intelligence; HC: Hilar cholangiocarcinoma; H-E: Hematoxylin-eosin; PNI: Perineural invasion. |
2.4 AI算法与病理医师诊断效能比较
由AI算法模型和3名具有不同工作经验的病理科医师独立评估相同的75例肿瘤组织、23例正常胆管组织和2例高级别上皮内瘤变组织标本的H-E染色病理切片图像(图 4A),比较AI算法与病理医师的诊断效能。AI算法成功检出91.3%(21/23)的正常胆管组织和98.7%(74/75)的肿瘤,2例高级别上皮内瘤变误诊为肿瘤(图 4B)。图 4C展示了具有代表性的38例肿瘤组织和2例高级别上皮内瘤变组织的诊断结果,其余60例的AI算法和3名病理医师诊断结果较一致。由图 4C可见,在被误诊的正常胆管组织中,只有少数细胞被AI算法错误识别为肿瘤细胞。在肿瘤组织中,与金标准相比,副主任医师的诊断结果准确度较高,仅有2例判断为疑似,PNI的诊断准确度达100%;主治医师有2例判断错误,7例为疑似,在识别PNI方面有2例疑似;而住院医师有5例判断错误,在识别PNI方面有1例判断错误、2例疑似。疑似病例均可通过审核整张病理切片和结合免疫组织化学染色结果明确诊断。对于图像中肿瘤细胞百分比,与病理科医师相比,AI算法识别准确度较高,只有2例肿瘤标本图像中AI算法所得结果与金标准差异超过10%,而副主任医师、主治医师和住院医师分别有10例、20例和36例识别结果与金标准差异超过10%。表明AI算法诊断肿瘤的能力可与经验丰富的病理科医师媲美,且在评估肿瘤细胞百分比方面更胜一筹。
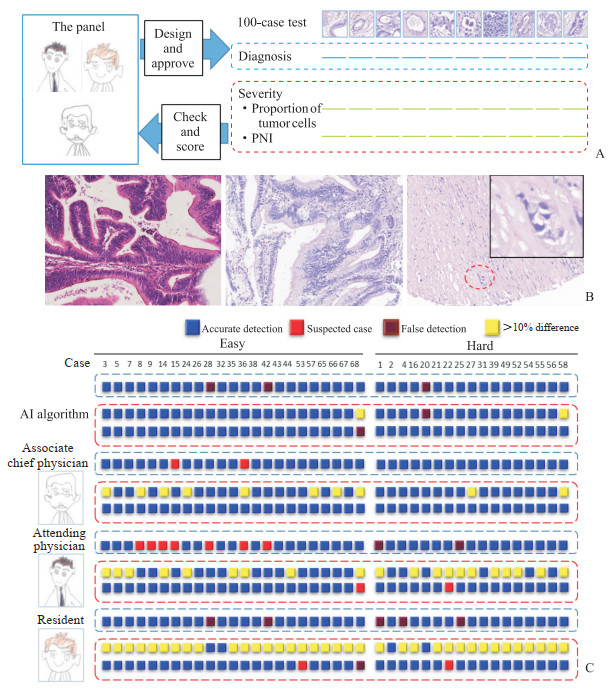
|
图 4 AI算法和病理医师诊断水平比较 Fig 4 Comparison of diagnostic accuracy between AI algorithms and pathologists A: Three pathologists selected 100 pathological pictures (H-E staining, 200×) to evaluate the use of AI algorithm in the real world. B: H-E staining images (40×) of 2 cases (No. 28 and 42) of high-grade intraepithelial neoplasia tissue samples and 1 case (No. 20) of carcinoma tissue sample which were inaccurately detected by AI algorithm. The image in the black box (400×) is an enlarged area of the red circle. C: The ability of pathologists and AI algorithm to identify whether it was a tumor and the proportion of tumor cells or PNI. The cases in the blue imaginary box were the diagnostic accuracy of tumor, and those in the red imaginary box were the proportion of tumor cells and whether there was PNI. AI: Artificial intelligence; H-E: Hematoxylin-eosin. PNI: Perineural invasion. |
3 讨论
靶向治疗和免疫疗法等新的治疗方法的决策都需要以更准确和更翔实的病理诊断结果为基础,病理已不仅仅用于疾病诊断,更是选择治疗策略和判断患者预后的重要依据[10]。准确诊断和评估每例患者整张病理切片中的信息有助于诊断和促进临床决策。AI算法和针对大数据集的深度学习是提高病理诊断的再现性、准确性和效率的一个很好的选择。
本研究使用AI算法进行HC评估取得了显著效果,并积累了大量经验。第一,HC细胞的表现非常复杂,建立肿瘤细胞的标准标注流程将避免漏检。第二,医师对疑难病例的解释有很大差异,这需要一个专家小组和新技术来统一诊断。第三,AI的最佳使用可能不局限于最终诊断,还可在制订临床治疗策略时充当助手。AI在病理领域中的应用具有广阔的前景,但是癌细胞的异质性和标注的困难限制了模型的精确性。在本研究中,由于没有学习到高级别上皮内瘤变及难以标注所有低分化的肿瘤细胞,2例高级别上皮内瘤变被AI诊断为恶性肿瘤,而1张只有7个肿瘤细胞的病理切片被漏诊。有研究用三分类法(阳性癌或怀疑癌、警惕腺瘤或怀疑有肿瘤病变、肿瘤病变阴性)判断活检样本,总体符合率为55.6%,阴性标本符合率为90.6%,阳性标本符合率不足50%[11]。因此,肿瘤细胞的多样性是AI深度学习的瓶颈,必须谨慎处理。免疫组织化学染色等特殊染色方法有助于显示所有的肿瘤细胞。此外,应由病理科医师和肿瘤科医师组成专家组,积极解释原始数据。我们需要建立一个AI平台以尽可能多地收集HC组织病理数据,该平台最终将覆盖所有类型的HC,并不断增强AI算法的能力。AI、医师和实验室之间的合作必将提高从复杂的病例中检出HC细胞的能力。
肿瘤间质在肿瘤侵袭、转移中具有重要作用,病理标本中肿瘤间质比与肿瘤的恶性生物学行为和不良预后密切相关[12]。研究显示低肿瘤间质比与临床进展、浸润深度和淋巴结转移显著相关,肿瘤组织中间质比例高的肿瘤患者预后更差;肿瘤间质比可以通过H-E染色进行评估,与分子标记相比,其测定方法简单、快速,不需要额外的特殊技术和额外费用[12-13]。然而,基于病理形态学的肿瘤间质比定量方法较为主观,准确性因病理医师的经验会有偏差。而计算病理学则能够精确的识别和计算出肿瘤间质比,使其真正成为一种方便、实用的临床工具。本研究也发现AI算法在评估肿瘤细胞百分比方面优于病理科副主任医师。由于目前肿瘤间质比尚没有被列入指南或达成共识意见,还需要多中心临床试验明确其临床和病理实际意义。
PNI是HC的另外一个重要的病理特征,与肿瘤患者的预后密切相关[14]。识别PNI可以预测HC患者的预后,甚至有助于做出最佳的临床决策[15]。神经组织的结构非常典型,深度学习易于识别外周神经,从而判断肿瘤细胞和神经的空间结构关系。本研究发现AI算法能够准确识别PNI。根据肿瘤细胞侵犯外周神经的部位,将之分为肿瘤细胞包绕神经型、肿瘤细胞侵犯神经包膜型、肿瘤细胞侵犯神经纤维型和混合型,这种分型既能反映肿瘤组织中是否存在神经侵犯,又能够给出侵犯的严重程度。
在一些临床场景中,AI算法具有较高的准确度,如评估肿瘤细胞的数量和免疫组织化学染色细胞所占的比例。然而,要将AI应用于临床病理诊断还有很长的路要走。在研发胆管癌AI算法诊断的过程中,我们发现以下问题:(1)染色质量参差不齐导致AI诊断出错。(2)“疑癌”“疑腺瘤”“疑肿瘤病变”难以区分,导致误诊;尤其低分化或未分化的肿瘤细胞难以发现,导致漏诊。为了克服这些缺点,需要为AI算法的深度学习提供大量的数据,还要采用多中心数据进行验证,以分析其在临床中的应用前景。相信病理AI在未来将给患者和医务工作者带来曙光。
| [1] |
BERA K, SCHALPER K A, RIMM D L, VELCHETI V, MADABHUSHI A. Artificial intelligence in digital pathology-new tools for diagnosis and precision oncology[J]. Nat Rev Clin Oncol, 2019, 16: 703-715. DOI:10.1038/s41571-019-0252-y |
| [2] |
NIAZI M K K, PARWANI A V, GURCAN M N. Digital pathology and artificial intelligence[J/OL]. Lancet Oncol, 2019, 20: e253-e261. DOI: 10.1016/S1470-2045(19)30154-8.
|
| [3] |
于观贞, 魏培莲, 陈颖, 朱明华. 人工智能在肿瘤病理诊断和评估中的应用与思考[J]. 第二军医大学学报, 2017, 38: 1349-1354. |
| [4] |
EHTESHAMI BEJNORDI B, VETA M, JOHANNES VAN DIEST P, VAN GINNEKEN B, KARSSEMEIJER N, LITJENS G, et al. Diagnostic assessment of deep learning algorithms for detection of lymph node metastases in women with breast cancer[J]. JAMA, 2017, 318: 2199-2210. DOI:10.1001/jama.2017.14585 |
| [5] |
CAMPANELLA G, HANNA M G, GENESLAW L, MIRAFLOR A, WERNECK KRAUSS SILVA V, BUSAM K J, et al. Clinical-grade computational pathology using weakly supervised deep learning on whole slide images[J]. Nat Med, 2019, 25: 1301-1309. DOI:10.1038/s41591-019-0508-1 |
| [6] |
VANDENBERGHE M E, SCOTT M L, SCORER P W, SÖDERBERG M, BALCERZAK D, BARKER C. Relevance of deep learning to facilitate the diagnosis of HER2 status in breast cancer[J/OL]. Sci Rep, 2017, 7: 45938. DOI: 10.1038/srep45938.
|
| [7] |
BLECHACZ B. Cholangiocarcinoma: current knowledge and new developments[J]. Gut Liver, 2017, 11: 13-26. DOI:10.5009/gnl15568 |
| [8] |
LI C G, ZHOU Z P, TAN X L, ZHAO Z M. Perineural invasion of hilar cholangiocarcinoma in Chinese population: one center's experience[J]. World J Gastrointest Oncol, 2020, 12: 457-466. DOI:10.4251/wjgo.v12.i4.457 |
| [9] |
KIANI A, UYUMAZTURK B, RAJPURKAR P, WANG A, GAO R, JONES E, et al. Impact of a deep learning assistant on the histopathologic classification of liver cancer[J/OL]. NPJ Digit Med, 2020, 3: 23. DOI: 10.1038/s41746-020-0232-8.
|
| [10] |
VOLYNSKAYA Z, CHOW H, EVANS A, WOLFF A, LAGMAY-TRAYA C, ASA S L. Integrated pathology informatics enables high-quality personalized and precision medicine: digital pathology and beyond[J]. Arch Pathol Lab Med, 2018, 142: 369-382. DOI:10.5858/arpa.2017-0139-OA |
| [11] |
YOSHIDA H, SHIMAZU T, KIYUNA T, MARUGAME A, YAMASHITA Y, COSATTO E, et al. Automated histological classification of whole-slide images of gastric biopsy specimens[J]. Gastric Cancer, 2018, 21: 249-257. DOI:10.1007/s10120-017-0731-8 |
| [12] |
WU J, LIANG C, CHEN M, SU W. Association between tumor-stroma ratio and prognosis in solid tumor patients: a systematic review and meta-analysis[J]. Oncotarget, 2016, 7: 68954-68965. DOI:10.18632/oncotarget.12135 |
| [13] |
RANI P, GUPTA A J, MEHROL C, SINGH M, KHURANA N, PASSEY J C. Clinicopathological correlation of tumor-stroma ratio and inflammatory cell infiltrate with tumor grade and lymph node metastasis in squamous cell carcinoma of buccal mucosa and tongue in 41 cases with review of literature[J]. J Cancer Res Ther, 2020, 16: 445-451. DOI:10.4103/0973-1482.193113 |
| [14] |
CHEN S H, ZHANG B Y, ZHOU B, ZHU C Z, SUN L Q, FENG Y J. Perineural invasion of cancer: a complex crosstalk between cells and molecules in the perineural niche[J]. Am J Cancer Res, 2019, 9: 1-21. |
| [15] |
SALOMAN J L, ALBERS K M, RHIM A D, DAVIS B M. Can stopping nerves, stop cancer?[J]. Trends Neurosci, 2016, 39: 880-889. DOI:10.1016/j.tins.2016.10.002 |
 2021, Vol. 42
2021, Vol. 42


