2018年全球数据显示,每年有近57万宫颈癌新发病例,约31.1万例宫颈癌患者死亡[1]。尽管宫颈癌早期患者的治愈率不断提高,但转移或复发患者的预后仍然较差[2]。国际妇产科联合会(International Federation of Gynecology and Obstetrics)分期Ⅰb~Ⅱa期宫颈癌患者的复发风险为10%~20%,局部晚期(Ⅱb~Ⅳa期)宫颈癌患者的复发风险达30%~70%[3-4],且晚期宫颈癌患者缺少有效的治疗选择。因此,深入研究宫颈癌进展的确切分子机制对于该病的诊断和治疗策略很重要。
随着高通量测序和微阵列技术的快速发展,生物信息学技术为研究疾病分子机制及寻找生物标志物提供了新的手段,并进一步促进了疾病分子诊断、靶向及个体化治疗、预后预测等的发展。宫颈癌是一种分子基础具有显著差异的异质性疾病。目前宫颈癌的诊断和预后指标(包括肿瘤大小、浸润深度、淋巴结转移和病理分型)已显示出局限性。传统的分类方式并不能准确地提示宫颈癌预后,甚至可能会延误患者治疗。寻找潜在分子生物标志物的预测模式有可能会改善这种情况。本研究通过基因表达汇编(Gene Expression Omnibus,GEO)数据库和癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库筛选出与宫颈癌相关的关键基因微小染色体维持蛋白(mini-chromosome maintenance protein,MCM)2,并在多种肿瘤中对MCM家族基因进行深入分析,探讨其表达特征和生物学功能。
1 材料和方法 1.1 数据获取与预处理在GEO数据库搜索关键词“ cervical cancer ”,下载了4个基因芯片数据集(GSE6791、GSE39001、GSE55940、GSE63678)。GSE6791包含8个正常样本和20个宫颈癌样本,GSE39001包含17个正常样本和62个宫颈癌样本,GSE55940包含5个正常样本和5个宫颈癌样本,GSE63678包含5个正常样本和5个宫颈癌样本。为了增加样本量,本研究合并了4个微阵列芯片的数据。因为仪器型号、实验人员的技术水平及试剂等不同可能会导致实验结果的批次差异,使用R 4.0.3软件“ SVA”工具包对芯片数据进行了批次校正。
1.2 表达差异分析利用R 4.0.3软件“ limma ”工具包比较肿瘤组织和正常组织的RNA表达差异。以校正P值[错误发现率(false discovery rate,FDR)P]<0.05且|log2倍数差异(fold change,FC)|>1为差异表达基因的筛选标准。
1.3 功能富集分析利用R 4.0.3软件的“ clusterProfiler ”工具包进行基因本体(gene ontology,GO)、京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路富集分析(以P<0.05且FDR P<0.05为条件)。以条形图和点图分别显示GO和KEGG分析的前10种途径。
1.4 蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络建设利用在线数据库STRING(https://string-db.org/)分析差异表达基因的PPI网络,设置最低关联度为0.700,并隐藏网络中未连接的节点。将分析结果导入Cytoscape软件进行可视化,并选择节点前10位的差异表达基因作为关键基因进行生存分析。
1.5 生存分析通过R 4.0.3软件“ survival ”工具包对关键基因进行Kaplan-Meier生存分析和对MCM家族基因进行单因素Cox回归分析(以P<0.05为条件)。所有泛癌数据均从TCGA数据库下载。
1.6 表达验证与功能分析利用GEPIA2(http://gepia2.cancer-pku.cn)对MCM2基因进行表达差异分析,其数据基于TCGA和基因型-组织表达(Genotype-Tissue Expression,GTEx)数据库。利用人类蛋白质图谱(the Human Protein Atlas,HPA)网站(https://www.proteinatlas.org/)比较正常组织和宫颈癌组织的MCM2蛋白表达差异,明确MCM2蛋白在细胞内的定位。利用TCGA数据对MCM2进行基因功能集富集分析,以P<0.05且FDR P<0.05为条件筛选出显著富集的通路。利用TCGA数据库选取140条MCM2的KEGG富集的信号通路进行MCM2的基因功能集富集分析。
1.7 基因相关性分析与免疫亚型分析利用R 4.0.3软件的“ corrplot ”工具包对MCM家族基因进行Pearson相关性分析。基于TCGA数据,利用“ limma ”工具包比较不同免疫亚型肿瘤组织中MCM的表达差异。通过加利福尼亚大学圣克鲁兹分校(University of California Santa Cruz,UCSC)Xena平台从TCGA数据库下载免疫亚型分类数据,共有6种泛癌免疫亚型。其中,免疫亚型C1指伤口愈合,C2指γ干扰素显著表达,C3指炎症,C4指淋巴细胞枯竭,C5指免疫沉默,C6指TGF-β显著表达。
1.8 肿瘤微环境与肿瘤干性分析利用UCSC Xena平台从TCGA数据库下载泛癌的肿瘤微环境及肿瘤干细胞相关数据,通过R 4.0.3软件“estimate”工具包评估每种肿瘤组织中基质细胞、免疫细胞及肿瘤细胞的比例,利用R 4.0.3软件的“ corrplot ”工具包分析MCM与肿瘤微环境成分比例的相关性。
干细胞评分是用于描述肿瘤细胞与干细胞相似性的指标,干细胞评分越高说明细胞的分化程度越低、干细胞特征越强。利用R 4.0.3软件的“corrplot”工具包分析MCM与干细胞评分的相关性。
2 结果 2.1 差异表达基因对宫颈癌组织和正常宫颈组织的测序结果进行表达差异分析,共筛选出336个差异表达基因,包括153个下调基因和183个上调基因(图 1)。

|
图 1 宫颈癌与正常宫颈组织基因测序整合数据的火山图 Fig 1 Volcano chart of integrated data of gene sequencing in cervical cancer and normal cervical tissues Red dots: Up-regulated genes; Green dots: Down-regulated genes; Black dots: Genes without significant difference in expression level. FC: Fold change; FDP: False discovery rate. |
2.2 GO和KEGG富集分析
GO富集分析结果(图 2A)显示,差异表达基因主要参与胶原蛋白细胞外基质、蛋白酶体核心复合体、内肽酶复合物、染色体区域等细胞组成,在细胞周期、DNA复制、细胞周期过程的负调控、对缺氧的反应等生物学过程中富集,同时可能涉及苏氨酸型内肽酶活性、硫化合物结合、肝素结合、胶原结合等分子功能。KEGG富集分析结果(图 2B)显示,差异表达基因主要富集于DNA复制、错配修复、前列腺癌、癌症转录失调、核苷酸切除修复等信号通路。
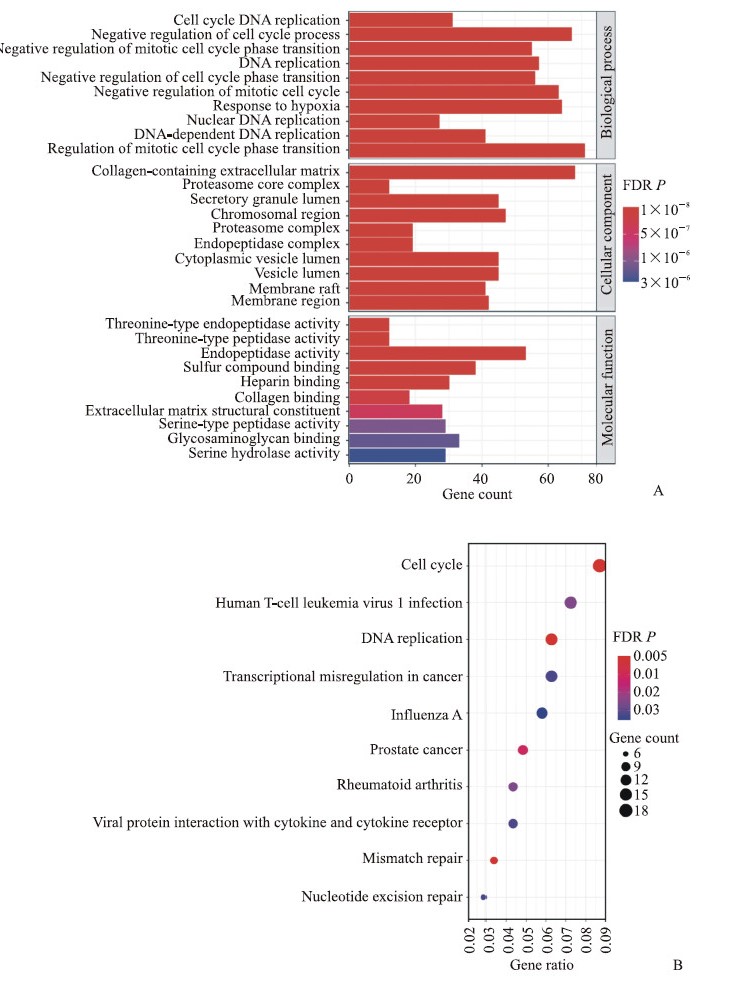
|
图 2 差异表达基因的GO和KEGG富集分析结果 Fig 2 Enrichment analyses of differentially expressed genes by GO and KEGG A: Top 10 GO terms of differentially expressed genes; B: KEGG pathways of differentially expressed genes. GO: Gene ontology; KEGG: Kyoto encyclopedia of genes and genomes; FDR: False discovery rate. |
2.3 PPI网络分析和生存分析筛选
通过STRING在线分析获得了包括336个节点和1 809个边的差异表达基因PPI网络(图 3)。核心交互网络用Cytoscape平台进行分子复合物检测(molecular complex detection,MCODE)分析,发现3个交互作用较强的模块,提示这3个基因群可能存在相关性(图 4)。

|
图 3 差异表达基因PPI网络分析图 Fig 3 PPI analysis of differentially expressed genes PPI network of differentially expressed genes by STRING. Circles: Genes; Lines: Protein interaction between genes; Results within the circle: Structure of proteins; Line color: Evidence of the interaction between the proteins. PPI: Protein-protein interaction. |
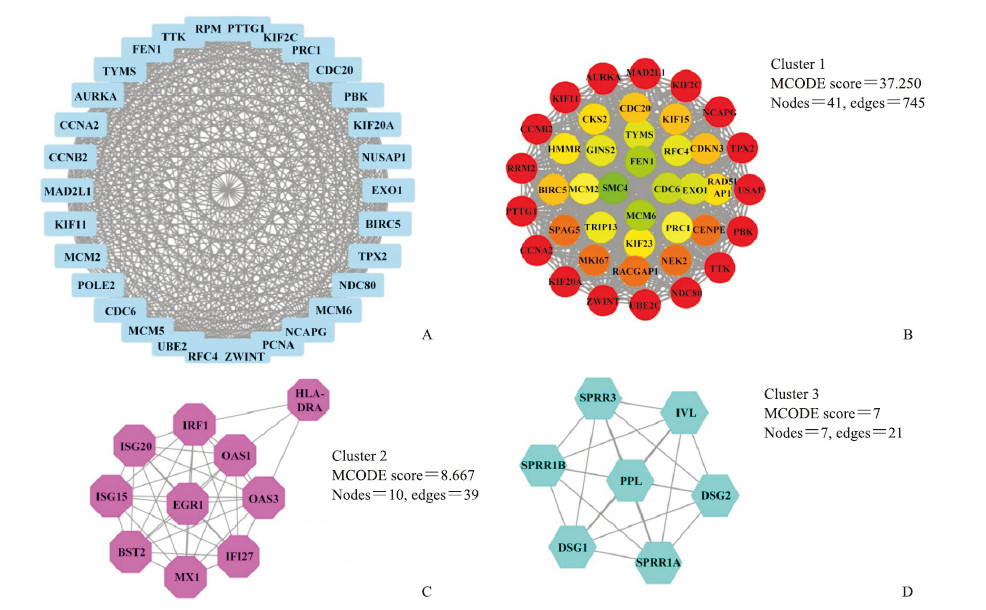
|
图 4 利用Cytoscape插件对PPI核心交互网络进行MCODE分析 Fig 4 MCODE analysis of PPI core integration network using Cytoscape plug-in A: PPI network of top 30 genes with the highest degree; B: MCODE result of cluster 1 network (green to red: MCODE score increased from low to high); C: MCODE result of cluster 2 network; D: MCODE result of cluster 3 network. PPI: Protein-protein interaction; MCODE: Molecular complex detection. |
取关联度排名前10的10个差异表达基因作为宫颈癌的重要基因,利用TCGA数据库对其进行Kaplan-Meier生存分析,以P<0.05为条件筛选出MCM2作为关键基因。结果(图 5)表明,MCM2表达与宫颈癌5年总生存率呈正相关(P=0.017),即MCM2高表达的宫颈癌患者生存时间更长。

|
图 5 不同MCM2表达水平宫颈癌患者的Kaplan-Meier生存分析结果 Fig 5 Kaplan-Meier survival analysis of cervical cancer patients with different MCM2 expression levels The median expression level was set as the cutoff value for the Kaplan-Meier curves. MCM2: Mini-chromosome maintenance protein 2. |
2.4 表达验证和细胞定位
再次利用GEPIA2网站验证MCM2在宫颈癌中的表达水平,该结果与基于GEO数据集的分析结果一致。与正常组织(n=13)相比,MCM2在宫颈癌组织(n=306)中高表达(P<0.05,图 6A)。HPA数据库的免疫组织化学(immunohistochemistry,IHC)分析结果显示,MCM2在宫颈癌组织中表达较高,在正常组织中表达较低(图 6B)。细胞中MCM2主要定位于细胞核(图 6C)。
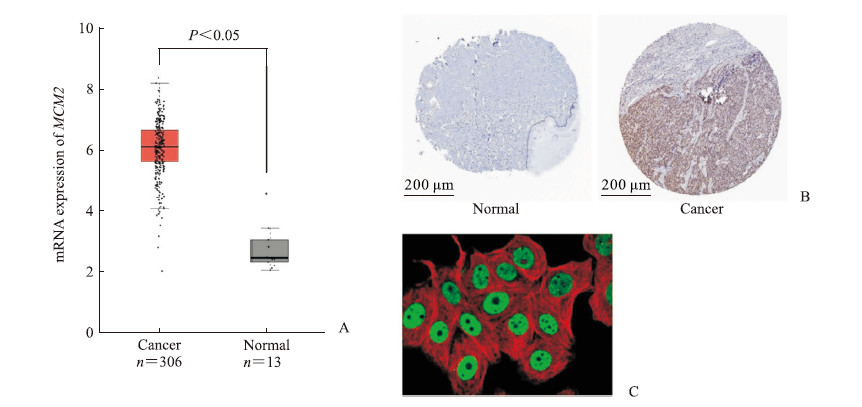
|
图 6 MCM2在宫颈癌中的表达验证和细胞定位结果 Fig 6 Expression and location of MCM2 in cervical cancer cells A: RNA expression of MCM2 analyzed by GEPIA2 based on the databases of TCGA and GTEx; B: Protein expression analysis of MCM2 in normal tissue and cervical cancer tissue by HPA-based immunohistochemistry database; C: Localization of MCM2 in cells (400×). Green: MCM2 antibody; Red: Microtubules. MCM2: Mini-chromosome maintenance protein 2; GEPIA2: Gene Expression Profiling Interactive Analysis; TCGA: The Cancer Genome Atlas; GTEx: Genotype-Tissue Expression. |
KEGG基因功能集富集分析结果表明,显著富集的信号通路涵盖了多方面生物学研究领域。(1)在肿瘤与免疫方面,包括Fc段γ受体(Fc γ receptor,FcγR)介导的吞噬作用、MAPK信号通路、Notch信号通路、自噬调控、T细胞受体信号通路和WNT信号通路等(图 7A)。(2)在细胞增殖、死亡与运动方面,包括黏附连接、细胞凋亡、细胞周期、DNA复制、肌动蛋白细胞骨架调节和RNA聚合酶等(图 7B)。(3)在清除和降解方面,包括胞吞作用、溶酶体、RNA降解、泛素介导的蛋白水解等(图 7C)。(4)在合成和代谢方面,包括胰岛素信号转导途径、过氧化物酶、嘌呤代谢、嘧啶代谢,以及缬氨酸、亮氨酸和异亮氨酸降解等(图 7D)。
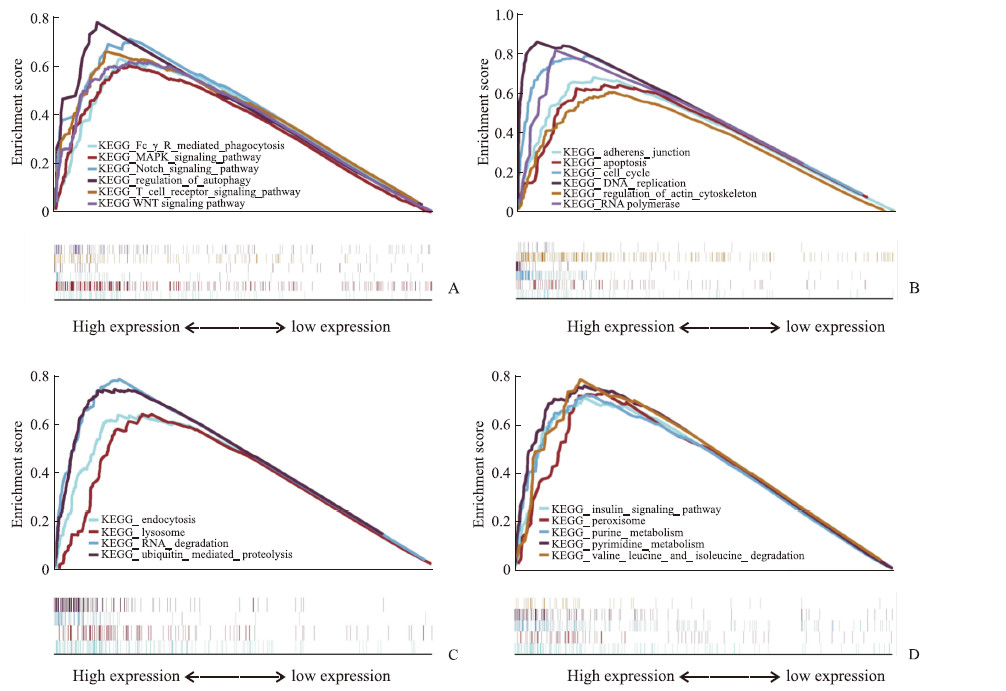
|
图 7 MCM2在宫颈癌中的KEGG基因功能集富集分析结果 Fig 7 KEGG gene set enrichment analysis results of MCM2 in cervical cancer A: Tumor and immunity; B: Cell proliferation, death, and motility; C: Scavenging and degradation; D: Synthesis and metabolism. MCM2: Mini-chromosome maintenance protein 2; KEGG: Kyoto encyclopedia of genes and genomes. |
2.5 MCM2在泛癌中的生存分析
Kaplan-Meier生存分析结果(图 5、图 8)显示,MCM2的表达水平与包括宫颈癌在内的11种癌症患者的生存率相关(P均<0.05),其中与宫颈癌(图 5)、弥漫大B细胞淋巴瘤(图 8B)、直肠腺癌(图 8H)和葡萄膜黑素瘤(图 8J)5年总生存率呈正相关;与肾上腺皮质癌(图 8A)、肾嫌色细胞癌(图 8C)、急性髓细胞样白血病(图 8D)、脑低级别胶质瘤(图 8E)、肝细胞肝癌(图 8F)、间皮瘤(图 8G)、肉瘤(图 8I)5年总生存率呈负相关。
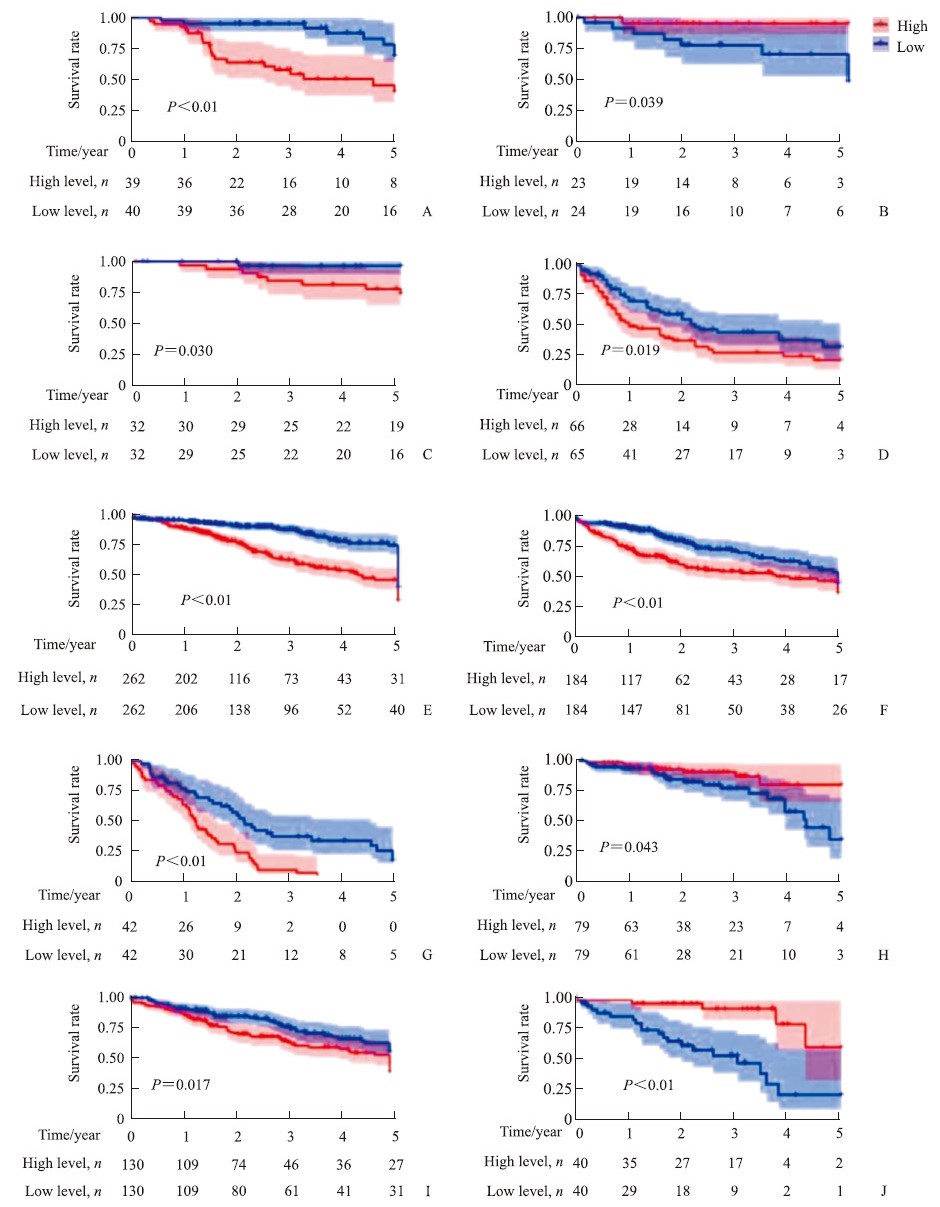
|
图 8 不同MCM2表达水平泛癌的Kaplan-Meier生存分析 Fig 8 Kaplan-Meier survival analysis of pan-cancer patients with different MCM2 expression levels A: Adrenocortical carcinoma; B: Diffuse large B-cell lymphoma; C: Kidney chromophobe; D: Acute myeloid leukemia; E: Brain lower grade glioma; F: Liver hepatocellular carcinoma; G: Mesothelioma; H: Rectum adenocarcinoma; I: Sarcoma; J: Uveal melanoma. The median expression level was set as the cutoff value for the Kaplan-Meier curves. MCM2: Mini-chromosome maintenance protein 2. |
2.6 MCM家族基因共表达分析
除了少于3个正常组织的队列外,基于TCGA数据库的21种癌症的热图(图 9A)显示,在大多数类型的癌症中MCM家族基因的表达上调,且宫颈癌组织中MCM的表达均增加。
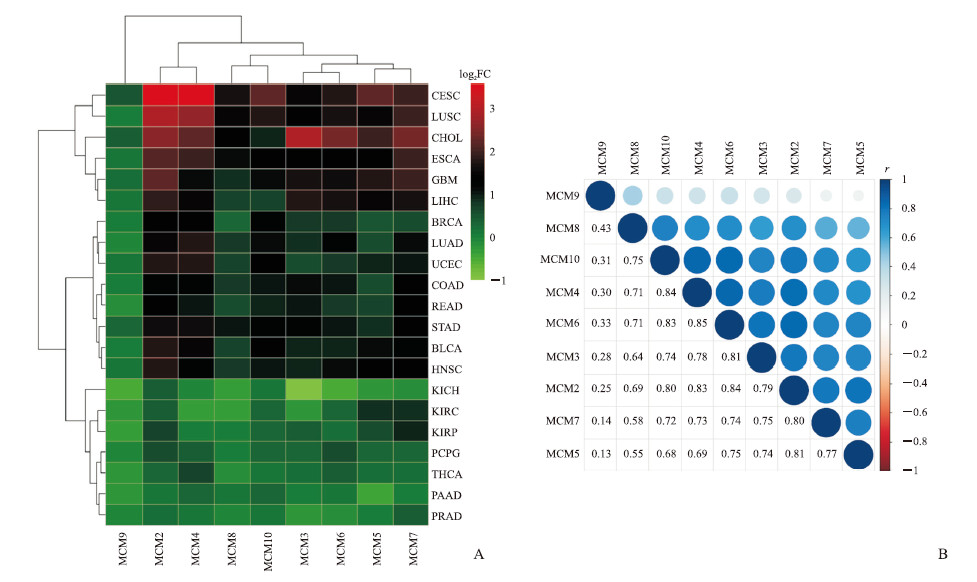
|
图 9 MCM家族基因在泛癌中的表达差异分析及基因共表达分析结果 Fig 9 Differential expression and co-expression of MCMs in pan-cancer A: Heatmap of MCMs in pan-caner; B: Correlation analysis of MCMs transcript in pan-cancer. MCM: Mini-chromosome maintenance protein; CESC: Cervical squamous cell carcinoma and endocervical adenocarcinoma; LUSC: Lung squamous cell carcinoma; CHOL: Cholangiocarcinoma; ESCA: Esophageal carcinoma; GBM: Glioblastoma multiforme; LIHC: Liver hepatocellular carcinoma; BRCA: Breast invasive carcinoma; LUAD: Lung adenocarcinoma; UCEC: Uterine corpus endometrial carcinoma; COAD: Colon adenocarcinoma; READ: Rectum adenocarcinoma; STAD: Stomach adenocarcinoma; BLCA: Bladder urothelial carcinoma; HNSC: Head and neck squamous cell carcinoma; KICH: Kidney chromophobe; KIRC: Kidney renal clear cell carcinoma; KIRP: Kidney renal papillary cell carcinoma; PCPG: Pheochromocytoma and paraganglioma; THCA: Thyroid carcinoma; PAAD: Pancreatic adenocarcinoma; PRAD: Prostate adenocarcinoma. |
利用TCGA数据库对泛癌中MCM进行了Pearson相关性分析,结果(图 9B)表明大多数MCM成员之间存在正相关性,如MCM4和MCM6(r=0.85)、MCM2和MCM6(r=0.84)、MCM4和MCM10(r=0.84)、MCM2和MCM4(r=0.83)、MCM6和MCM10(r=0.83)、MCM3和MCM6(r=0.81)、MCM2和MCM5(r=0.81)、MCM2和MCM7(r=0.80)。
2.7 单因素Cox回归分析单因素Cox回归结果(图 10)显示,MCM2和MCM5是宫颈癌的保护因素,高表达MCM2、MCM5的宫颈癌患者可能预后更好。同时,大部分MCM家族成员是肾上腺皮质癌、肾嫌色细胞癌和间皮瘤的危险因素,表明MCM可能在这些癌症的进展中起重要作用。
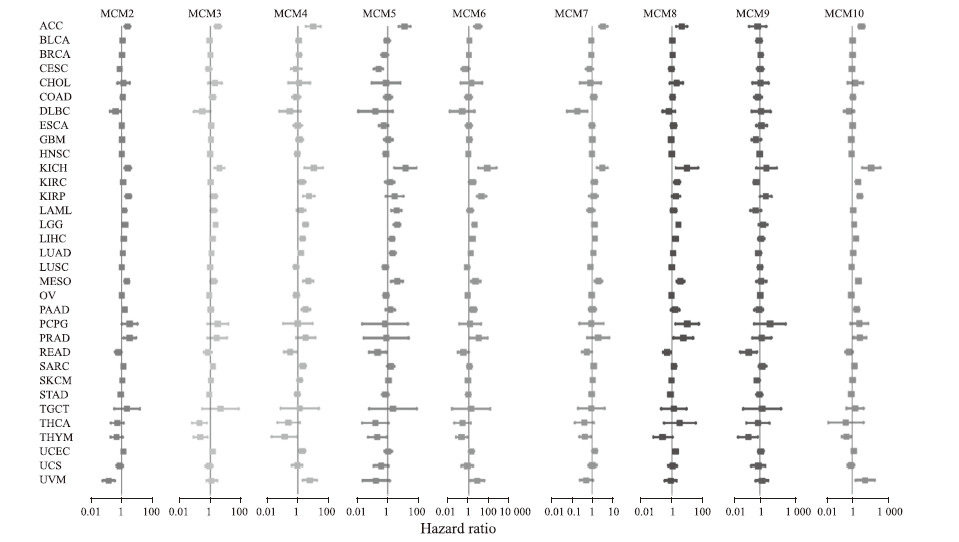
|
图 10 MCM家族基因影响泛癌预后的单因素Cox回归分析 Fig 10 Univariate Cox regression analysis of MCMs influencing prognosis of pan-cancer patients MCM: Mini-chromosome maintenance protein; ACC: Adrenocortical carcinoma; BLCA: Bladder urothelial carcinoma; BRCA: Breast invasive carcinoma; CESC: Cervical squamous cell carcinoma and endocervical adenocarcinoma; CHOL: Cholangiocarcinoma; COAD: Colon adenocarcinoma; DLBC: Lymphoid neoplasm diffuse large B-cell lymphoma; ESCA: Esophageal carcinoma; GBM: Glioblastoma multiforme; HNSC: Head and neck squamous cell carcinoma; KICH: Kidney chromophobe; KIRC: Kidney renal clear cell carcinoma; KIRP: Kidney renal papillary cell carcinoma; LAML: Acute myeloid leukemia; LGG: Brain lower grade glioma; LIHC: Liver hepatocellular carcinoma; LUAD: Lung adenocarcinoma; LUSC: Lung squamous cell carcinoma; MESO: Mesothelioma; OV: Ovarian serous cystadenocarcinoma; PAAD: Pancreatic adenocarcinoma; PCPG: Pheochromocytoma and paraganglioma; PRAD: Prostate adenocarcinoma; READ: Rectum adenocarcinoma; SARC: Sarcoma; SKCM: Skin cutaneous melanoma; STAD: Stomach adenocarcinoma; TGCT: Testicular germ cell tumor; THCA: Thyroid carcinoma; THYM: Thymoma; UCEC: Uterine corpus endometrial carcinoma; UCS: Uterine carcinosarcoma; UVM: Uveal melanoma. |
2.8 免疫亚型分析
见图 11,MCM家族成员在C1亚型和C2亚型中表达均相对较高;MCM家族成员在不同免疫亚型泛癌组织中的表达水平有所不同差异有统计学意义(P<0.01),可能参与肿瘤免疫的生物学过程。
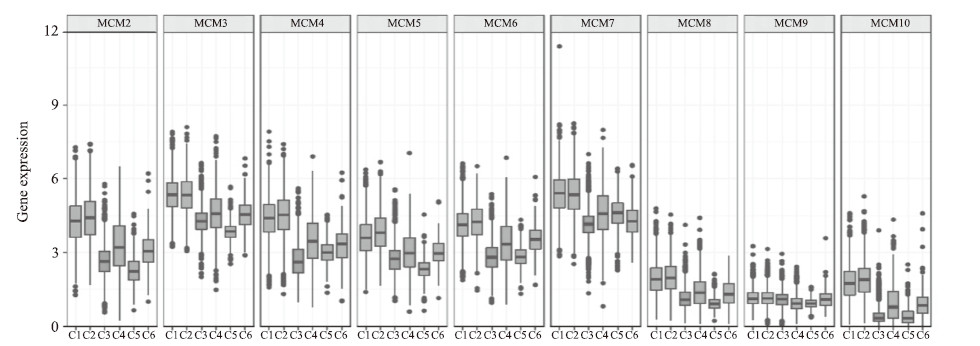
|
图 11 MCM家族基因在不同免疫亚型泛癌组织中的表达 Fig 11 Expression of MCMs in pan-cancer tissues with different immune types C1: Wound healing; C2: Interferon-γ dominant; C3: Inflammatory; C4: Lymphocyte depleted; C5: Immunologically quiet; C6: Transforming growth factor-β dominant. There were significant differences in the expression levels of MCMs among different immune subtypes (P < 0.01). MCM: Mini-chromosome maintenance protein. |
2.9 肿瘤微环境相关性及干细胞分析
MCM家族成员的肿瘤相关性分析结果显示,大多数肿瘤中MCM家族成员的表达与基质评分(图 12A)和免疫评分(图 12B)呈负相关,而与肿瘤纯度(图 12C)呈正相关,表明MCM家族基因高表达的肿瘤组织可能具有较低比例的基质细胞、免疫细胞及较高比例的肿瘤细胞。尤其在多形性胶质母细胞瘤中,大多数MCM家族成员均与肿瘤微环境的组成比例呈高度相关,表明MCM家族成员高表达的多形性胶质母细胞瘤组织中可能含有高比例的肿瘤细胞和较低比例的基质细胞及免疫细胞,该结果提示MCM家族成员可能是影响多形性胶质母细胞瘤微环境的重要因素。
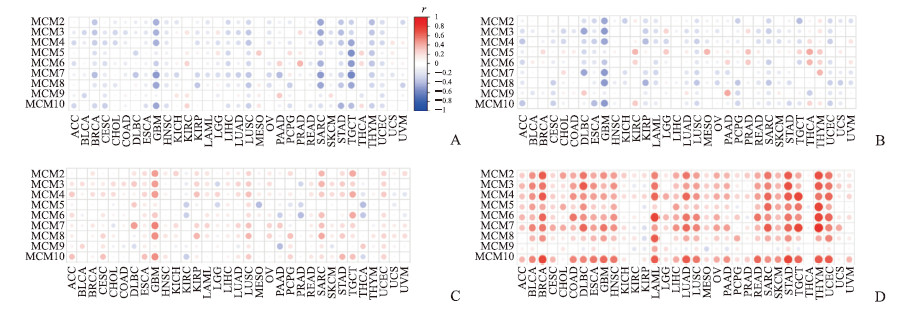
|
图 12 MCM家族基因与肿瘤免疫微环境和肿瘤干性的相关性 Fig 12 Correlation analysis between MCMs and tumor immune microenvironment and tumor stemness A: Stromal score; B: Immune score; C: Tumor purity; D: Stemness score. MCM: Mini-chromosome maintenance protein; ACC: Adrenocortical carcinoma; BLCA: Bladder urothelial carcinoma; BRCA: Breast invasive carcinoma; CESC: Cervical squamous cell carcinoma and endocervical adenocarcinoma; CHOL: Cholangiocarcinoma; COAD: Colon adenocarcinoma; DLBC: Lymphoid neoplasm diffuse large B-cell lymphoma; ESCA: Esophageal carcinoma; GBM: Glioblastoma multiforme; HNSC: Head and neck squamous cell carcinoma; KICH: Kidney chromophobe; KIRC: Kidney renal clear cell carcinoma; KIRP: Kidney renal papillary cell carcinoma; LAML: Acute myeloid leukemia; LGG: Brain lower grade glioma; LIHC: Liver hepatocellular carcinoma; LUAD: Lung adenocarcinoma; LUSC: Lung squamous cell carcinoma; MESO: Mesothelioma; OV: Ovarian serous cystadenocarcinoma; PAAD: Pancreatic adenocarcinoma; PCPG: Pheochromocytoma and paraganglioma; PRAD: Prostate adenocarcinoma; READ: Rectum adenocarcinoma; SARC: Sarcoma; SKCM: Skin cutaneous melanoma; STAD: Stomach adenocarcinoma; TGCT: Testicular germ cell tumors; THCA: Thyroid carcinoma; THYM: Thymoma; UCEC: Uterine corpus endometrial carcinoma; UCS: Uterine carcinosarcoma; UVM: Uveal melanoma. |
MCM家族成员与肿瘤干细胞评分的相关性分析结果(图 12D)显示,大多数MCM家族成员(尤其MCM2)的表达与肿瘤干性评分呈正相关,这意味着MCM家族成员的表达可能会促进癌细胞干性特征的表达。
3 讨论近年来,由于人乳头瘤病毒疫苗的普及和手术方式的不断革新,宫颈癌的发病率和死亡率一直在稳步下降,但宫颈癌晚期患者仍然有较高的复发风险,对于远处转移或复发的患者始终缺少有效的治疗方式。新兴的生物信息学是一个高效且便捷的工具,可用来探索宫颈癌进展的分子机制,从而寻找宫颈癌的治疗新策略和预后标志物。
MCM2是一种基因编码蛋白,属于高度保守的MCM家族,后者含有MCM1~10[5]。MCM1是MADS-box转录因子家族的成员[6],因超出了TCGA转录谱的范围而未包含在本研究中。MCM2~7复合物(双环异六聚体)是真核复制解旋酶的核心成分,在DNA复制起始和延伸中起关键作用。静止期细胞中MCM2~7复合物的含量非常低,在增殖和转化的细胞中,MCM2~7复合物在G1期开始增多,在G1末期和S期早期达到顶峰,并最终与染色质结合。由于MCM2~7复合物与细胞增殖过程周期性变化一致,已被认为是S期细胞的标志物和特定的增殖相关因子[7-8]。MCM8、9复合物在DNA复制中的功能仍不清楚,MCM10蛋白可能参与DNA损伤反应[9]。越来越多的证据表明,MCM家族成员在肿瘤进展中具有重要作用,可能是恶性肿瘤的潜在干预靶标[10-11]。在乳腺癌[12]、卵巢癌[13]、宫颈癌[14]、肾细胞癌[15]中,MCM2蛋白的表达与肿瘤增殖呈正相关。MCM4的表达水平可能与食管癌的病理分期有关[16]。MCM7基因多态性可能与急性髓细胞白血病的复发和总生存期密切相关[17]。以上这些证据提示,MCM家族成员对肿瘤的增殖能力和恶性程度至关重要,且在不同的恶性肿瘤中的表达及作用可能不同。
本研究结果表明,MCM2的mRNA和蛋白均在人类宫颈癌组织中高表达,且与宫颈癌的预后相关。本研究还探讨了宫颈癌中MCM2的相关途径,结果显示恶性肿瘤中的MAPK和自噬信号通路及T细胞受体信号通路均显著富集。利用泛癌数据进行分析显示,MCM2与11种类型的肿瘤(包括宫颈癌)预后相关。本研究还探讨了MCM2~10在泛癌中的表达和功能,大多数MCM家族成员在肿瘤组织中上调,并且基因共表达分析显示多数MCM家族基因之间存在正相关关系,MCM家族成员可能与肿瘤微环境和肿瘤免疫亚型相关。
本研究发现,在泛癌中MCM家族成员与肿瘤干性呈正相关。研究显示MCM2表达下调可能会导致严重的干细胞缺乏[18]。在结肠癌中,NF-κB通过上调MCM2的表达维持肿瘤干性[19]。这些结果进一步证实MCM家族成员对肿瘤干性可能存在普遍的调控作用。
综上所述,MCM2可能成为宫颈癌诊疗和预后评估的一个潜在分子标志物;在泛癌中MCM家族成员的研究有广阔前景,特别是在免疫亚型、肿瘤微环境和干性特征方面。本研究基于生物信息学分析的研究结果,应通过动物实验和临床试验进行验证。
| [1] |
ARBYN M, WEIDERPASS E, BRUNI L, DE SANJOSÉ S, SARAIYA M, FERLAY J, et al. Estimates of incidence and mortality of cervical cancer in 2018: a worldwide analysis[J/OL]. Lancet Glob Health, 2020, 8: e191-e203. DOI: 10.1016/S2214-109X(19)30482-6.
|
| [2] |
HANNA T P, KING W D, THIBODEAU S, JALINK M, PAULIN G A, HARVEY-JONES E, et al. Mortality due to cancer treatment delay: systematic review and meta-analysis[J/OL]. BMJ, 2020, 371: m4087. DOI: 10.1136/bmj.m4087.
|
| [3] |
DIAZ-PADILLA I, MONK B J, MACKAY H J, OAKNIN A. Treatment of metastatic cervical cancer: future directions involving targeted agents[J]. Crit Rev Oncol Hematol, 2013, 85: 303-314. DOI:10.1016/j.critrevonc.2012.07.006 |
| [4] |
VORA C, GUPTA S. Targeted therapy in cervical cancer[J/OL]. ESMO Open, 2018, 3(Suppl 1): e000462. DOI: 10.1136/esmoopen-2018-000462.
|
| [5] |
ISHIMI Y. Regulation of MCM2-7 function[J]. Genes Genet Syst, 2018, 93: 125-133. DOI:10.1266/ggs.18-00026 |
| [6] |
SHORE P, SHARROCKS A D. The MADS-box family of transcription factors[J]. Eur J Biochem, 1995, 229: 1-13. DOI:10.1111/j.1432-1033.1995.tb20430.x |
| [7] |
TYE B K. MCM proteins in DNA replication[J]. Annu Rev Biochem, 1999, 68: 649-686. DOI:10.1146/annurev.biochem.68.1.649 |
| [8] |
SEDLACKOVA H, RASK M B, GUPTA R, CHOUDHARY C, SOMYAJIT K, LUKAS J. Equilibrium between nascent and parental MCM proteins protects replicating genomes[J]. Nature, 2020, 587: 297-302. DOI:10.1038/s41586-020-2842-3 |
| [9] |
CHATTOPADHYAY S, BIELINSKY A K. Human Mcm10 regulates the catalytic subunit of DNA polymerase-alpha and prevents DNA damage during replication[J]. Mol Biol Cell, 2007, 18: 4085-4095. DOI:10.1091/mbc.e06-12-1148 |
| [10] |
LI Z, XU X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication[J/OL]. Genes (Basel), 2019, 10: 331. DOI: 10.3390/genes10050331.
|
| [11] |
SEO Y S, KANG Y H. The human replicative helicase, the CMG complex, as a target for anti-cancer therapy[J/OL]. Front Mol Biosci, 2018, 5: 26. DOI: 10.3389/fmolb.2018.00026.
|
| [12] |
WOJNAR A, PULA B, PIOTROWSKA A, JETHON A, KUJAWA K, KOBIERZYCKI C, et al. Correlation of intensity of MT-Ⅰ/Ⅱ expression with Ki-67 and MCM-2 proteins in invasive ductal breast carcinoma[J]. Anticancer Res, 2011, 31: 3027-3033. |
| [13] |
LEVIDOU G, VENTOURI K, NONNI A, GAKIOPOULOU H, BAMIAS A, SOTIROPOULOU M, et al. Replication protein A in nonearly ovarian adenocarcinomas: correlation with MCM-2, MCM-5, Ki-67 index and prognostic significance[J]. Int J Gynecol Pathol, 2012, 31: 319-327. DOI:10.1097/PGP.0b013e31823ef92e |
| [14] |
BECCATI M D, BURIANI C, PEDRIALI M, ROSSI S, NENCI I. Quantitative detection of molecular markers ProEx C (minichromosome maintenance protein 2 and topoisomeraseⅡa) and MIB-1 in liquid-based cervical squamous cell cytology[J]. Cancer, 2008, 114: 196-203. DOI:10.1002/cncr.23496 |
| [15] |
MEHDI M Z, NAGI A H, NASEEM N. MCM-2 and Ki-67 as proliferation markers in renal cell carcinoma: a quantitative and semi-quantitative analysis[J]. Int Braz J Urol, 2016, 42: 1121-1128. DOI:10.1590/s1677-5538.ibju.2015.0388 |
| [16] |
HUANG X P, RONG T H, WU Q L, FU J H, YANG H, ZHAO J M, et al. MCM4 expression in esophageal cancer from southern China and its clinical significance[J]. J Cancer Res Clin Oncol, 2005, 131: 677-682. DOI:10.1007/s00432-005-0011-6 |
| [17] |
LEE J S, CHEONG H S, KOH Y, AHN K S, SHIN H D, YOON S S. MCM7 polymorphisms associated with the AML relapse and overall survival[J]. Ann Hematol, 2017, 96: 93-98. DOI:10.1007/s00277-016-2844-2 |
| [18] |
PRUITT S C, BAILEY K J, FREELAND A. Reduced Mcm2 expression results in severe stem/progenitor cell deficiency and cancer[J]. Stem Cells, 2007, 25: 3121-3132. DOI:10.1634/stemcells.2007-0483 |
| [19] |
WANG L, GUO J, ZHOU J, WANG D, KANG X, ZHOU L. NF-κB maintains the stemness of colon cancer cells by downregulating miR-195-5p/497-5p and upregulating MCM2[J/OL]. J Exp Clin Cancer Res, 2020, 39: 225. DOI: 410.1186/s13046-020-01704-w.
|
 2021, Vol. 42
2021, Vol. 42


