2. 太原理工大学大数据学院, 太原 030024;
3. 山西医科大学附属人民医院病理科, 太原 030000;
4. 山西医科大学第二医院病理科, 太原 030000
2. College of Big Data, Taiyuan University of Technology, Taiyuan 030024, Shanxi, China;
3. Department of Pathology, People's Hospital Affiliated to Shanxi Medical University, Taiyuan 030000, Shanxi, China;
4. Department of Pathology, the Second Hospital of Shanxi Medical University, Taiyuan 030000, Shanxi, China
慢性肾脏病(chronic kidney disease,CKD)是危害公众健康的常见病和多发病。肾小球是由相互交织的毛细血管构成,作为肾皮质的组织结构,主要负责血液过滤。而足细胞增殖异常和凋亡、足突广泛融合可导致肾小球滤过屏障受损,进而造成慢性进行性肾功能损害,最终发生肾功能衰竭[1]。CKD患者的主要组织学表现是肾小球硬化及肾间质纤维化[2],因而临床上患者是否存在肾小球硬化及有效阻断肾小球硬化形成并延缓其进展成为治疗CKD的关键环节。肾脏疾病的诊断依赖于组织学分析,病理科医师通过显微镜高强度阅片耗时、费力且误诊率高。国内病理科医师缺口大、病理工作流程冗杂、高强度病理阅片等医疗体系问题不容忽视。
人工智能(AI)在肾脏病学的应用主要体现在超声影像[3-4]、转录组学[5]、蛋白质组学[6]。近年来深度学习模型卷积神经网络(convolutional neural network,CNN)被引入肾脏组织病理学,通过全视野数字切片(whole slide image,WSI)获得数字化图像,而深度学习技术在输入WSI到输出(如结构检测)的转换过程中自主学习复杂的结构[7]。这些结果证实深度学习在评估数字化人体肾脏活检中复杂的组织学结构方面适应性良好。对标准矩形梯度朝向直方图(rectangular histogram of oriented gradient,r-HOG)特征向量改进后的分段梯度朝向直方图(segmental histogram of oriented gradient,s-HOG)检测过程相对复杂,所检测到的肾小球精确度和召回率分别为87.4%、89.7%,F值为0.866[8]。CNN模型能够从正常、部分硬化和整体硬化肾小球图像中准确区分非肾小球的准确度达到(92.67±2.02)%,Kappa值为0.868 1±0.039 2[9]。Bukowy等[10]开发了由2种机器学习分类器组成的肾小球定位器,用于自动识别整个肾脏切片中的肾小球。第一阶段由基于区域的CNN组成得到肾小球候选图像,第二阶段是CNN最终分类为“肾小球”或“背景”。大鼠和人类肾脏的平均精确度分别为96.94%和80.2%,平均召回率分别为96.79%和81.67%。
肾小球分析是肾脏活检组织学评估的重要步骤之一。有研究者提出采用弱监督的方法来识别与分割肾组织病理学上的WSI肾小球,但效果欠佳,为网络学习过程增加了难度,考虑与切片染色的质量有关[11]。也有研究者通过无监督方法识别肾小球,但最后得到肾小球的数量较少且耗时长[12]。我们前期采用快速区域卷积神经网络(faster region-convolutional neural network,faster R-CNN)检测方法检测肾小球与此分类器有相似之处,但是“接近但不正确”的边界框的假阳性问题尚不能解决。当前针对CNN对肾小球的检测结果参差不齐,无论采用监督还是无监督模式,肾小球边界的不确定性、染色质量差异性都为检测结果增加了难度。本研究提供了一种准确检测肾并定位肾小球图像的工具,该方法能快速、有效地自动检测肾小球,不仅为病理医师节省时间,也提高了肾脏病理专业医师的诊断效率。
1 资料和方法 1.1 研究对象本研究为回顾性研究,通过山西医科大学附属人民医院和山西医科大学第二医院伦理委员会审批。收集2014-2019年山西医科大学第二医院和山西医科大学附属人民医院2 870份行肾穿刺活检术患者六胺银(periodic acid-silver metheramine,PASM)染色、过碘酸雪夫(periodic acid-Schiff,PAS)染色、H-E染色和Masson染色4种染色肾脏病理切片,所收集患者的病因包括IgA肾病、膜性肾病、糖尿病肾病、高血压肾损害、狼疮性肾炎、过敏性紫癜性肾炎。所有患者病历资料齐全。
1.2 建立肾小球数据集运用KF-PRO-005-EX数字幻灯片扫描仪(宁波江丰生物信息技术有限公司)扫描病理切片,目镜分辨率为0.25毫米/像素,并以KFB格式通过远程病理系统传输到云端并储存,每张图像的大小为25 000像素×35 000像素,包含8~9亿像素点。转换为无损分辨率的SVS格式。PASM染色较其他3染色方式可以较好地将肾小球从肾小管间质等背景结构中区别出来,故本研究选用PASM染色切片。剔除模糊不清、染色质量差的图像数据,同时满足时间为2017-2019年3年内两院染色质量等无明显差异的病理切片图像,最终得到1 180张PASM染色图像。将所有样本随机划分为包括940张图像的训练集和包括240张图像的测试集。
在放大400倍0.25毫米/像素的图像下采样放大200倍0.5 mm/像素图像。再对数字病理切片的组织区域进行切割,每张图像都在放大400倍下直接进行重叠裁剪,将肾组织病理切片图像裁剪为多个局部,每张图像的大小为2 000像素×2 000像素,以适合神经网络读取和学习。所有的图像都由2名经验丰富的病理科主治医师标注和纠正。
1.3 训练数据、建立模型对WSI动态比例下采样后输入测试图像。每张局部图片(patch)由2 000像素×2 000像素压缩成600像素×600像素后作为测试图像。肾小球数据集制作过程如图 1所示。
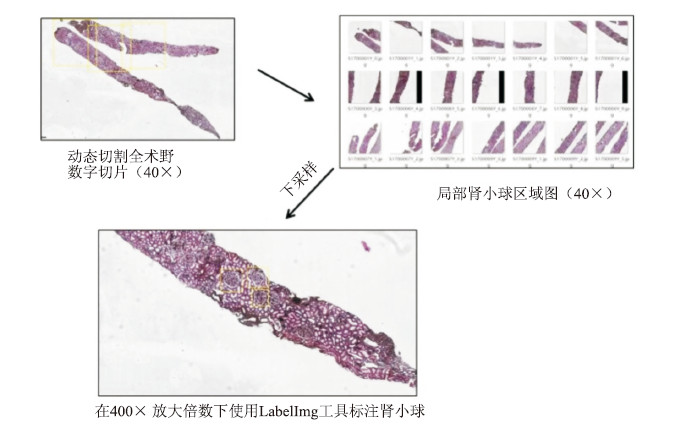
|
图 1 肾小球数据集制作过程 |
Faster R-CNN和级联区域卷积神经网络(cascade region-convolutional neural network,cascade R-CNN)结构特征见图 2[13]。第一阶段是进行肾小球的候选区域提取的子网络(H0)做初步检测假设,应用于整张图片。第二阶段进行候选识别区域(region of interest,ROI)检测的子网络(H1),最终分类分数(c)和为每个假设分配边界框(b)。检测模型是基于第一阶段的输出进行训练,对输出的预测范围(bounding boxes,bbox)的标签界定由前往后分别采用交并比(intersection over union,IOU)值为0.5、0.6、0.7,训练2万次得到大概的轮廓特征;为了更精细地找到细微结构,将数据再进行1 000像素×1 000像素训练1万次。Cascade R-CNN是迭代式的bbox回归,前一个检测模型回归得到的bbox坐标初始化下一个检测模型的bbox,每次迭代后边界框的分布都会发生变化。重采样是通过前一阶段的回归输出实现的,除标准水平图像翻转外,不做数据增强处理。
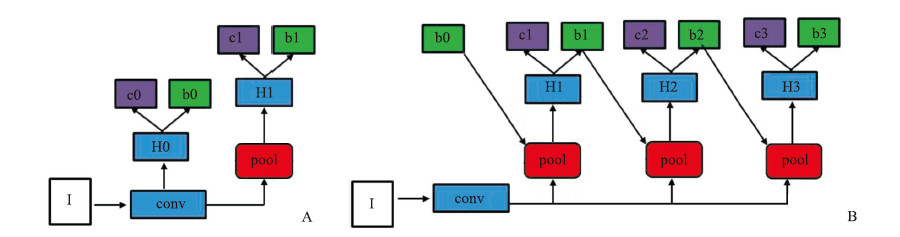
|
图 2 2种区域卷积神经网络结构特征[13] A:快速区域卷积神经网络;B:级联区域卷积神经网络.I:图片输入;conv:骨干卷积;pool:区域特征提取;H0~H3:各阶段候选区域提取的子网络;c0~c3:分类分数;b0~b3:边界框的回归结果. |
本实验使用cascade R-CNN网络训练模型,在Linux服务器上运用4个NVIDIA Tesla k80 GPU加速,采用Python 3.6.4语言和openslide工具包,基于TensorFlow框架实现cascade R-CNN算法。所有训练样本图片都框级标注出相对完整的肾小球。
1.4 性能评价方法所提出的模型的性能是基于被正确检测到的肾小球的数量来衡量的。在此基础上,使用精确度、召回率和F1值进行评估[14]。精确度是所有被检测到的样本中被正确检测的样本所占的比例,计算公式:精确度=TP/(TP+FP),其中TP表示正样本(肾小球)预测正确的个数,FP表示被错分为正样本的负样本个数。召回率是所有检测到的正样本占所有应该被正确检测到的样本的比例,计算公式:召回率=TP/(TP+FN),其中FN表示被错分为负样本(非肾小球)的正样本个数。F1值是精确度和召回率的调和均值,计算公式:F1值=2×(召回率×精确度)/(召回率+精确度)。
1.5 肾脏病理医师识别测试测试集(240张图像)分别由3名工作至少3年的肾脏病理科医师阅片,并利用labelImg阅读标记。计算3名病理医师识别肾小球的精确度和时间。
1.6 统计学处理应用SPSS 22.0软件对数据资料进行统计学处理。计量资料以x±s表示,病理医师与网络识别肾小球精确度比较采用独立样本t检验,3名病理医师识别每张图像肾小球区域的时间比较采用方差分析。计数资料以张数表示。检验水准(α)为0.05。
2 结果 2.1 数据分析训练集940张图像,重叠切割得到5 344张3 000像素×3 000像素的肾小球区域图像,42 112个肾小球的边界框由病理医师标注。测试集的240张图像重叠切割得到1 274张3 000像素×3 000像素的肾小球区域图像。
2.2 cascade R-CNN、faster R-CNN算法比较cascade R-CNN算法通过采用不同的IOU阈值训练,以提升faster R-CNN的性能;文献提出基于faster R-CNN算法肾组织病理切片中肾小球识别精确度、召回率、F1值分别达到97.7%、92.9%、95.2%[15]或93.9%、91.8%、92.8%[16];而本研究通过cascade R-CNN算法模型识别肾小球精确度、召回率、F1值分别达到93.90%、98.00%、95.91%。前期基于faster R-CNN识别肾小球的准确度采用均值平均精确度确定每个模型的性能,得到平均精确度为94.37%,整张玻片图像处理时间约为1 s[17]。本实验结果显示,cascade R-CNN网络训练完成的深度学习模型处理整张玻片图像的时间约为1 s。cascade R-CNN是在faster R-CNN的基础上级联了几个不同IOU阈值的检测网络的、基于深度学习的目标检测算法。就检测结构来说,特征提取耗时最多。根据本实验结果,尽管cascade R-CNN增加了较多的参数,但未对检测速度造成明显影响。
2.3 CNN模型与病理医师诊断结果对比3名病理医师识别每张图像肾小球区域时间分别为(3.57±0.05)s、(4.57±0.07)s、(3.98±0.02)s,差异无统计学意义(P>0.05)。3名病理医师识别肾小球的精确度分别为88.08%、89.69%、89.98%,差异亦无统计学意义(P>0.05)。cascade R-CNN算法对于肾小球识别每张图像肾小球区域时间为(0.20±0.02)s,精确度为93.90%,高于3名病理医师的平均精确度(89.25%),差异有统计学意义(t=-5.607,P=0.009)。
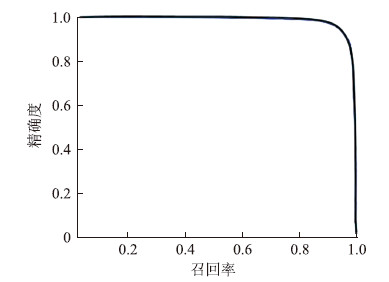
2.4 cascade R-CNN训练模型的质量检测在测试集上测试cascade R-CNN训练模型得到结果如图 3、4所示,边缘化的肾小球可能会被病理医师漏诊(图 4A);肾小球内毛细血管结构可能使其显示不明显,而被误认为是肾小管(图 4B);肾小球硬化后球内结构异常,可能导致病理医师漏掉标注(图 4C、4D)。但对于图 4A~4C中的肾小球,cascade R-CNN算法可以很快识别并定位。准确率与召回率曲线图(图 5)显示,通过cascade R-CNN算法自动学习获得的特征有很好的表达性。
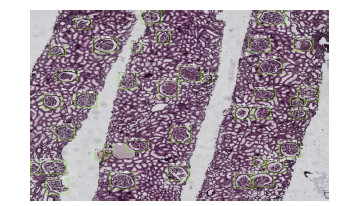
|
图 3 级联区域卷积神经网络算法检测肾小球六胺银染色图像(400×) |
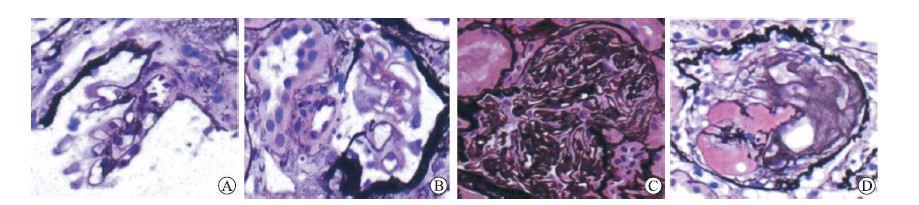
|
图 4 级联区域卷积神经网络算法检测形状各异的肾小球六胺银染色图像(400×) A:边缘化的肾小球;B:结构不清晰的肾小球;C:全部硬化性肾小球(分界不清);D:全部硬化性肾小球(肾小球内部结构发生明显变化). |
|
图 5 级联区域卷积神经网络算法的精确度与召回率曲线图 |
3 讨论
肾小球是肾脏疾病在病理诊断过程中检查的主要病变部位,检出和分类病变的肾小球是肾脏疾病病理诊断的基础环节。在本研究中识别肾小球及肾小球的准确定位为肾小球病变疾病辅助诊断打下基础。原病理切片的肾小球结构尽管较为明显,但同时存在原切片肾小球组织学形态多样、肾小球边缘化(不完整)、染色不均等情况,肾小球的异质性为病理医师标注注释集及训练网络模型带来挑战。肾小球在组织学图像中通常是椭圆形的,有研究者通过最小化肾小球标注边界(椭圆形)发现肾小球边界与横切的肾小管和小动脉有相似之处,说明采用椭圆形标注得到的拟合方式有很高的假阳性率[18]。本实验采用了常用的框级标注,有效避免了可能由标注方式造成的肾小球高假阳性,为后续研究任务识别肾小球内部病理特征奠定基础。
由网络进行目标检测工作包括目标分类、回归。cascade R-CNN采用多阈值检测子网络的级联结构,级联顺序按照阈值由小到大排列。检测器依次级联设计方式对假阳性的边界框更具有选择性[13]。算法的前半部分同faster R-CNN,后一部分由区域生成网络(region proposal network,RPN),即网络训练模型后通过映射关系生成一系列可能存在肾小球为目标的候选框,IOU为0.5时正样本数量足够,保证模型不会过拟合,从而保障准确度。并且阈值为0.5时对于低IOU样本位置的修正效果最显著,而当修正后的高IOU由更高阈值的回归器进行修正可大大提高位置的准确性。这种方法与检测数据集的增强方法有相似之处[19]。本实验采用cascade R-CNN模型的精确度、召回率分别达到93.90%、98.00%,F1值为95.91%。值得肯定的是,在本实验中cascade R-CNN测试结果得到的召回率高于其他研究者通过faster R-CNN对肾小球识别研究结果(召回率均小于93%)[15-16]。在病理诊断方面,召回率更为重要,它是医学诊断时判断漏诊情况的一个重要指标。召回率与精确度是2个相对的指标,想要提高其中1个则另1个会降低,本实验中召回率达到了较好效果,为病理医师漏诊时做了填补,而精确度也是很可观的。
病理医师通过显微镜观察病理切片肾小球病变特征确定肾脏病变类型,但观察肾小球包括肾小球个数是一个繁杂的过程。本实验将训练的分类模型的精确度与工作年限至少3年的病理医师的识别结果进行比较,结果表明模型鉴别肾小球的能力优于病理医师。并且模型处理整张玻片图像的时间约为1 s,体现了运用cascade R-CNN网络运行的高效率,说明使用cascade结构识别肾小球是可行且有效的。
后续的工作是对肾小球病变类型进行自动识别,将膜性肾病作为研究模型批量从原图上切割、提取出单独的肾小球,对基底膜部分做像素级分类,标注空泡样变性、“钉突”样改变、假双轨、基底膜明显增厚等特征,以实现一种对膜性肾病肾小球病灶识别以判断病变的深度学习算法,最终辅助病理医师通过AI阅片做出肾脏疾病诊断。
| [1] |
HELAL I, FICK-BROSNAHAN G M, REED-GITOMER B, SCHRIER R W. Glomerular hyperfiltration: definitions, mechanisms and clinical implications[J]. Nat Rev Nephrol, 2012, 8: 293-300. DOI:10.1038/nrneph.2012.19 |
| [2] |
MUÑOZ-FELIX J M, OUJO B, LOPEZ-NOVOA J M. The role of endoglin in kidney fibrosis[J/OL]. Expert Rev Mol Med, 2014, 16: e18. DOI: 10.1017/erm.2014.20.
|
| [3] |
COY H, HSIEH K, WU W, NAGARAJAN M B, YOUNG J R, DOUEK M L, et al. Deep learning and radiomics: the utility of Google TensorFlowTM Inception in classifying clear cell renal cell carcinoma and oncocytoma on Multiphasic CT[J]. Abdom Radiol (NY), 2019, 44: 2009-2020. DOI:10.1007/s00261-019-01929-0 |
| [4] |
BLACK K M, LAW H, ALDOUKHI A, DENG J, GHANI K R. Deep learning computer vision algorithm for detecting kidney stone composition[J]. BJU Int, 2020, 125: 920-924. DOI:10.1111/bju.15035 |
| [5] |
KONG Y C, YU T W. A graph-embedded deep feedforward network for disease outcome classification and feature selection using gene expression data[J]. Bioinformatics, 2018, 34: 3727-3737. DOI:10.1093/bioinformatics/bty429 |
| [6] |
VITSIOS D, PETROVSKI S. Mantis-ml: disease-agnostic gene prioritization from high-throughput genomic screens by stochastic semi-supervised learning[J]. Am J Hum Genet, 2020, 106: 659-678. DOI:10.1016/j.ajhg.2020.03.012 |
| [7] |
HERMSEN M, DE BEL T, DEN BOER M, STEENBERGEN EJ, KERS J, FLORQUIN S, et al. Deep learning-based histopathologic assessment of kidney tissue[J]. J Am Soc Nephrol, 2019, 30: 1968-1979. DOI:10.1681/ASN.2019020144 |
| [8] |
KATO T, RELATOR R, NGOUV H, HIROHASHI Y, TAKAKI O, KAKIMOTO T, et al. Segmental HOG: new descriptor for Glomerulus detection in kidney microscopy image[J/OL]. BMC Bioinformatics, 2015, 16: 316. DOI: 10.1186/s12859-015-0739-1.
|
| [9] |
KANNAN S, MORGAN L A, LIANG B, CHEUNG M G, LIN C Q, MUN D, et al. Segmentation of glomeruli within trichrome images using deep learning[J]. Kidney Int Rep, 2019, 4: 955-962. DOI:10.1016/j.ekir.2019.04.008 |
| [10] |
BUKOWY J D, DAYTON A, CLOUTIER D, MANIS A D, STARUSCHENKO A, LOMBARD J H, et al. Region-based convolutional neural nets for localization of glomeruli in trichrome-stained whole kidney sections[J]. J Am Soc Nephrol, 2018, 29: 2081-2088. DOI:10.1681/ASN.2017111210 |
| [11] |
GADERMAYR M, ESCHWEILER D, JEEVANESAN A, KLINKHAMMER B M, BOOR P, MERHOF D. Segmenting renal whole slide images virtually without training data[J]. Comput Biol Med, 2017, 90: 88-97. DOI:10.1016/j.compbiomed.2017.09.014 |
| [12] |
KOTYK T, DEY N, ASHOUR A S, BALAS-TIMAR D, CHAKRABORTY S, ASHOUR A S, et al. Measurement of glomerulus diameter and Bowman's space width of renal albino rats[J]. Comput Methods Programs Biomed, 2016, 126: 143-153. DOI:10.1016/j.cmpb.2015.10.023 |
| [13] |
CAI Z, VASCONCELOS N. Cascade R-CNN: high quality object detection and instance segmentation[J]. IEEE Trans Pattern Anal Mach Intell, 2021, 43: 1483-1498. DOI:10.1109/TPAMI.2019.2956516 |
| [14] |
MAHMOOD T, ARSALAN M, OWAIS M, LEE M B, PARK K R. Artificial intelligence-based mitosis detection in breast cancer histopathology images using faster R-CNN and deep CNNs[J/OL]. J Clin Med, 2020, 9: 749. DOI: 10.3390/jcm9030749.
|
| [15] |
杨会, 张兴娜, 姜秋竹, 原成英, 屈重霄, 刘云霄, 等. 基于Faster R-CNN算法开发的肾小球病理人工智能识别系统的速度与效率分析[J]. 临床肾脏病杂志, 2020, 20: 189-193. DOI:10.3969/j.issn.1671-2390.2020.03.003 |
| [16] |
LIU R, WANG L, HE J, CHEN W. Towards staining independent segmentation of glomerulus from histopathological images of kidney[J/OL]. bioRxiv, 2019. DOI: http://dx.doi.org/10.1101/821181.
|
| [17] |
KAWAZOE Y, SHIMAMOTO K, YAMAGUCHI R, SHINTANI-DOMOTO Y, UOZAKI H, FUKAYAMA M, et al. Faster R-CNN-based glomerular detection in multistained human whole slide images[J/OL]. J Imaging, 2018, 4: 91. DOI: 10.3390/jimaging4070091.
|
| [18] |
MARÉE R, DALLONGEVILLE S, OLIVO-MARIN J C, MEAS-YEDID V. An approach for detection of glomeruli in multisite digital pathology[C]//2016 IEEE 13th International Symposium on Biomedical Imaging (ISBI). Prague, Czech Republic, 2016: 1033-1036.
|
| [19] |
SINGH B, DAVIS L S. An analysis of scale invariance in object detection-SNIP[J/OL]. 2017: arXiv: 1711.08189v2[2020-07-30]. https://arxiv.org/pdf/1711.08189.pdf.
|
 2021, Vol. 42
2021, Vol. 42


