2. 中南大学数学与统计学院, 长沙 410083;
3. 平安科技智慧医疗影像二队, 上海 200030
2. School of Mathematics and Statistics, Central South University, Changsha 410083, Hunan, China;
3. Ping An Technology Smart Medical Imaging Team 2, Shanghai 200030, China
鲍温病(Bowen's disease)和脂溢性角化病(seborrheic keratosis,SK)是2种常见的皮肤肿瘤,临床上两者均可为暗红色或褐色,好发于中老年人,均来源于表皮,其中鲍温病为恶性病变(又称原位皮肤鳞状细胞癌),SK为良性病变。皮肤肿瘤的诊断通常依据临床表现结合病理检查[1],皮肤病理检查不仅可为临床诊断不明的皮肤肿瘤提供线索,也对许多皮肤肿瘤的确诊、治疗方案选择及预后判断等发挥着决定性作用[2]。传统的病理诊断需要病理科医师在显微镜下对组织切片进行专业分析,耗时耗力,且诊断准确性与病理医师的经验直接相关,存在一定主观性。目前,我国病理科医师的缺口高达9万,而他们的工作量仍在不断增加[3]。
随着数字病理和人工智能(artificial intelligence,AI)技术的兴起和发展,利用机器视觉对图像进行自动识别与分析成为近年国内外学者关注的热点,并在医学领域对组织细胞图像自动分析的研究中取得了重要进展[4-5]。全玻片数字扫描技术(whole slide imaging,WSI)及大量定量分析算法的出现突破了病理学家局限于显微镜观察的传统,其不仅可以对病理切片进行定量分析及远程会诊,还可与AI、大数据及云技术结合开发计算机辅助诊断系统(computer-aided diagnosis,CAD)[6-8],使智能化和云端化成为病理学发展的必然趋势[9]。
目前,病理AI的应用范围主要有细胞核特征的检测和分割、良恶性疾病鉴别、疾病分级、染色分析及早期筛查等[10],已被用于多种肿瘤如乳腺癌、神经胶质瘤、胃肠癌、前列腺癌、肺癌和宫颈癌等的诊断过程[11-13]。Lu和Mandal[14-15]基于WSI和CAD完成表皮区域的自动分割与分析,使CAD进驻皮肤病理领域成为可能。本研究通过AI两阶段诊断框架(patch诊断和slide诊断)对鲍温病和SK的组织病理H-E切片进行综合分析和判断,探讨AI在皮肤肿瘤病理诊断和鉴别诊断中的价值。
1 材料和方法 1.1 肿瘤标注流程病理切片的标准化和数字化为AI的深度学习提供了大数据背景,为保证研究结果客观、准确,在建立切片资源库的过程中,标本的保存及处理、组织切片及染色、切片扫描及图像标注等环节都需要标准化流程[16]。
1.1.1 病理切片收集和扫描本研究选择的组织病理H-E切片来自复旦大学附属华山医院皮肤病理室,切片染色标准、病理诊断明确,鲍温病和SK切片数量均为210张。使用全玻片数字扫描仪对病理切片放大40倍扫描,得到清晰、完整的电子图像。
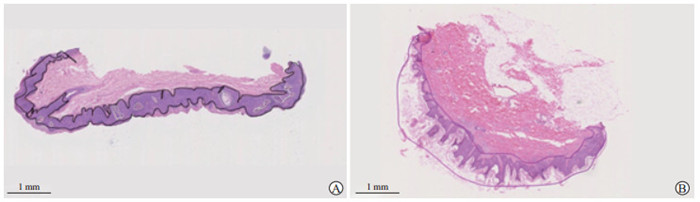
1.1.2 肿瘤区域标注使用NDP View 2软件中的标注工具,由4名病理医师(1名副主任医师审核,1名主治医师辅助指导,2名住院医师执行)对图像进行标注,连续线性标定肿瘤区域,挖空肿瘤区域中的非肿瘤成分。鲍温病、SK和非肿瘤成分的标定线颜色分别选择黑色、紫色和白色(图 1)。
|
图 1 苏木精-伊红染色切片扫描图像中病变区域的人工标注 Fig 1 Manual labeling of lesion areas in scanned images of hematoxylin-eosin stained sections A: Bowen's disease (the area outlined by black line is the diseased tissue); B: Seborrheic keratosis (the area outlined by purple line is the diseased tissue). |
1.1.3 病变病理诊断标准
鲍温病的病理诊断要点是表皮全层细胞排列紊乱,大小不一,有异型,可见核分裂象。SK的病理诊断要点是表皮角化过度,棘层肥厚及乳头瘤样生长,基底样细胞增生,可有数量不等的角囊肿。
1.2 两阶段诊断模型的构建AI的核心是深度学习技术[17],深度学习技术可完全自动地学习抽象的知识表达,将原始数据浓缩成某种知识[18],只要有大规模结构化的数据作为训练集,AI即能从未经处理的数据中发现用于检测或分类的代表性信息。病理图像属于结构化的数据,是AI学习的最佳材料之一。对于图像识别领域,深度卷积神经网络(deep convolutional neural network,DCNN)是深度学习的代表算法[3],其工作流程分为数据预处理-图像分割、特征提取、选择、分类、识别、结果输出[10]。
1.2.1 网络整体框架本实验的诊断框架包括2个部分:第一阶段(patch诊断)的卷积神经网络(convolutional neural network,CNN)特征提取器和第二阶段(slide诊断)的patch特征聚合器。第一阶段将每张病理图片预先切割为多个补丁(patch),得到patch级别的预测结果及训练模型。第二阶段将同一张病理切片的多个patch特征及预测标签序列结合起来,即通过长短时记忆网络、图卷积神经网络(graph convolutional network,GCN)等算法来融合第一阶段patch提取到的特征,并得出模块框架(slide)级别的预测结果,也就是最终的诊断结果。
1.2.2 第一阶段patch诊断由于扫描图像的分辨率为数万级像素,需要很大的感受野,无法直接使用CNN进行训练,本实验采用区域重叠分割法将大小为2 048像素×2 048像素的图片分割成大小为512像素×512像素的小图片,作为深度学习网络的输入图片。此外,跨步设置对于特征学习非常重要,本实验选择步幅为256。数据预处理中将数据集分为2组:训练集(包括验证集)和测试集。详细地说,首先将训练集(包括验证集)随机打乱,然后按验证集的比例大小分为训练集和验证集。本实验中验证集的比例大小取0.1,鲍温病的训练集和验证集图像数量分别为4 356、484张,SK分别为2 700、300张。测试集由病理诊断明确且未经标注病变区域的组织病理H-E切片组成,结果可信度高。本实验测试集中鲍温病图像68张,SK图像127张。
对于训练集和验证集,由于玻片标记了癌变区域的边界,于是选择标记的病变区域来切分patch。在测试中先使用自定义的图像二值化、图像腐蚀、图像膨胀、泛洪算法、连通区域提取和图像滤波等获取组织区域的边界。第一阶段,使用图像分类思路,在slide上做滑动窗口得到的patch上使用CNN提取图像特征,考虑到对于分辨率高的图像需要更大的感受野,应该用更深的网络,同时也需增加网络宽度以获得更细粒度的特征,且网络深度、宽度和分辨率三者不该是独立的,故采用一定的规则统一考虑。因此,本实验对比了5种SOTA(start-of-the-art)技术模型(Xception、Resnet50、InceptionV3、DPN68、Efficientnet_b6[19])在patch模型中的分类效果。
1.2.3 第二阶段slide诊断CNN无法处理非欧几里德结构(non Euclidean structure)的数据,而循环神经网络(recurrent neural network,RNN)可以处理序列数据,但是不适合对空间特征进行提取。在现实中复杂的拓扑结构上的数据场景下有效提取空间特征进行机器学习便成为难点。GCN的提出[20]可有效解决上述问题。本实验使用ConvLSTM描述多个patch之间的序列关系,有效地规避了随机森林、逻辑回归、聚类等算法造成的假阳性率和灵敏度同时降低的问题[21]。采用了强化(augment)GCN方法对多个patch之间的特征进行融合,对于不同的连接节点赋予不同的权重,无需整张图像,鲁棒性强,解决了ConvLSTM的邻近序列的定式设定、不能很好地表达图像的空间相关性、无法并行计算等问题。为了增强鲁棒性,本研究使用金融服务数据模型(financial service data model,FSDM)和半结构化数据模型(semi-structured data model,SSDM)2种模型,通过验证两者在patch特征聚合方面的效能选择更优的模型,并结合统计分析后处理得到最终的slide预测结果。在统计分析时尝试不同的参数后发现,当β0为3时验证集特异度最高。
在FSDM模型中,分别尝试了单位区组(block)数对模型效果的影响、从CNN模型提取的位置对patch分类的影响及576×16×16的特征图,在2 304维特征向量后采用Python和Keras实现Softmax分类。在验证集上,采用特异度作为评估指标,当特异度增大或减小的阈值超过0.000 1时,认为模型效果已更改。批量大小设置为32。损失函数使用分类交叉熵。
在SSDM模型中,尝试使用2 304维特征向量,通过Python和Tensorflow实现Softmax分类。网络由RMSprop优化器训练,RMSprop实验的动量项γ为0.9,学习率的良好建议值为0.001。在验证集上,采用特异度作为评估指标。批量大小设置为12。
1.3 诊断效能的评估利用测试集计算两阶段诊断模型诊断鲍温病和SK的准确度,分别绘制ROC曲线,通过AUC值判断该模型对2种疾病的诊断效能。采用sklearn工具,使用微平均(micro-average)和宏平均(macro-average)2种方式绘制总体ROC曲线,通过AUC值判断该模型对2种疾病的鉴别诊断效能。
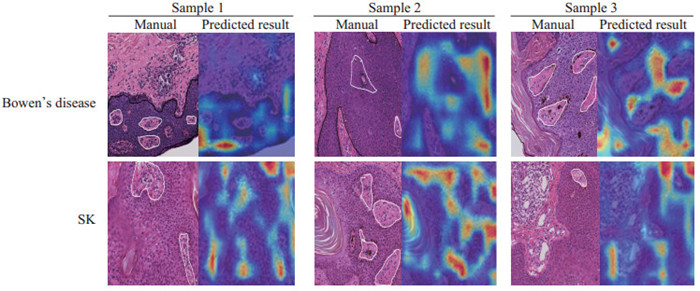
2 结果 2.1 第一阶段patch诊断结果对比了5种SOTA模型在patch模型中的分类效果,结果如表 1所示。Efficientnet_b6模型在patch模型中的分类效果最佳,训练集的灵敏度达到了94.67%(6 680/7 056),验证集的灵敏度达到了95.79%(751/784)。此外,在第一阶段所使用的CNN模型效果较好,并且可以产生准确的病变区域图像,本研究使用梯度加权分类激活映射(gradient-weighted class activation mapping,Grad-CAM)[22]分析对鲍温病和SK有至关重要影响力的关键特征区域(高亮部分),结果如图 2所示,Grad-CAM图显示的病变区域预测结果和人工标注的病变区域在很大程度上是吻合的,表明patch特征提取效果较好。
|
|
表 1 5种start-of-the-art模型在patch模型中的分类效果(灵敏度)比较 Tab 1 Comparison of classification effect (sensitivity) of 5 start-of-the-art models in patch model |
|
图 2 梯度加权分类激活映射分析鲍温病和SK苏木精-伊红染色切片的关键特征区域(200×) Fig 2 Gradient-weighted class activation mapping analysis of saliency feature area in scanned images of hematoxylin-eosin stained sections of Bowen's disease and SK (200×) The areas outlined by black line in manual labeling images are the diseased tissue of Bowen's disease, those outlined by purple line are the diseased tissue of SK, and those outlined by white line are non-tumor tissue. SK: Seborrheic keratosis. |
2.2 第二阶段slide诊断结果
FSDM的训练集特异度为91.16%(6 432/7 056),验证集特异度为82.78%(649/784);SSDM的训练集特异度为95.00%(6 703/7 056),验证集特异度为95.28%(747/784)。可见,SSDM模型在patch特征聚合方面优于FSDM模型。
2.3 两阶段诊断模型的测试结果本研究提出的两阶段诊断模型在鲍温病和SK这2种常见皮肤肿瘤的组织病理H-E切片测试集中,测试准确度分别为92.65%(63/68)和99.21%(126/127)。
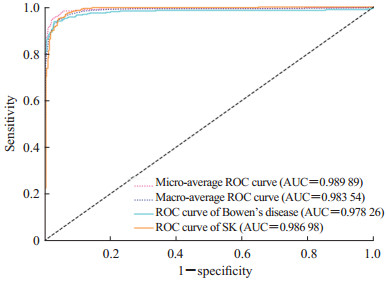
2.4 两阶段诊断模型的诊断效能绘制两阶段诊断模型诊断鲍温病和SK的ROC曲线,得到AUC值分别为0.978 26和0.986 98。采用Sklearn工具,使用微平均、宏平均2种方式绘制总体ROC曲线,AUC值分别为0.989 89、0.983 54,体现出该模型能很好地表达不同类肿瘤之间的区分度。见图 3。
|
图 3 两阶段诊断模型诊断鲍温病和SK的ROC曲线 Fig 3 ROC curves of two-stage diagnostic model for diagnosis of Bowen's disease and SK SK: Seborrheic keratosis; ROC: Receiver operating characteristic; AUC: Area under curve. |
3 讨论
本研究提出的两阶段诊断模型(patch诊断和slide诊断)较有效地实现了鲍温病和SK这2种皮肤肿瘤在病理组织切片中的诊断和鉴别诊断,达到较满意的效果。
对于AI在皮肤肿瘤诊断中的应用,目前绝大多数文献是从皮肤镜入手[19, 23-25],如从皮肤镜角度分析黑色素瘤[26-27],鲜有从组织玻片角度入手的研究。但病理才是皮肤肿瘤诊断的金标准,当临床上皮疹不典型或需要与其他疾病相鉴别时即会通过病理检查明确诊断,本研究的数据来源正是病理组织玻片。
本研究提出的AI对鲍温病和SK的两阶段诊断模型在第一阶段中使用的技术模型Efficientnet_b6优于目前已有的大多数研究结果[28]。本研究中Xception、Resnet50及DPN68模型的训练集灵敏度分别为89.73%、98.47%及97.22%,验证集灵敏度分别为94.77%、91.07%及90.69%;而Efficientnet_b6模型训练集灵敏度达到94.67%,验证集灵敏度达到95.79%,更好地实现了patch的分类。Grad-CAM图显示此方法对病变区域的预测结果与AI标记的病变区域基本一致,说明该模型可以产生较准确的病变区域图像。在第二阶段的patch特征融合中,以往的聚合方法如随机森林、逻辑回归及聚类等虽然降低了假阳性率,但也带来了灵敏度的降低[21]。本研究提出的算法更好地表达了patch之间的空间关系,这在以往的研究中并未提及。两阶段诊断框架的应用进一步提高了鲍温病和SK病理诊断模型的特异度和灵敏度。
AI不仅可以用于病理形态数据的分析,未来还有望将AI整合于免疫组织化学、分子检测数据和临床信息等以获得综合的病理诊断报告,从而为患者提供更多的预后信息和精准的治疗指导。虽然病理AI依然处于研发阶段,准确度未达到100%,但其在一定程度上可以帮助病理医师提高工作效率、减轻工作负担,并且随着临床医师参与度的提升及社会资本的投入,病理AI技术将逐渐填补病理科医师的缺口。
| [1] |
陈琢, 周欣, 张健滔, 毕新岭. 人工智能领域基底细胞癌的诊治研究进展[J]. 第二军医大学学报, 2019, 40: 471-477. CHEN Z, ZHOU X, ZHANG J T, BI X L. Advances in diagnosis and treatment of basal cell carcinoma with artificial intelligence[J]. Acad J Sec Mil Med Univ, 2019, 40: 471-477. |
| [2] |
刘念, 陈宏翔. 人工智能在皮肤科领域的应用与发展[J]. 中华皮肤科杂志, 2019, 52: 63-66. |
| [3] |
郑闪, 孙丰龙, 张慧娟, 史文钊, 马建辉. 人工智能在肿瘤组织病理学的研究现状[J]. 中华肿瘤杂志, 2018, 40: 885-889. DOI:10.3760/cma.j.issn.0253-3766.2018.12.002 |
| [4] |
YU K H, ZHANG C, BERRY G J, ALTMAN R B, RÉ C, RUBIN D L, et al. Predicting non-small cell lung cancer prognosis by fully automated microscopic pathology image features[J/OL]. Nat Commun, 2016, 7: 12474. DOI: 10.1038/ncomms12474.
|
| [5] |
ZHAO X Y, WU X, LI F F, LI Y, HUANG W H, HUANG K, et al. The application of deep learning in the risk grading of skin tumors for patients using clinical images[J/OL]. J Med Syst, 2019, 43: 283. DOI: 10.1007/s10916-019-1414-2.
|
| [6] |
WEBSTER J D, DUNSTAN R W. Whole-slide imaging and automated image analysis: considerations and opportunities in the practice of pathology[J]. Vet Pathol, 2014, 51: 211-223. DOI:10.1177/0300985813503570 |
| [7] |
XU H M, LU C, BERENDT R, JHA N, MANDAL M. Automated analysis and classification of melanocytic tumor on skin whole slide images[J]. Comput Med Imaging Graph, 2018, 66: 124-134. DOI:10.1016/j.compmedimag.2018.01.008 |
| [8] |
张楠, 鲁海珍, 应建明, 马建辉. 人工智能在诊断病理中的应用进展[J]. 诊断病理学杂志, 2019, 26: 183-185. |
| [9] |
步宏, 刘洪红, 包骥, 薛伟. 未来病理: 智能化和云端化是必然的趋势[J]. 中华医学信息导报, 2020, 35: 11. |
| [10] |
于观贞, 魏培莲, 陈颖, 朱明华. 人工智能在肿瘤病理诊断和评估中的应用与思考[J]. 第二军医大学学报, 2017, 38: 1349-1354. YU G Z, WEI P L, CHEN Y, ZHU M H. Artificial intelligence in pathological diagnosis and assessment of human solid tumor: application and thinking[J]. Acad J Sec Mil Med Univ, 2017, 38: 1349-1354. |
| [11] |
HOUSSAMI N, LEE C I, BUIST D S M, TAO D. Artificial intelligence for breast cancer screening: opportunity or hype?[J]. Breast, 2017, 36: 31-33. DOI:10.1016/j.breast.2017.09.003 |
| [12] |
HAJ-HASSAN H, CHADDAD A, HARKOUSS Y, DESROSIERS C, TOEWS M, TANOUGAST C. Classifications of multispectral colorectal cancer tissues using convolution neural network[J/OL]. J Pathol Inform, 2017, 8: 1. DOI: 10.4103/jpi.jpi_47_16.
|
| [13] |
KWAK J T, HEWITT S M. Multiview boosting digital pathology analysis of prostate cancer[J]. Comput Methods Programs Biomed, 2017, 142: 91-99. DOI:10.1016/j.cmpb.2017.02.023 |
| [14] |
LU C, MANDAL M. Automated segmentation and analysis of the epidermis area in skin histopathological images[J]. Annu Int Conf IEEE Eng Med Biol Soc, 2012, 2012: 5355-5359. |
| [15] |
LU C, MANDAL M. Efficient epidermis segmentation for whole slide skin histopathological images[J]. Annu Int Conf IEEE Eng Med Biol Soc, 2014, 2014: 5546-5549. |
| [16] |
ERTOSUN M G, RUBIN D L. Automated grading of gliomas using deep learning in digital pathology images: a modular approach with ensemble of convolutional neural networks[J]. AMIA Annu Symp Proc, 2015, 2015: 1899-1908. |
| [17] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521: 436-444. |
| [18] |
MAMOSHINA P, VIEIRA A, PUTIN E, ZHAVORONKOV A. Applications of deep learning in biomedicine[J]. Mol Pharm, 2016, 13: 1445-1454. |
| [19] |
BISSOTO A, PEREZ F, VALLE E, AVILA S. Skin lesion synthesis with generative adversarial networks[J/OL]. 2019. arXiv: 1902.03253. DOI: 10.1007/978-3-030-01201-4_32.
|
| [20] |
RYU S, LIM J, HONG S H, KIMW Y. Deeply learning molecular structure-property relationships using attention-and gate-augmented graph convolutional network[J/OL]. 2018, arXiv: 1805.10988. (2018-10-08)[2020-12-01]. https://arxiv.org/pdf/1805.10988.pdf.
|
| [21] |
CAMPANELLA G, HANNA M G, GENESLAW L, MIRAFLOR A, WERNECK KRAUSS SILVA V, BUSAM K J, et al. Clinical-grade computational pathology using weakly supervised deep learning on whole slide images[J]. Nat Med, 2019, 25: 1301-1309. |
| [22] |
SELVARAJU R R, COGSWELL M, DAS A, VEDANTAM R, PARIKH D, BATRA D. Grad-CAM: visual explanations from deep networks via gradient-based localization[J]. Int J Comput Vis, 2020, 128: 336-359. |
| [23] |
FAROOQ M A, AZHAR M A M, RAZA R H. Automatic lesion detection system (ALDS) for skin cancer classification using SVM and neural classifiers[J/OL]. 2020. arXiv: 2003.06276. DOI: 10.1109/BIBE.2016.53.
|
| [24] |
KINYANJUI N M, ODONGA T, CINTAS C, CODELLA N C F, PANDA R, SATTIGERI P, et al. Estimating skin tone and effects on classification performance in dermatology datasets[J/OL]. 2019. arXiv: 1910.13268. (2019-10-29)[2020-12-01]. https://arxiv.org/pdf/1910.13268.pdf.
|
| [25] |
NUNNARI F, SONNTAG D. A CNN toolbox for skin cancer classification[J/OL]. 2019. arXiv: 1908.08187. (2019-08-21)[2020-12-01]. https://arxiv.org/pdf/1908.08187.pdf.
|
| [26] |
MENEGOLA A, FORNACIALI M, PIRES R, BITTENCOURT F V, AVILA S, VALLE E. Knowledge transfer for melanoma screening with deep learning[C]//2017 IEEE 14th International Symposium on Biomedical Imaging (ISBI 2017). 2017, Melbourne, VIC, Australia. IEEE, 2017: 297-300.
|
| [27] |
ZUNAIR H, BEN HAMZA A. Melanoma detection using adversarial training and deep transfer learning[J/OL]. Phys Med Biol, 2020, 65: 135005. DOI: 10.1088/1361-6560/ab86d3.
|
| [28] |
TAN M, LE Q V. EfficientNet: rethinking model scaling for convolutional neural networks[J/OL]. 2019. arXiv: 1905.11946. (2020-09-11)[2020-12-01]. https://arxiv.org/pdf/1905.11946.pdf.
|
 2021, Vol. 42
2021, Vol. 42


