2. 海军军医大学(第二军医大学)长海医院心血管外科,上海 200433
2. Department of Cardiovascular Surgery, Changhai Hospital, Naval Medical University (Second Military Medical University), Shanghai 200433, China
肿瘤蛋白p63基因定位于人染色体3q27~29区域,与抑癌基因p53属于同一基因家族,对于肿瘤细胞的增殖、迁移、分化及侵袭等均具有重要的调控作用[1]。研究显示,p63基因的蛋白产物主要包括2类异构体,即带有N端转录活性域的肿瘤蛋白p63(tumor protein 63 with transactivation domain,TAp63)和N端转录活性域缺失的肿瘤蛋白p63(tumor protein 63 with truncated transactivation domain,ΔNp63)[1]。临床病理分析结果发现,ΔNp63在多种不同类型的肿瘤组织中表达异常,并且与肿瘤的发生、发展相关。在包括食管鳞状细胞癌在内的多种鳞状上皮细胞起源的恶性肿瘤组织中ΔNp63的表达水平高于对应的癌旁组织[2-3]。Kaymak等[4]研究发现,野生型p53可通过抑制甲羟戊酸途径促进肿瘤凋亡;Freed-Pastor等[5]研究发现,突变型p53可通过上调甲羟戊酸途径的代谢水平促进乳腺癌的发生、发展,但具体分子机制不明确。3-羟-3-甲戊二酸单酰辅酶A还原酶(3-hydroxy-3-methylglutaryl-coenzyme A reductase,HMGCR)是细胞内甲羟戊酸途径的关键限速酶[6],其转录启动子区域存在ΔNp63的潜在结合位点,推测ΔNp63可能通过调控HMGCR表达的转录途径从而调控食管鳞状细胞癌甲羟戊酸途径的代谢水平。本研究拟揭示ΔNp63调控HMGCR表达的分子机制,分析ΔNp63通过直接调控HMGCR表达对食管鳞状细胞癌细胞增殖、迁移及凋亡的影响。
1 材料和方法 1.1 材料人食管鳞状细胞癌细胞株ECA109购自中国科学院上海生命科学研究院细胞资源中心。DMEM、FBS试剂及Lipofectamine 2000均购自美国Invitrogen公司。RNA抽提、PrimeScript反转录及SYBR Premix Ex Taq定量PCR试剂盒均购自日本TaKaRa公司。BCA蛋白定量试剂盒购自武汉博士德生物工程有限公司。LightCycle 96定量PCR仪购自美国Roche公司。分别表达ΔNp63(LV.ΔNp63)及特异性针对HMGCR的短发夹RNA(short hairpin RNA,shRNA)片段(LV.HMGCRshRNA)的慢病毒载体由和元生物技术(上海)股份有限公司构建。特异针对HMGCR的定量PCR引物(上游引物序列5'-TGA-TTGACCTTTCCAGAGCAAG-3',下游引物序列5'-CTAAAATTGCCATTCCACGAGC-3'),检测免疫共沉淀后DNA片段含量的PCR引物(HMGCR启动子-1~-324 bp片段上游引物序列5'-TTCACATAATGAGCATTTC-3',下游引物序列5'-GGACAGGAATTGTATTTCAC-3';HMGCR启动子-259~-597 bp片段上游引物序列5'-GTGTTT-TCTTCGTAATGTG-3',下游引物序列5'-CTGTGC-GAACCTTACAGTTC-3';HMGCR启动子-562~-891 bp片段上游引物序列5'-CCAGTTACTCCTG-GCTGAAC-3',下游引物序列5'-TGAAATGTGCCT-CTCCTGCG-3';HMGCR启动子-848~-1 172 bp片段上游引物序列5'-AGAGCAGAAGGAAGAAC-GCACAG-3',下游引物序列5'-GTGATAGGAAGC-ATCGG-3';HMGCR启动子-1 118~-1 438 bp片段上游引物序列5'-AGTCATCGACCTGACCC-TGC-3',下游引物序列5'-CTACGTCACGAACG-GTCGC-3'),以及野生型及突变型HMGCR启动子序列均由生工生物工程(上海)股份有限公司合成。萤光素酶报告检测载体pGL4.2及内参载体pRL-CMV均购自美国Promega公司。小鼠抗人HMGCR单克隆抗体及HRP标记的山羊抗小鼠二抗均购自美国Santa Cruz公司。
1.2 细胞培养及慢病毒感染ECA109细胞使用DMEM(含10% FBS)于5% CO2、37 ℃条件下培养至对数生长期用于细胞转染实验。96孔培养板中ECA109细胞培养至约80%融合时,按感染复数为100加入重组慢病毒感染细胞。细胞感染LV.ΔNp63或共感染LV.ΔNp63+LV.HMGCRshRNA为实验组,感染空病毒LV.Null作为对照。病毒感染24 h可进行下一步实验。
1.3 定量PCR检测HMGCR mRNA表达采用TRIzol试剂抽提各组细胞总RNA,取1 μg总RNA进行反转录。使用定量PCR试剂盒定量检测细胞内HMGCR mRNA的表达水平。定量PCR反应体系:SYBR Premix Ex Taq试剂10 μL,Rox Reference Dye 0.4 μL,10 μmol/L上下游引物各0.4 μL,反转录产物0.5 μL,补充去离子水8.3 μL至20 μL。PCR反应条件:95 ℃变性60 s;95 ℃ 5 s,60 ℃ 20 s,共40个循环。以18S rRNA反转录获得的cDNA作为内参,通过ΔΔCt法分析目的基因的相对表达量。
1.4 蛋白质印迹法检测HMGCR蛋白表达采用常规胰酶消化法收集各组细胞,加入自制细胞裂解液(50 mmol/L pH 8.0 Tris,5 mmol/L EDTA,150 mmol/L NaCl,0.5% 非离子去污剂NP-40,10 mmol/L PMSF,1 mmol/L二硫苏糖醇,1 mg/mL抑肽酶),提取细胞总蛋白质,采用BCA法检测总蛋白质浓度。各样本取20 μg总蛋白质进行12% SDS-PAGE,采用半干法将凝胶中蛋白质转印至PVDF膜,再以5%脱脂牛奶常温封闭2 h,加入TBST稀释相应HMGCR抗体4 ℃过夜。取出后用TBST洗膜3次,加入二抗室温孵育2 h,然后用TBST洗膜3次,使用增强型化学发光液显影。采用图像分析软件ImageJ 1.52a进行条带光密度扫描分析,以β肌动蛋白为内参,计算HMGCR的相对表达量。
1.5 染色质免疫沉淀(chromatin immunoprecipitation,ChIP)检测ΔNp63结合HMGCR启动子位置采用常规胰酶消化法收集1×107个ECA109细胞,用1%甲醛交联细胞内的DNA结合蛋白与DNA,于4 ℃进行超声裂解,获得大小为200~1 000 bp的DNA片段。用特异性针对ΔNp63的ChIP级抗体结合蛋白-DNA复合物,通过免疫沉淀法分离出复合物并纯化获得DNA。采用PCR检测捕获的DNA中HMGCR启动子序列不同区域片段的丰度。
1.6 萤光素酶报告实验采用PCR扩增人基因组中HMGCR第一外显子区域前1 500 bp片段启动子(野生型)序列,-728~-747 bp片段区域原始序列为5'-TGGGAAGTGCATGCGTGTAA-3'。人工合成突变启动子序列将-728~-747 bp片段区域改为5'-GTTTCCTGTACGTATGTGCC-3',其余序列维持不变。将野生型及突变型启动子序列分别克隆入萤光素酶报告检测载体pGL4.2。接种感染LV.ΔNp63的ECA109细胞于24孔板,共转染重组萤光素酶报告检测载体及内参载体pRL-CMV。转染48 h后裂解细胞,吸取上清10 μL于萤光素酶检测用96孔板,加入50 μL萤火虫/海参萤光素酶缓冲液,用酶标仪检测发光活性,根据萤火虫萤光素酶荧光强度/海参萤光素酶荧光强度的比值得到各组细胞裂解液中萤光素酶相对活性。
1.7 CCK-8实验采用常规胰酶消化法收集细胞,在96孔板中接种细胞悬液(100 μL每孔,含1 000个细胞)培养至24、48、72 h进行CCK-8实验。检测时向每孔加入10 μL CCK-8试剂,将培养板在培养箱内孵育1 h,用酶标仪测定波长450 nm处的光密度(D)值。
1.8 细胞凋亡检测实验按膜联蛋白Ⅴ-FITC/PI染色检测试剂盒说明书操作。采用胰酶消化后收集ECA109细胞,PBS洗涤3次,重悬于500 μL缓冲液A。加入膜联蛋白Ⅴ-FITC和PI,混匀后室温避光放置15 min,在反应管中加入400缓冲液A,行流式细胞术检测。
1.9 Transwell检测实验制备待测细胞单细胞悬液,取1×104个ECA109细胞重悬于200 μL无血清DMEM,加入Transwell上室,下室中加入500 μL含10% FBS的DMEM,于37 ℃、5% CO2条件下孵育24 h。取出小室,用PBS清洗3次,加入4%多聚甲醛溶液固定30 min后,用棉棒轻柔擦除上室中残留细胞。用0.1%结晶紫染液染15 min,再用双蒸水漂洗3次后室温干燥。于光学显微镜下对下室细胞进行计数及拍照,并计算细胞迁移率:细胞迁移率(%)=(下室细胞数/细胞总数)×100%。
1.10 统计学处理数据采用SPSS 15.0软件进行分析。服从正态分布的计量资料用x±s表示,两组间比较采用独立样本t检验;多组间比较采用单因素方差分析,多重比较采用Tukey法。检验水准(α)为0.05。
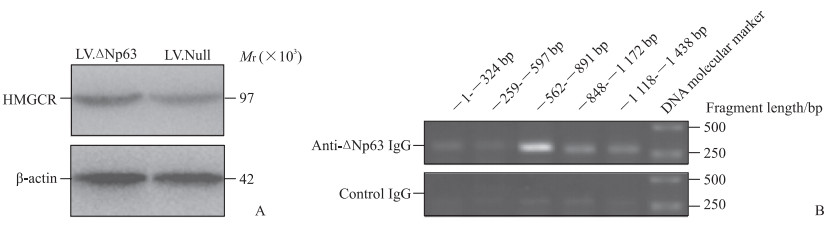
2 结果 2.1 ECA109细胞中ΔNp63转录上调HMGCR表达定量PCR结果显示,LV.ΔNp63感染后ECA109细胞中HMGCR mRNA相对表达水平(3.433±0.233)高于LV.Null感染组(1.006±0.037,P < 0.01)。蛋白质印迹法检测结果显示,LV.ΔNp63感染组ECA109细胞中HMGCR蛋白相对表达水平高于LV.Null感染组(0.29±0.03 vs 0.16±0.02,P < 0.01,图 1A)。定量PCR检测结果显示,HMGCR启动子-562~-891 bp片段经ChIP捕获后富集程度高于其他各区域片段(图 1B),提示ΔNp63蛋白结合于HMGCR启动子-562~-891 bp区域。萤光素酶报告实验结果显示携带-728~-747 bp区域突变的HMGCR启动子相对活性较携带野生型启动子下降[(19.9±2.1)% vs(104.6±3.2)%,P < 0.01]。上述结果提示,ECA109细胞中ΔNp63可促进HMGCR基因转录和HMGCR蛋白表达。
|
图 1 ECA109细胞中ΔNp63对HMGCR表达的影响及转录调控位点分析 Fig 1 Effect of ΔNp63 on expression of HMGCR and the transcriptional regulation site analysis in ECA109 cells A: The protein expression of HMGCR detected by Western blotting in ECA109 cells infected with LV.ΔNp63 or LV.Null; B: The abundance of DNA fragment in HMGCR gene 5'-flanking region recognized by ΔNp63 after chromatin immunoprecipitation. ΔNp63: Tumor protein 63 with truncated transactivation domain; HMGCR: 3-hydroxy-3-methylglutaryl-coenzyme A reductase; LV.ΔNp63: ΔNp63 overexpression lentivirus; LV.Null: Empty lentivirus; IgG: Immunoglobulin G. |
2.2 过表达ΔNp63同时抑制HMGCR表达能够下调ECA109细胞增殖
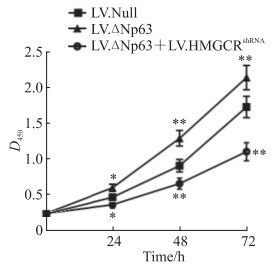
CCK-8检测结果(图 2)显示,LV.ΔNp63感染组ECA109细胞的增殖活性高于LV.Null感染组(P < 0.05),而LV.ΔNp63+LV.HMGCRshRNA共感染组ECA109细胞的增殖活性较LV.Null感染组下降(P < 0.05),提示抑制HMGCR表达能逆转ΔNp63对ECA109细胞增殖的促进作用。
|
图 2 CCK-8检测过表达ΔNp63同时抑制HMGCR表达对ECA109细胞增殖的调控作用 Fig 2 Regulatory effect of ΔNp63 overexpression and HMGCR inhibition on proliferation of ECA109 cells detected by CCK-8 *P < 0.05, **P < 0.01 vs LV.Null group. n=4, x±s. CCK-8: Cell counting kit 8; ΔNp63: Tumor protein 63 with truncated transactivation domain; HMGCR: 3-hydroxy-3-methylglutaryl-coenzyme A reductase; LV.Null: Empty lentivirus; LV.ΔNp63: ΔNp63 overexpression lentivirus; shRNA: Short hairpin RNA. |
2.3 过表达ΔNp63同时抑制HMGCR表达能够上调ECA109细胞凋亡率
流式细胞术检测结果(图 3)显示,LV.ΔNp63+LV.HMGCRshRNA共感染组、LV.ΔNp63感染组及LV.Null感染组的细胞凋亡率分别为(26.9±1.9)%、(16.6±1.5)%及(19.8±1.6)%,LV.ΔNp63+LV.HMGCRshRNA共感染组高于LV.Null感染组,而LV.ΔNp63感染组低于LV.Null感染组(P均 < 0.01),提示抑制HMGCR表达能逆转ΔNp63对ECA109细胞凋亡的抑制作用。

|
图 3 流式细胞术检测过表达ΔNp63同时抑制HMGCR表达对ECA109细胞凋亡的影响 Fig 3 Effect of ΔNp63 overexpression and HMGCR inhibition on apoptosis of ECA109 cells detected by flow cytometry ΔNp63: Tumor protein 63 with truncated transactivation domain; HMGCR: 3-hydroxy-3-methylglutaryl-coenzyme of A reductase; LV.Null: Empty lentivirus; LV.ΔNp63: ΔNp63 overexpression lentivirus; shRNA: Short hairpin RNA. |
2.4 过表达ΔNp63同时抑制HMGCR表达能够抑制ECA109细胞迁移率
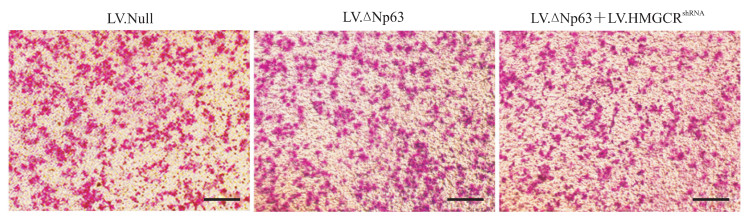
Transwell实验检测结果(图 4)显示,LV.ΔNp63感染组细胞迁移率高于LV.Null感染组[(52.2±2.6)% vs(40.9±3.0)%,P < 0.01],而LV.ΔNp63+LV.HMGCRshRNA共感染组则低于LV.Null感染组[(33.4±3.1)% vs(40.9±3.0)%,P < 0.01],提示抑制HMGCR表达能逆转ΔNp63对ECA109细胞迁移的促进作用。
|
图 4 Transwell实验检测过表达ΔNp63同时抑制HMGCR表达对ECA109细胞迁移的影响 Fig 4 Effect of ΔNp63 overexpression and HMGCR inhibition on cell migration of ECA109 cells detected by Transwell assay Scale bar=100 μm. ΔNp63: Tumor protein 63 with truncated transactivation domain; HMGCR: 3-hydroxy-3-methylglutaryl-coenzyme A reductase; LV.Null: Empty lentivirus; LV.ΔNp63: ΔNp63 overexpression lentivirus; shRNA: Short hairpin RNA. |
3 讨论
p53家族成员通过调控共有或者独特的靶基因表达,在肿瘤细胞的增殖、迁移及分化等过程中发挥重要的调节作用[7-9]。近年来有学者尝试通过调控该家族成员或其下游靶点的表达及活性开展肿瘤生物治疗的探索,取得了长足的进展[10]。由于ΔNp63被发现的时间较晚,在多个方面的研究上处于相对滞后的状态[1]。
有研究显示,ΔNp63在膀胱癌、卵巢癌等癌组织中的表达水平较在相对应的癌旁组织中表达水平降低[11-12];相反,在多种鳞状上皮细胞起源的恶性肿瘤中ΔNp63的表达水平较相对应的癌旁组织高[2]。前期结果证实,在食管鳞状细胞癌细胞中ΔNp63表达升高,体内外实验证实ΔNp63具有促进食管鳞状细胞癌细胞增殖、成瘤的作用[13]。鉴于ΔNp63在不同肿瘤组织中表达变化趋势不一致,其对肿瘤细胞增殖潜能的调控作用可能是通过不同的信号通路实现的,因此本实验进一步探索食管鳞状细胞癌细胞中ΔNp63调控细胞生物学活性的可能机制。本研究发现过表达ΔNp63能够上调食管鳞状细胞癌细胞中HMGCR的mRNA及蛋白质表达,提示ΔNp63可能在转录水平调控HMGCR表达。ChIP及萤光素酶报告实验进一步明确了ΔNp63在HMGCR基因启动子区域的结合位点,在使HMGCR启动子区域-728~-747 bp位置序列突变后过表达ΔNp63不能正常转录启动子下游序列,证实ΔNp63蛋白在食管鳞状细胞癌细胞中具有转录因子活性,并直接增强了HMGCR基因的转录表达。
本实验发现,抑制HMGCR表达能够逆转过表达ΔNp63对食管鳞状细胞癌细胞增殖、迁移的促进作用及对凋亡的抑制作用,提示ΔNp63通过调控甲羟戊酸途径限速酶HMGCR的表达影响甲羟戊酸代谢,进而影响食管鳞状细胞癌细胞的生物学活性。临床研究结果显示,他汀类药物可能通过抑制甲羟戊酸途径抑制肿瘤的发展进程[14-16],但是高剂量服用这类药物可能导致横纹肌溶解等不良反应[17]。探索调控细胞内甲羟戊酸途径的分子机制并获得新的基因干预靶点,有望在提高抑制甲羟戊酸途径效果的同时避免药物带来的不良反应。本研究明确了ΔNp63通过直接转录上调HMGCR表达促进食管鳞状细胞癌细胞增殖、迁移活性的分子机制,为基于甲羟戊酸代谢途径开展的食管鳞状细胞癌基因治疗提供了潜在靶点。
| [1] |
FISHER M L, BALINTH S, MILLS A A. p63-related signaling at a glance[J/OL]. J Cell Sci, 2020, 133: jcs. 228015. DOI: 10.1242/jcs.228015.
|
| [2] |
HSIEH M H, CHOE J H, GADHVI J, KIM Y J, ARGUEZ M A, PALMER M, et al. p63 and SOX2 dictate glucose reliance and metabolic vulnerabilities in squamous cell carcinomas[J/OL]. Cell Rep, 2019, 28: 1860-1878. e9. DOI: 10.1016/j.celrep.2019.07.027.
|
| [3] |
ROBINSON D J, PATEL A, PURDIE K J, WANG J, RIZVI H, HUFBAUER M, et al. Epigenetic regulation of iASPP-p63 feedback loop in cutaneous squamous cell carcinoma[J/OL]. J Invest Dermatol, 2019, 139: 1658-1671. e8. DOI: 10.1016/j.jid.2019.01.020.
|
| [4] |
KAYMAK I, MAIER C R, SCHMITZ W, CAMPBELL A D, DANKWORTH B, ADE C P, et al. Mevalonate pathway provides ubiquinone to maintain pyrimidine synthesis and survival in p53-deficient cancer cells exposed to metabolic stress[J]. Cancer Res, 2020, 80: 189-203. DOI:10.1158/0008-5472.CAN-19-0650 |
| [5] |
FREED-PASTOR W A, MIZUNO H, ZHAO X, LANGERØD A, MOON S H, RODRIGUEZ-BARRUECO R, et al. Mutant p53 disrupts mammary tissue architecture via the mevalonate pathway[J]. Cell, 2012, 148(1/2): 244-258. |
| [6] |
COATES H W, BROWN A J. A wolf in sheep's clothing: unmasking the lanosterol-induced degradation of HMG-CoA reductase[J]. J Lipid Res, 2019, 60: 1643-1645. DOI:10.1194/jlr.C119000358 |
| [7] |
MCDADE S S, PATEL D, MORAN M, CAMPBELL J, FENWICK K, KOZAREWA I, et al. Genome-wide characterization reveals complex interplay between TP53 and TP63 in response to genotoxic stress[J]. Nucleic Acids Res, 2014, 42: 6270-6285. DOI:10.1093/nar/gku299 |
| [8] |
CHEN Y, PENG Y, FAN S, LI Y, XIAO Z X, LI C. A double dealing tale of p63: an oncogene or a tumor suppressor[J]. Cell Mol Life Sci, 2018, 75: 965-973. DOI:10.1007/s00018-017-2666-y |
| [9] |
MOSES M A, GEORGE A L, SAKAKIBARA N, MAHMOOD K, PONNAMPERUMA R M, KING K E, et al. Molecular mechanisms of p63-mediated squamous cancer pathogenesis[J/OL]. Int J Mol Sci, 2019, 20: 3590. DOI: 10.3390/ijms20143590.
|
| [10] |
BYKOV V J N, ERIKSSON S E, BIANCHI J, WIMAN K G. Targeting mutant p53 for efficient cancer therapy[J]. Nat Rev Cancer, 2018, 18: 89-102. DOI:10.1038/nrc.2017.109 |
| [11] |
HE Y, WU X, TANG W, TIAN D, LUO C, YIN Z, et al. Impaired delta Np63 expression is associated with poor tumor development in transitional cell carcinoma of the bladder[J]. J Korean Med Sci, 2008, 23: 825-832. DOI:10.3346/jkms.2008.23.5.825 |
| [12] |
CHOW S E, CHANG Y L, CHUANG S F, WANG J S. Wogonin induced apoptosis in human nasopharyngeal carcinoma cells by targeting GSK-3β and ΔNp63[J]. Cancer Chemother Pharmacol, 2011, 68: 835-845. DOI:10.1007/s00280-010-1552-1 |
| [13] |
YUAN Y, ZENG Z Y, LIU X H, GONG D J, TAO J, CHENG H Z, et al. MicroRNA-203 inhibits cell proliferation by repressing ΔNp63 expression in human esophageal squamous cell carcinoma[J/OL]. BMC cancer, 2011, 11: 57. DOI: 10.1186/1471-2407-11-57.
|
| [14] |
MANTHRAVADI S, SHRESTHA A, MADHUSUDHANA S. Impact of statin use on cancer recurrence and mortality in breast cancer: a systematic review and meta-analysis[J]. Int J Cancer, 2016, 139: 1281-1288. DOI:10.1002/ijc.30185 |
| [15] |
NAYAN M, PUNJANI N, JUURLINK D N, FINELLI A, AUSTIN P C, KULKARNI G S, et al. Statin use and kidney cancer survival outcomes: a systematic review and meta-analysis[J]. Cancer Treat Rev, 2017, 52: 105-116. DOI:10.1016/j.ctrv.2016.11.009 |
| [16] |
GACHPAZAN M, KASHANI H, KHAZAEI M, HASSANIAN S M, REZAYI M, ASGHARZADEH F, et al. The impact of statin therapy on the survival of patients with gastrointestinal cancer[J]. Curr Drug Targets, 2019, 20: 738-747. |
| [17] |
ITO M K, MAKI K C, BRINTON E A, COHEN J D, JACOBSON T A. Muscle symptoms in statin users, associations with cytochrome P450, and membrane transporter inhibitor use: a subanalysis of the USAGE study[J]. J Clin Lipidol, 2014, 8: 69-76. DOI:10.1016/j.jacl.2013.10.006 |
 2021, Vol. 42
2021, Vol. 42


