病理学是一门研究疾病的病因、发病机制及形态结构、功能、代谢等病理改变,揭示疾病的发生、发展规律和阐明疾病本质,辅助医师诊断疾病和预测疾病进展的医学学科。近年来,病理切片数字化技术结合人工智能技术为病理学科打开了一扇新的大门。数字病理学指使用病理切片扫描仪扫描整张病理切片,再用计算机方法对数字化的整张切片进行分析,用于疾病诊断和指导疾病治疗[1]。Mukhopadhyay等[2]对数字病理学和常规显微镜之间的诊断性能进行了大规模多中心比较,研究中纳入了1 992例患者不同肿瘤类型的标本和16名外科病理学家分析报告,结果显示基于数字化全切片图像(whole slide image,WSI)的主要诊断性能不逊于传统的基于显微镜的方法(与参考标准相比,WSI的不一致率为4.9%,显微镜为4.6%)。病理切片数字化催生了建设规模性数字病理库的可能,其中最著名的就是癌症基因组图谱(The Cancer Genome Atlas,TCGA)[3],世界各地的研究人员能够自由获取具有标注信息的病理图像及对应的临床、预后和基因组学信息,这激发了人们将人工智能在数字病理和肿瘤研究中应用的热情。
人工智能是研究、开发用于模拟、延伸和扩展人的智能的理论、方法、技术及应用系统的一门新的技术科学。机器学习主要指把数据输入机器,机器从中"学习"做出预测,是人工智能的大范畴。深度学习是随着人工神经网络的发展而发展起来的一种特殊的机器学习方法。深度学习网络通常包括多层人工神经网络,并倾向于包含1个输入层、1个输出层和多个隐藏层,通过训练足够数量的实例,可以用于识别感兴趣区域[4]。随着计算能力的增长,人工智能扩展了数字病理学的范畴,由最初的数字化过程进化成为数字化图像检测、分割、诊断和分析方法。目前,人工智能已用于探索基于病理图像的细胞学初筛、定量分析、组织病理诊断和预后判断等方面[5]。
1 基于数字病理的人工智能发展规律肿瘤患者的病情管理是一个复杂、长期的过程,包括早期检测、肿瘤诊断、测定肿瘤边缘、辅助治疗、跟踪肿瘤演变、预测肿瘤复发和转移模式及评估患者转归等阶段。病理是肿瘤诊断的金标准,是肿瘤治疗和预后评估的重要参考,其参与了癌症管理的全过程(图 1)。因此,病理人工智能的发展必然推动肿瘤学的进步。现阶段,数字病理人工智能包含了初级任务目标(图像识别、检测和分割等)及高级任务目标(根据图像特征进行诊断、预测疗效和预后)。随着病理人工智能的发展,人们已开始关注如何利用数字病理人工智能帮助解决临床肿瘤学家面临的问题,譬如开发预测预后模型评估疾病的严重程度和转归及预测疾病对某种药物的反应等。
基于病理在癌症管理中的重要性及医学自身的发展规律,把基于数字病理图像识别的人工智能分为3个阶段:第1阶段即诊断和辅助诊断,明确诊断是肿瘤诊治的最核心环节,也是目前病理人工智能期待解决的热点和难点问题;第2阶段即精准医疗,肿瘤确诊之后,治疗方案的选择是重点,基于人工智能提取的病理特征有助于指导治疗方案的选择[6];第3阶段即生命探索,肿瘤自然演变或药物诱导下的进化是肿瘤患者治疗失败的主要原因,病理人工智能有助于探索肿瘤进化史,为肿瘤防治提供建议。
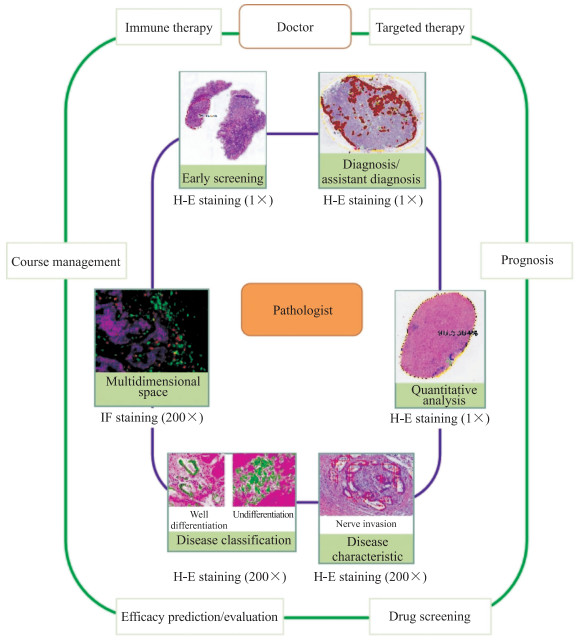
|
图 1 人工智能在肿瘤病理诊断和治疗决策方面的潜在应用 Fig 1 Potential application of artificial intelligence in tumor pathological diagnosis and treatment decision-making H-E: Hematoxylin-eosin; IF: Immumofluorescence. |
2 人工智能与病理诊断
病理诊断和辅助诊断是病理人工智能发展的第1阶段,也是现阶段从事病理图像分析的国内外投资机构、企业和研究机构关注的热点,并且取得了许多突破。近年来病理人工智能的发展有几个里程碑事件。其中奠基性的工作是医学顶级会议——国际生物医学成像研讨会(International Symposium on Biomedical Imaging,ISBI)在2016年举办的Camelyon16竞赛,竞赛的任务是判断测试集的399张淋巴结病理切片是否有癌变(classification)并对癌变的区域精准定位(segmentation),以110个有转移和160个无转移的乳腺癌淋巴结作为训练数据,49张有转移和80张无转移的淋巴结切片作为测试数据,将算法模型的诊断性能与12名病理医师进行比较。结果证明,部分基于深度学习的人工智能算法的诊断性能可媲美病理医师[5]。后续的研究将病理学家的分析与人工智能自动计算诊断方法结合,使诊断乳腺癌前哨淋巴结转移的准确度提高到了99.5%[7]。随后,人们在前列腺癌、乳腺癌、胃癌、结直肠癌和肝癌等多个病种进行了大量病理人工智能研究,也取得了一定的成果。上述研究为人工智能在病理诊断或辅助诊断中的应用推开了一扇窗。
另一项重要研究就是学者们采用弱监督学习而不是人工标注的方法识别肿瘤细胞,也取得了不错的效果。类似于其他医疗影像领域中人工智能研究,人工智能在数字病理中的研发亦需要高质量标注,确保模型的无差别学习。但区别于其他影像,肿瘤异质性决定了肿瘤细胞人工标注的不确定性,如胃部肿瘤的病理类型有10余种,每一种类型都有独特特征[8],因此,胃癌细胞的标注首先要精准识别,然后要标注所有细胞,这对于标注人员的专业性要求非常高,而且工作量巨大。经验丰富的病理医师虽然具有识别各种类型肿瘤细胞的能力,但不可能放弃临床诊疗和科研工作,专门花费精力投入到肿瘤细胞的标注工作中。Campanella等[9]建立了一个无需病理学家人工标注就能识别病理切片中肿瘤细胞的深度学习模型。该研究构建了一个大规模真实世界数据集,共有来自44个国家和地区的15 187例恶性肿瘤患者的病理切片信息,包括24 859张前列腺癌切片、9 962张基底细胞癌切片、9 894张淋巴结转移乳腺癌切片。这些切片无需进一步处理,在存在气泡、形状不规则、固定不好、组织不平整、数字化过程导致的图像模糊等情况下,该模型对肿瘤的诊断仍能达到临床水平,诊断前列腺癌、基底细胞癌和淋巴结转移乳腺癌的AUC值分别为0.991、0.989、0.965。该研究将人工智能在病理诊断或辅助诊断中的应用又推进了一步。
人们对人工智能的期望是借助人工智能技术提高病理诊断效率、简化临床工作流程、提升病理从业人员诊断水平。然而鉴于肿瘤病理的特殊性和复杂性,距这一愿景落地还有较长的一段路要走。具体体现在以下几个方面:第一,要有泛化能力,即某一企业或研究机构研发的产品性能必须在所有医疗机构得到认证,这对于数据来源要求较高,病理切片的质量具有地域性差异,该产品需要学习各个地域各个级别的病理数据,资本投入要求较高。第二,识别肿瘤细胞的性能必须足够高,病理诊断作为肿瘤诊断的金标准,误诊和漏诊对于患者是不可接受的。第三,必须要识别出大部分甚至全部的肿瘤细胞,其目的是提取肿瘤内部特征,强化临床决策,目前的弱监督学习完不成该任务。
3 人工智能和预后评估及治疗方案预测病理诊断是肿瘤治疗决策的必要条件,可以展示疾病进展的多样化形式,能够反映患者的个体状况、接受治疗的能力、对治疗的反应及临床转归等。人工智能能够可视化和量化病理切片中的某些特征,为临床决策提供深度支持。现阶段,肿瘤学家在选择治疗手段时,除了根据影像学检查,还需要参考病理学信息。例如,ⅠB期非小细胞肺癌患者是否需要辅助化学治疗存在较大争议,但普遍认为病理上具有高危因素如低分化、淋巴管或血管癌栓等情况时则需要辅助化学治疗,而这些病理特征可以通过人工智能识别和可视化。人工智能对于推动基于病理特征的精准医疗也取得了极大进展,其中研究较多的有肿瘤浸润淋巴细胞(tumor-infiltrating lymphocyte,TIL)、肿瘤间质比(tumor-stroma ratio,TSR)、免疫组织化学分析、微卫星不稳定性和肿瘤患者预后评估等。
3.1 肿瘤淋巴结转移面积与预后评估在肿瘤诊疗中,TNM分期是指导患者治疗和评估其预后的决策性指标。N分期基于淋巴结转移数目,然而,即使同样N分期的胃癌患者,其预后也参差不齐。还有其他因素影响胃癌患者预后,如淋巴结转移比率和淋巴结转移面积。淋巴结转移比率虽然和预后有关,但应用性不强。淋巴结转移面积既往由于人力和算力所限难以计算,而人工智能使精准计算淋巴结转移面积成为可能。本团队对胃癌淋巴结组织切片(近2万个淋巴结)进行识别,提出了肿瘤细胞区域占转移淋巴结区域的比值(tumor/metastatic lymph node,T/MLN)概念,并通过Kaplan-Meier检验证实T/MLN能有效地将N分期分为高风险与低风险组,提高了预测患者预后的准确性[10]。将人工智能用于肿瘤淋巴结转移的识别和精准评估,T/MLN是可行和可靠的,也是易于推广的病理人工智能项目。
3.2 TIL的空间结构与预后评估TIL作为生物标志物,在基础研究和临床研究中被广泛应用[11]。利用机器学习对病理图像进行分类、分割和计算分析,有望实现TIL的可视化、标准化和量化,而可视化的肿瘤组织内TIL不同类型有望预测肿瘤患者的临床转归和免疫治疗效果。Saltz等[12]描述使用卷积神经网络自动检测从TCGA下载的组织切片图像中TIL的空间分布,TIL的空间结构类型与13种不同癌症患者的预后有关。另一项研究通过分析早期非小细胞肺癌中TIL相互之间及TIL与肿瘤细胞核之间的距离计算出TIL簇的空间分布,结果显示其比TIL密度本身更能有效地预测肺癌的复发风险[13]。Yuan[14]提出了一种三阴性乳腺癌WSI中肿瘤细胞间淋巴细胞空间分布的建模和分析方法。利用这个模型,他根据淋巴细胞与肿瘤细胞的空间距离确定了3种不同类型的淋巴细胞特征,肿瘤内淋巴细胞与肿瘤细胞的比例是预测生存时间的独立预后指标,并与细胞毒性T淋巴细胞蛋白4表达水平相关,而细胞毒性T淋巴细胞蛋白4是伊匹木单抗的治疗靶点。
3.3 TSR与预后评估肿瘤间质在肿瘤侵袭和转移中起着重要作用。TSR在预测肿瘤患者预后方面越来越受到重视。一篇对14项有关TSR与实体瘤预后研究共4 238例患者的meta分析发现,低TSR与严重的临床分期、晚期浸润深度、淋巴结转移显著相关,表明TSR可能是实体肿瘤的独立预后因素[15]。人工智能技术能够较为容易地可视化和量化TSR,为TSR的临床应用提供有力武器。
Beck等[16]在一项开创性研究中,从数字化的乳腺癌患者病理全玻片中提取了6 642个乳腺癌上皮和间质的形态学、结构、关系和全局图像特征。这些特征被用来训练一个预后模型,该模型评分与来自2个不同机构的乳腺癌患者的总体生存密切相关,从肿瘤间质提取的特征比从上皮成分提取的特征具有更强的预后价值。而来自诺丁汉城市医院的1 794例乳腺癌患者的H-E染色病理切片评估中,TSR是无复发生存期的独立预后因素[17]。
3.4 微卫星(microsatellite)不稳定与免疫治疗微卫星指基因组中一些短的、重复的DNA序列,微卫星不稳定与DNA错配修复相关,参与多种恶性肿瘤尤其结直肠癌等的发病。研究发现高度微卫星不稳定肿瘤对免疫治疗反应良好[18],美国FDA批准程序性细胞死亡配体1阻断剂帕博立珠单抗治疗高度微卫星不稳定/错配修复缺陷患者。目前检测微卫星不稳定主要通过免疫组织化学分析或基因检测。Kather等[19]利用深度学习技术从肿瘤组织H-E染色切片图像中直接预测胃肠道肿瘤患者的微卫星稳定性状态,从而对其接受免疫治疗的反应性进行预判。研究人员首先在一个小样本数据中集中测试了5种不同的算法,最终名为Resnet18的深度残差学习(deep residual learning)算法脱颖而出,AUC值高于0.99。研究者将经由神经网络识别的肿瘤区域进行分割、修正和微卫星状态标定,然后从TCGA获取了1 053个胃肠道肿瘤样本对应的病理切片数据来训练Resnet18网络,结果显示在胃癌石蜡样本、结直肠癌石蜡样本和结直肠癌冷冻样本中其检测微卫星不稳定的AUC值分别为0.81、0.84和0.77。之后以德国患者为主的结直肠癌DACHS(Darmkrebs: Chancen der Verhütung durchScreening)研究中的数据为验证集对该模型进行评估,得到其检测微卫星不稳定的AUC值高达0.84。尽管该研究存在训练数据量较小、训练样本代表性单一(以白种人患者为主)和网络可解释性不足等缺陷,但所用方法已经在快速、精准筛选肿瘤免疫治疗适用患者上展现了巨大潜力。
3.5 人表皮生长因子受体2(human epidermal growth factor receptor 2,HER-2)与靶向治疗在乳腺癌和胃癌中,HER-2是一种预测疗效和预后的标志物。HER-2的检测主要依靠免疫组织化学和荧光原位杂交,但结果需要人工判读,因此具有主观性。2016年6月诺丁汉PathSoc年度会议举办了基于人工智能的HER-2自动评分方法比赛[20]。比赛结果中人工智能自动分析与组织病理学家的表现相当,许多算法都达到了很高的精度。对于HER-2评分为┼┼┼的病例,所有参与者(无论是人还是计算机)都能正确识别。对于HER-2评分为┼┼的病例,人工智能方法需要进一步微调。该研究结果表明,自动化的免疫组织化学评分算法可以提供形态学特征的定量评估,有助于客观的计算机辅助诊断及疗效和预后的预测建模。此外,研究者们采用机器学习方法结合拉曼光谱对乳腺癌组织进行亚型分析,根据脂质、胶原蛋白和核酸含量相关的生物化学变化,预测Luminal A型、Luminal B型、HER-2过表达型和三阴性乳腺癌的准确度分别达到了70%、100%、90%和96.7%[21]。
3.6 其他治疗方案预测为改善机器学习方法的预后预测性能,一些研究小组正在探索将肿瘤的分子和形态属性结合起来的方法。肿瘤学家还必须应对迅速变化的治疗指南,其中纳入了新的治疗药物,如免疫检查点抑制剂和靶向药物,这些药物中大多数需要自付费用,而且可能没有保险覆盖。在这种情况下,优化的基于人工智能的生物标记方法可以为肿瘤学家提供一种准确且廉价的工具,用于筛选适合接受这些药物治疗的患者,以提高这些新型药物的疗效,避免产生不必要的费用和不必要的系统性不良反应。
肿瘤患者的精准医疗取决于其病理和基因组蕴含的信息,利用人工智能技术有望提取这些信息,从某个点为个体化治疗提供数据支持。
4 人工智能与基因组学TCGA涵盖了33种癌症类型,包括超过2万种恶性肿瘤及相应正常组织的病理特征和分子特征。全基因组泛癌分析(pan-cancer analysis of whole genomes,PCAWG)联盟整合分析了38种肿瘤类型,获得并分析了2 658个肿瘤全基因组,涵盖了肿瘤驱动突变、非编码区域、突变特征、结构变异、肿瘤进化和RNA改变6个方面,并通过对这些突变特征的深入分析挖掘出了不同突变特征与不同分子事件的关系[22]。研究人员利用这些数据重建了突变进程和驱动突变的进化史,早期肿瘤发生与少数驱动突变和拷贝数变异有关,但在随后的发展过程中突变图谱的变化则很大,在晚期阶段驱动基因突变的多样性可达4倍以上,同时基因组不稳定性也增加[23]。肿瘤进化知识图谱为深入了解肿瘤及其治疗方式提供了很大帮助。肿瘤内异质性指肿瘤内亚克隆细胞群的多样性,肿瘤内异质性越低,患者存活率越高。除肿瘤细胞外,肿瘤微环境的复杂性和多样性对肿瘤的发生、发展和免疫治疗也有重要影响。肿瘤微环境包括基质细胞、成纤维细胞、构成肿瘤内血管的内皮细胞、细胞外基质及各类免疫细胞。肿瘤通过分泌生长因子、免疫抑制分子和其他信号分子影响其微环境,后者在恶性肿瘤免疫治疗中的作用也受到广泛重视[24]。结合人工智能手段可以对肿瘤异质性、肿瘤微环境和基因组特征等信息进行更加深入的研究。
基于数字病理的人工智能第3个阶段就是整合生物医学领域和病理形态学,利用计算机程序可视化和量化肿瘤异质性及肿瘤微环境,甚至识别导致肿瘤细胞增殖和迁移的异常基因和信号通路等,助力研究肿瘤进化、筛选新的靶点、开发新的药物,最终开发出精准、有效的个体化治疗方案。Coudray等[25]训练了一个深度卷积神经网络——Inception v3,用来分析从TCGA获得的肺癌病理图像,该模型能分辨出肺腺癌和肺鳞状细胞癌,准确度高达97%。该模型在冷冻组织、甲醛固定石蜡包埋组织和活检组织的独立数据集上进行了验证,均取得了不错的效果。训练该模型预测肺腺癌中最常见的10个突变基因,结果显示丝氨酸/苏氨酸激酶11(serine/threonine kinase 11,stk11)、表皮生长因子受体(epidermal growth factor receptor,EGFR)、脂肪非典型钙黏蛋白1(FAT tumor suppressor homolog 1,FAT1)、SET结合蛋白1(SET binding protein 1,SETBP1)、Kirsten大鼠肉瘤病毒癌基因同源物(Kirsten rat sarcoma viral oncogene homolog,KRAS)和P53 6个基因可以通过病理图像进行预测,其准确度为73%~86%。该结果表明深度学习模型可以帮助病理学家检测恶性肿瘤亚型或基因突变,发现肿瘤细胞及其周围组织可见特征或以前未发现的模式,从而帮助患者更早地进行精准治疗。
人工智能通过病理信息探索肿瘤发生和肿瘤进化有助于推动医疗进步、实现精准医疗。然而如何将病理特征和组学特征相融合值得思考。近年出现的空间转录组能够将病理H-E染色切片与RNA测序相对应,以观察病理切片上某一区域的基因表达情况,这是人工智能-病理-组学交叉融合的一个创新性尝试,有助于通过病理切片获取基因组学信息。
5 困境和解决方案一个成功的基于人工智能的数字病理模型需要病理学家和肿瘤学家通力合作。病理学家为参与设计和研发人工智能算法的算法科学家提供知识储备,指导研发人员掌握病理图像的特定属性,通过对细胞、组织类型、生物结构或感兴趣区域的注释和分割来训练算法。在验证过程中,也需要病理医师提供诊断参考或标准,与机器学习算法进行比较,反馈性促进算法的成熟。肿瘤学家则在应用场景方面提供研究方向及进行算法的临床验证。最终将开始利用病理人工智能探索肿瘤发生和肿瘤进化,研究成果再助益病理医师和肿瘤医师。
人工智能在数字病理中的应用遇到的最大困难是需要多名病理学家进行高质量标注,确保模型可以充分学习各种病变细胞的形态[26]。但是,如前述,由于肿瘤的异质性,准确地识别出肿瘤及识别出所有的肿瘤细胞极其困难。而且,肿瘤的诊断不仅是诊断出是否存在肿瘤细胞,还要判断肿瘤细胞的分化程度、浸润程度、神经侵犯、癌栓、TSR及TIL等肿瘤微环境中的信息[27]。
为了实现数字病理人工智能3个阶段的跨越式发展,我们提出了"镜像拓画"标注理念和初步想法。首先,对一张H-E染色病理切片进行数字化,而后对这张切片进行处理,脱去H-E染色,利用免疫组织化学、免疫荧光、多重免疫组织化学等技术显示肿瘤细胞或要识别的病理特征,如神经侵犯和TIL等。提取这些染色结果,配准至预先数字化的H-E染色病理切片上,这些染色结果就可以作为标记进行后续的人工智能研究。本方法能够根据病理医师、肿瘤医师和科研人员的需求快速地产生高质量和高容量的数据库,助力研发高诊断准确性、预后和疗效预测的人工智能辅助系统,降低病理人工智能研究的入门门槛,扩展病理人工智能的应用范围。
| [1] |
NIAZI M K K, PARWANI A V, GURCAN M N. Digital pathology and artificial intelligence[J/OL]. Lancet Oncol, 2019, 20: e253-e261. DOI: 10.1016/S1470-2045(19)30154-8.
|
| [2] |
MUKHOPADHYAY S, FELDMAN M D, ABELS E, ASHFAQ R, BELTAIFA S, CACCIABEVE N G, et al. Whole slide imaging versus microscopy for primary diagnosis in surgical pathology: a multicenter blinded randomized noninferiority study of 1992 cases (pivotal study)[J]. Am J Surg Pathol, 2018, 42: 39-52. DOI:10.1097/PAS.0000000000000948 |
| [3] |
TOMCZAK K, CZERWIŃSKA P, WIZNEROWICZ M. The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge[J]. Contemp Oncol (Pozn), 2015, 19: A68-A77. |
| [4] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521: 436-444. |
| [5] |
于观贞, 刘西洋, 张彦春, 杨晶东, 田建辉, 朱明华. 人工智能在临床医学中的应用与思考[J]. 第二军医大学学报, 2018, 39: 358-365. YU G Z, LIU X Y, ZHANG Y C, YANG J D, TIAN J H, ZHU M H. Artificial intelligence in clinical medicine: application and thinking[J]. Acad J Sec Mil Med Univ, 2018, 39: 358-365. |
| [6] |
ACS B, RANTALAINEN M, HARTMAN J. Artificial intelligence as the next step towards precision pathology[J]. J Intern Med, 2020, 288: 62-81. DOI:10.1111/joim.13030 |
| [7] |
LIU Y, KOHLBERGER T, NOROUZI M, DAHL G E, SMITH J L, MOHTASHAMIAN A, et al. Artificial intelligence-based breast cancer nodal metastasis detection: insights into the black box for pathologists[J]. Arch Pathol Lab Med, 2019, 143: 859-868. DOI:10.5858/arpa.2018-0147-OA |
| [8] |
国家"863"重大项目"胃癌分子分型与个体化诊疗"课题组. 胃癌病理分型和诊断标准的建议[J]. 中华病理学杂志, 2010, 39: 266-269. DOI:10.3760/cma.j.issn.0529-5807.2010.04.013 |
| [9] |
CAMPANELLA G, HANNA M G, GENESLAW L, MIRAFLOR A, WERNECK KRAUSS SILVA V, BUSAM K J, et al. Clinical-grade computational pathology using weakly supervised deep learning on whole slide images[J]. Nat Med, 2019, 25: 1301-1309. DOI:10.1038/s41591-019-0508-1 |
| [10] |
WANG X, CHEN Y, GAO Y, ZHANG H, GUAN Z, DONG Z, et al. Predicting gastric cancer outcome from resected lymph node histopathology images using deep learning[J/OL]. Nat Commun, 2021, 12: 1637. DOI: 10.1038/s41467-021-21674-7.
|
| [11] |
周超, 施晓倩, 洪涵涵, 尹基忠, 王昱升, 李兵. 基于癌症基因组图谱肺鳞状细胞癌免疫细胞浸润图景及预后分析[J]. 第二军医大学学报, 2021, 42: 391-398. ZHOU C, SHI X Q, HONG H H, YIN J Z, WANG Y S, LI B. The infiltration of immune cells in lung squamous cell carcinoma and its prognostic analysis based on The Cancer Genome Atlas[J]. Acad J Sec Mil Med Univ, 2021, 42: 391-398. |
| [12] |
SALTZ J, GUPTA R, HOU L, KURC T, SINGH P, NGUYEN V, et al. Spatial organization and molecular correlation of tumor-infiltrating lymphocytes using deep learning on pathology images[J/OL]. Cell Rep, 2018, 23: 181-193. e7. DOI: 0.1016/j.celrep.2018.03.086.
|
| [13] |
CORREDOR G, WANG X X, ZHOU Y, LU C, FU P F, SYRIGOS K, et al. Spatial architecture and arrangement of tumor-infiltrating lymphocytes for predicting likelihood of recurrence in early-stage non-small cell lung cancer[J]. Clin Cancer Res, 2019, 25: 1526-1534. DOI:10.1158/1078-0432.CCR-18-2013 |
| [14] |
YUAN Y. Modelling the spatial heterogeneity and molecular correlates of lymphocytic infiltration in triple-negative breast cancer[J/OL]. J R Soc Interface, 2015, 12: 20141153. DOI: 10.1098/rsif.2014.1153.
|
| [15] |
WU J, LIANG C, CHEN M, SU W. Association between tumor-stroma ratio and prognosis in solid tumor patients: a systematic review and meta-analysis[J]. Oncotarget, 2016, 7: 68954-68965. |
| [16] |
BECK A H, SANGOI A R, LEUNG S, MARINELLI R J, NIELSEN T O, VAN DE VIJVER M J, et al. Systematic analysis of breast cancer morphology uncovers stromal features associated with survival[J/OL]. Sci Transl Med, 2011, 3: 108ra113. DOI: 10.1126/scitranslmed.3002564.
|
| [17] |
VANGANGELT K M H, GREEN A R, HEEMSKERK I M F, COHEN D, VAN PELT G W, SOBRAL-LEITE M, et al. The prognostic value of the tumor-stroma ratio is most discriminative in patients with grade Ⅲ or triple-negative breast cancer[J]. Int J Cancer, 2020, 146: 2296-2304. DOI:10.1002/ijc.32857 |
| [18] |
LI K, LUO H, HUANG L, LUO H, ZHU X. Microsatellite instability: a review of what the oncologist should know[J/OL]. Cancer Cell Int, 2020, 20: 16. DOI: 10.1186/s12935-019-1091-8.
|
| [19] |
KATHER J N, PEARSON A T, HALAMA N, JÄGER D, KRAUSE J, LOOSEN S H, et al. Deep learning can predict microsatellite instability directly from histology in gastrointestinal cancer[J]. Nat Med, 2019, 25: 1054-1056. DOI:10.1038/s41591-019-0462-y |
| [20] |
QAISER T, MUKHERJEE A, REDDY PB C, MUNUGOTI S D, TALLAM V, PITKÄAHO T, et al. HER2 challenge contest: a detailed assessment of automated HER2 scoring algorithms in whole slide images of breast cancer tissues[J]. Histopathology, 2018, 72: 227-238. DOI:10.1111/his.13333 |
| [21] |
TALARI A C S, REHMAN S, REHMAN I U. Advancing cancer diagnostics with artificial intelligence and spectroscopy: identifying chemical changes associated with breast cancer[J]. Expert Rev Mol Diagn, 2019, 19: 929-940. |
| [22] |
ALEXANDROV L B, KIM J, HARADHVALA N J, HUANG M N, TIAN NG A W, WU Y, et al. The repertoire of mutational signatures in human cancer[J]. Nature, 2020, 578: 94-101. DOI:10.1038/s41586-020-1943-3 |
| [23] |
GERSTUNG M, JOLLY C, LESHCHINER I, DENTRO S C, GONZALEZ S, ROSEBROCK D, et al. The evolutionary history of 2, 658 cancers[J]. Nature, 2020, 578: 122-128. DOI:10.1038/s41586-019-1907-7 |
| [24] |
JIAO S, SUBUDHI S K, APARICIO A, GE Z, GUAN B, MIURA Y, et al. Differences in tumor microenvironment dictate T helper lineage polarization and response to immune checkpoint therapy[J/OL]. Cell, 2019, 179: 1177-1190. e13. DOI: 10.1016/j.cell.2019.10.029.
|
| [25] |
COUDRAY N, OCAMPO P S, SAKELLAROPOULOS T, NARULA N, SNUDERL M, FENYÖ D, et al. Classification and mutation prediction from non-small cell lung cancer histopathology images using deep learning[J]. Nat Med, 2018, 24: 1559-1567. |
| [26] |
于观贞, 魏培莲, 陈颖, 朱明华. 人工智能在肿瘤病理诊断和评估中的应用与思考[J]. 第二军医大学学报, 2017, 38: 1349-1354. YU G Z, WEI P L, CHEN Y, ZHU M H. Artificial intelligence in pathological diagnosis and assessment of human solid tumor: application and thinking[J]. Acad J Sec Mil Med Univ, 2017, 38: 1349-1354. |
| [27] |
顾小强, 俞文隆, 陈颖, 魏培莲, 董伟, 钱建新, 等. 人工智能在鉴别肝门部胆管癌细胞及周围神经侵袭中的应用[J]. 第二军医大学学报, 2021, 42: 724-730. GU X Q, YU W L, CHEN Y, WEI P L, DONG W, QIAN J X, et al. Application of artificial intelligence in identifying hilar cholangiocarcinoma and perineural invasion[J]. Acad J Sec Mil Med Univ, 2021, 42: 724-730. |
 2021, Vol. 42
2021, Vol. 42


