2. 上海大学生命科学学院, 上海 200444
2. School of Life Sciences, Shanghai University, Shanghai 200444, China
2020年3月11日(当地时间),WHO将新型冠状病毒肺炎(coronavirus disease 2019,COVID-19)列为全球性大流行病,目前全球已有200多个国家和地区先后暴发了COVID-19疫情。截至2020年4月15日,COVID-19已造成全球180多万人感染,死亡11万多例[1]。在COVID-19疫情出现初期,研究人员应用高通量测序技术迅速鉴定出引发此次疫情的病原体为一种新型冠状病毒,它与严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus,SARS-CoV)的基因组序列具有约80%的同源性,随后成功分离出该病毒[2-3]。国际病毒分类委员会将该病毒命名为严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)。研究发现SARS-CoV-2具有很强的传染性,致病性似乎不及SARS-CoV,病毒基因组经多代传播后仍保持了较高的稳定性,变异较慢[4-7]。这些遗传特性暗示SARS-CoV-2与它的祖先病毒相比发生了一些重要的遗传变异,但究竟是哪些病毒蛋白发生了较大变异,变异蛋白又涉及哪些关键的氨基酸残基,目前除了刺突蛋白(spike protein,S蛋白)外,其他蛋白则缺少相关研究报道。
已有研究指出SARS-CoV-2与蝙蝠冠状病毒RaTG13(bat CoV RaTG13)的基因组序列同源性最高(达96.2%)[3],与穿山甲冠状病毒基因组同源性也达到90%[8-9]。与SARS-CoV相似,这些冠状病毒的基因组构成高度保守,编码相似的结构和非结构蛋白(non-structural protein,Nsp),包括开放阅读框(open reading frame,Orf)1ab[编码木瓜样蛋白酶(papain-like protease,PLpro)、3C样蛋白酶(3C-like protease,3CLpro)、RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)、Nsp7、Nsp8、Nsp9、核糖核酸外切酶(exoribonuclease,ExoN)、尿苷酸特异性核糖核酸内切酶(uridylate-specific endoribonuclease,NendoU)和Nsp13等Nsp,主要与病毒RNA合成相关]、S蛋白、Orf3a、包膜蛋白(envelope protein,E蛋白)、膜蛋白(membrane protein,M蛋白)、Orf6、Orf7、Orf8和核衣壳蛋白(nucleocapsid protein,N蛋白)等[10-11]。位于冠状病毒颗粒表面的S蛋白含有受体结合区(receptor-binding domain,RBD),主要负责与靶细胞表面的受体结合及随后的膜融合,是病毒入侵宿主细胞的关键蛋白[12]。研究显示,与SARS-CoV S蛋白相比,SARS-CoV-2 S蛋白的RBD发生了较大变异,包括受体结合位点的氨基酸突变及4个小肽片段插入,但是这些变异并不影响受体结合构象的形成[13]。与bat CoV RaTG13 S蛋白相比,SARS-CoV-2 S蛋白的RBD发生了19个氨基酸残基的替换,但这些氨基酸残基变异的重要程度并不清楚[2]。与某些穿山甲冠状病毒的S蛋白相比,SARS-CoV-2 S蛋白的RBD高度保守,提示SARS-CoV-2 S蛋白的RBD可能来自于某些穿山甲冠状病毒[9, 14]。冷冻电镜研究显示,SARS-CoV-2的S蛋白由于PRRA序列的插入,导致形成一个新的RRAR蛋白酶剪切位点,并在三维结构上形成一个新的结构单元,极大提高了其与受体的结合能力,这也可能是SARS-CoV-2具有较强传染性的原因[15]。
由于多数SARS-CoV编码蛋白的三维结构已被解析,有了较多、较好的研究数据与分子模型,而且SARS-CoV-2中主要蛋白质分子与SARS-CoV编码蛋白具有较高的序列保守性,因此本研究应用蛋白质三维结构重建技术,分析SARS-CoV-2中主要蛋白质分子变异氨基酸残基电荷的相互作用变化,从而确定影响病毒蛋白构象和功能的特定氨基酸残基,以期为更好地理解SARS-CoV-2的遗传特性提供线索,也为后续临床治疗药物的研发提供理论参考。
1 材料和方法 1.1 蛋白质氨基酸序列同源性分析所有的冠状病毒基因组序列均来自GenBank[美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)],Orf1ab包含的功能蛋白应用CD Search(NCBI)进行确认分析,采用NCBI的BLAST、COBALT软件和欧洲生物信息研究所(European Bioinformatics Institute,EBI)的Clustal Omegal软件进行序列同源性分析。本研究使用的基因组序列包括SARS-CoV CV7(登录号:DQ898174)、SARS-CoV-2(登录号:MN908947)和bat CoV RaTG13(登录号:MN996532)。
1.2 蛋白质三维结构重建SARS-CoV蛋白质三维结构模板均来自NCBI的蛋白质数据库(Protein Database,PDB),SARS-CoV-2和bat CoV RaTG13主要蛋白质分子三维结构重建均在SWISS-MODEL服务器上完成。蛋白质三维结构重建的主要参数:GMQE(global model quality estimation)和QMEAN(qualitative model energy analysis)。GMQE为整体模型质量评估值,合理范围为0~1;QMEAN为模型能量值定量分析值,合理值在0附近[16]。
1.3 酸性与碱性氨基酸的电荷相互作用分析应用PyMOL软件确定带电氨基酸残基侧链氨基和羧基的氮氧原子间的距离(10 Å以内),判断蛋白质中突变氨基酸及空间上邻近氨基酸残基的电荷相互作用强弱。异种电荷相互吸引,形成盐键;同种电荷相互排斥,产生斥力。在特定的pH范围内,酸性氨基酸天冬氨酸(D)和谷氨酸(E)带负电,碱性氨基酸赖氨酸(K)和精氨酸(R)带正电,而组氨酸(H)质子化后也带正电。电荷之间的作用力与原子间距离的平方成反比。
2 结果和讨论 2.1 SARS-CoV-2与bat CoV RaTG13中的蛋白质分子氨基酸变异分析以SARS-CoV CV7株编码的主要蛋白作为出发序列,在GenBank数据库中进行同源比对,发现这些蛋白与蝙蝠冠状病毒(bat CoV RaTG13除外)编码蛋白相比变异率为0.2%~11.6%,其中Orf8的变异率最高,达到11.6%。由于目前病毒学界已经确定SARS-CoV来源于中华菊头蝠冠状病毒,以此作为参考,对SARS-CoV-2编码的蛋白作进一步细致的变异率分析。我们发现这些蛋白与SARS-CoV编码蛋白相比,变异率为6.6%~41.3%;而与蝙蝠冠状病毒(bat CoV RaTG13除外)编码的蛋白相比,变异率降低,为1.3%~19.7%。有趣的是,SARS-CoV-2编码的蛋白与bat CoV RaTG13编码蛋白相似度较高,变异率均不超过5.0%。而bat CoV RaTG13编码的蛋白与其他蝙蝠冠状病毒编码蛋白相比,变异率为0.4%~19.8%。见图 1。

|
图 1 SARS-CoV、蝙蝠冠状病毒和SARS-CoV-2中主要蛋白质分子的氨基酸变异分析 Fig 1 Analysis of amino acid variations of major proteins from SARS-CoV, bat CoV and SARS-CoV-2 The numbers are percentage of the amino acid variations. SARS-CoV: Severe acute respiratory syndrome coronavirus; SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; bat CoV: Bat coronavirus; Orf: Open reading frame; S: Spike protein; E: Envelope protein; M: Membrane protein; N: Nucleocapsid protein |
为进一步确认比对结果,我们对SARS-CoV-2和bat CoV RaTG13 Orf1ab编码的蛋白单独进行了氨基酸变异分析,发现这些蛋白与bat CoV RaTG13对应的蛋白相比,变异率不超过10.29%;而且在Orf1ab上存在2个高度变异的区域(Nsp1-Macro,变异率为4.34%~10.29%;Nsp3-NAR,变异率为2.21%~3.08%),其中DUF3655变异率高达10.29%,其余区域的变异率不超过0.72%(图 2)。氨基酸序列比对分析显示SARS-CoV-2编码的蛋白与bat CoV RaTG13编码的蛋白高度同源,这与之前文献报道的关于SARS-CoV-2与bat CoV RaTG13基因组高度同源[3]的观点一致。根据SARS-CoV编码蛋白的氨基酸变异率分析结果,结合现有的冠状病毒基因组数据,我们认为bat CoV RaTG13可能是SARS-CoV-2的祖先病毒。
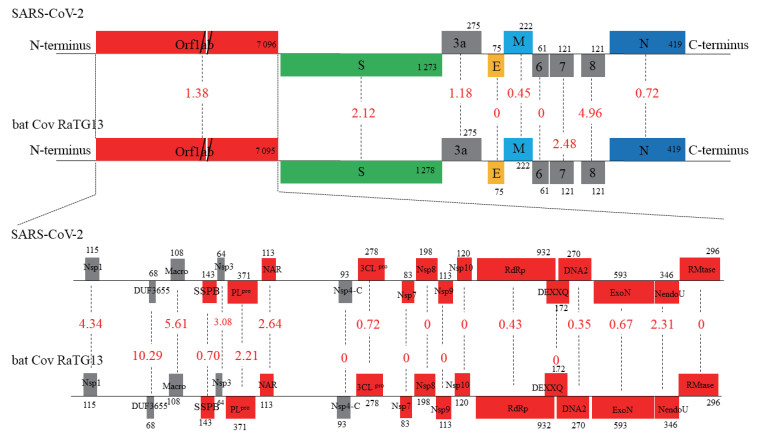
|
图 2 SARS-CoV-2和bat CoV RaTG13中主要蛋白质分子的氨基酸变异图谱 Fig 2 Amino acid variation map of major proteins from SARS-CoV-2 and bat CoV RaTG13 Annotations of predicted proteins are mainly from the server of CD search in National Center for Biotechnology Information (NCBI). The red numbers are percentage of the amino acid variations. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; bat CoV: Bat coronavirus; Orf: Open reading frame; S: Spike protein; E: Envelope protein; M: Membrane protein; N: Nucleocapsid protein; Nsp: Non-structural protein; DUF: Protein of unknown function; SSPB: Single-stranded poly(A)-binding domain; PLpro: Papain-like protease; NAR: Nucleic acid-binding domain; 3CLpro: 3C-like protease; RdRp: RNA-dependent RNA polymerase; DNA2: SuperfamilyⅠ DNA and/or RNA helicase; DEXXQ: DEXXQ-box helicase domain of Upf1-like helicase; ExoN: Exoribonuclease; NendoU: Uridylate-specific endoribonuclease; RMtase: Cap-0 specific (nucleoside-2'-O-)-methyltransferase |
为进一步分析确定编码蛋白中关键的氨基酸变异,我们将SARS-CoV-2和bat CoV RaTG13中主要蛋白质分子的氨基酸变异进行了更为细致的区分。将氨基酸错义突变区分为近似突变和非近似突变两类。所谓近似突变,即突变前后氨基酸的基本性质未变,蛋白质构象也不会发生明显变化,如电性(谷氨酸E-天冬氨酸D、精氨酸R-赖氨酸K)或极性(天冬酰胺N-谷氨酰胺Q,丝氨酸S-苏氨酸T-半胱氨酸C-酪氨酸Y,苯丙氨酸F-异亮氨酸I-亮氨酸L-缬氨酸V-丙氨酸A)相同的氨基酸之间的替换,也包括性质相似氨基酸之间的转换如苯丙氨酸F-酪氨酸Y、苏氨酸T-丙氨酸A的替换。非近似突变即突变后蛋白质构象发生明显的变化,功能出现异常,如特殊性质氨基酸的插入或缺失或不同电性的氨基酸的转换。根据这一界定,重新统计2种病毒中的主要蛋白(共统计28个蛋白)的氨基酸非近似变异率不超过5.88%,其中DUF3655的非近似变异率最高,为5.88%;共有14个蛋白没有发生非近似突变,另有11个蛋白分子的非近似变异率低于1.00%(图 3)。
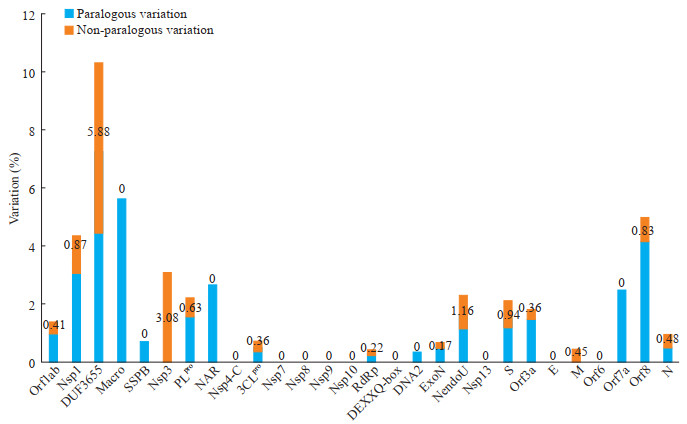
|
图 3 SARS-CoV-2和bat CoV RaTG13中主要蛋白质分子的非近似氨基酸变异分析 Fig 3 Analysis of non-paralogous amino acid variations of major proteins from SARS-CoV-2 and bat CoV RaTG13 The numbers are percentage of the non-paralogous amino acid variations. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; bat CoV: Bat coronavirus; Orf: Open reading frame; Nsp: Non-structural protein; DUF: Protein of unknown function; SSPB: Single-stranded poly(A)-binding domain; PLpro: Papain-like protease; NAR: Nucleic acid-binding domain; 3CLpro: 3C-like protease; RdRp: RNA-dependent RNA polymerase; DEXXQ: DEXXQ-box helicase domain of Upf1-like helicase; DNA2: Superfamily ⅠDNA and/or RNA helicase; ExoN: Exoribonuclease; NendoU: Uridylate-specific endoribonuclease; S: Spike protein; E: Envelope protein; M: Membrane protein; N: Nucleocapsid protein |
2.2 基于SARS-CoV-2与bat CoV RaTG13中蛋白质分子三维结构的变异氨基酸电荷相互作用分析
为了更加准确地界定SARS-CoV-2中发挥关键作用的氨基酸残基,我们重建了SARS-CoV-2和bat CoV RaTG13主要编码蛋白的三维结构(表 1)。这些蛋白三维结构的GMQE值为0.73~0.98;除了ExoN和S蛋白,其他蛋白的QMEAN值均约为0。尽管这2种病毒的ExoN和S蛋白的QMEAN值偏离了0点,但是数值非常接近,其结构具有可比性。
|
|
表 1 重建三维结构的SARS-CoV-2和bat CoV RaTG13中主要蛋白质分子 Tab 1 Major proteins with three-dimensional structure reconstruction from SARS-CoV-2 and bat CoV RaTG13 |
2.2.1 RNA合成相关酶的氨基酸变异对电荷相互作用的影响
通过氨基酸序列比对分析,发现负责SARS-CoV-2与bat CoV RaTG13基因组RNA合成的Nsp非常保守,变异率较低或根本没有变异(图 3)。RdRp在冠状病毒RNA复制过程中发挥了关键作用,其催化效率及保真性直接关系到病毒RNA合成的数量和变异速度[11]。SARS-CoV-2与bat CoV RaTG13的RdRp蛋白中共有4个氨基酸残基发生变异,其中2个位点属于近似变异(R-K、V-I),而第185位和第719位的变异属于非近似变异。
根据三维结构中侧链基团氮氧原子之间的距离测定,判断bat CoV RaTG13 RdRp蛋白氨基端D185与R184之间可以形成强盐键;而SARS-CoV-2中N185则无法形成盐键(图 4A~4C)。bat CoV RaTG13 RdRp蛋白羧基端质子化的H719可以与D717形成一个盐键,而SARS-CoV-2中的Y719则无法与D717形成盐键(图 4D~4F)。因此,与bat CoV RaTG13相比,SARS-CoV-2 RdRp蛋白靠近两端部分由于氨基酸变异各消除了一个盐键。而在SARS-CoV RdRp三维结构(PDB:6nur)中,D198(对应bat CoV RaTG13中的D185)与R197形成一个强盐键;其Y719(对应SARS-CoV-2中的Y719)无法与D717形成盐键。有意思的是,在bat CoV RaTG13中,K268(对应SARS-CoV中的C281)与R272通过正电排斥更靠近E265,从而形成K268-E265强盐键(对应SARS-CoV中的E278-R285弱盐键)。因此,与SARS-CoV相比,SARS-CoV-2 RdRp蛋白由于氨基酸变异在氨基端消除了一个盐键,再形成了一个强盐键。由此可见,SARS-CoV-2 RdRp蛋白两末端部位的盐键发生了明显的变化,可能会影响其构象和催化活性。
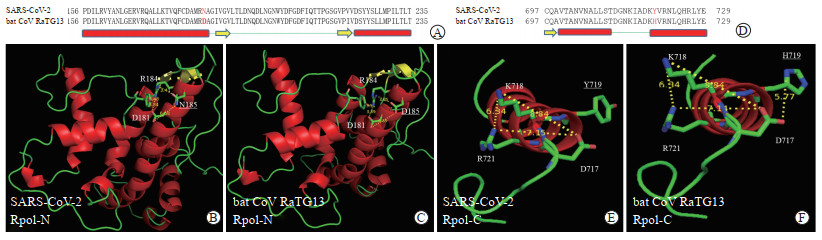
|
图 4 SARS-CoV-2和bat CoV RaTG13 RdRp蛋白中关键氨基酸变异及电荷相互作用 Fig 4 Key amino acid variations and electrostatic interactions in RdRp from SARS-CoV-2 and bat CoV RaTG13 A, D: Amino acid sequences shown in the three-dimensional structure. The variant amino acid residues are in red. Green lines, yellow arrows and red bars indicate loops, β-folds and α-helixes, respectively. B, C, E, F: Partial three-dimensional structure of RdRp. N185, D185, Y719 and H719 are the variant amino acid residues. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; bat CoV: Bat coronavirus; RdRp: RNA-dependent RNA polymerase; Rpol-N: N-terminus of RdRp; Rpol-C: C-terminus of RdRp |
通过比较SARS-CoV-2与bat CoV RaTG13中ExoN蛋白的氨基酸序列,发现有4个氨基酸残基出现变异,包括3个近似突变(V-I、I-V和R-K)和1个非近似突变(L-R)(图 5A)。根据三维结构中侧链基团氮氧原子间的距离判断:在bat CoV RaTG13 ExoN蛋白中,质子化的H281与E282形成一个较强的盐键,而R257所带的正电荷通过正电斥力使H281更靠近E282,增强二者形成的盐键;而在SARS-CoV-2中,L257取代了R257,因此不会对H281和E282之间的盐键产生增强作用(图 5B、5C)。而在SARS-CoV ExoN蛋白结构(PDB:5nfy)中,对应的H283-E284盐键由于质子化的H260的正电斥力而加强。因此,SARS-CoV-2 ExoN蛋白中的E282形成的盐键最弱,可能影响该酶的分子构象和生物化学功能。
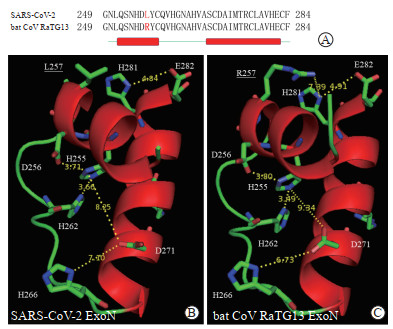
|
图 5 SARS-CoV-2和bat CoV RaTG13 ExoN蛋白中关键氨基酸变异及电荷相互作用 Fig 5 Key amino acid variations and electrostatic interactions in ExoN from SARS-CoV-2 and bat CoV RaTG13 A: Amino acid sequences shown in the three-dimensional structure. The variant amino acid residues are in red. Green lines and red bars indicate loops and α-helixes, respectively. B, C: Partial three-dimensional structure of ExoN. L257 and R257 are the variant amino acid residues. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; bat CoV: Bat coronavirus; ExoN: Exoribonuclease |
通过分析SARS-CoV-2和bat CoV RaTG13中NendoU蛋白氨基酸序列,发现变异率高达2.31%(图 2),非近似氨基酸变异率达到1.16%(图 3),是该区域变异率最高的一个蛋白。在SARS-CoV-2中这些非近似的氨基酸变异包括D128、D183、Q201和S243等4个氨基酸残基(图 6A)。D128通过负电斥力增强D124与R126形成的盐键,D183与R138形成一个强盐键(图 6B)。而在bat CoV RaTG13中,N128无法增强D124和R126之间的盐键,N183也无法与R138形成盐键(图 6C)。在该病毒中K201与E202形成盐键,R243通过正电斥力增强质子化的H242与D239之间的盐键(图 6C)。但是,在SARS-CoV-2中Q201不能与E202形成盐键,S243也不能增强质子化的H242与D239之间的盐键(图 6B)。因此,与bat CoV RaTG13相比,SARS-CoV-2 NendoU蛋白中的盐键发生了明显变化。在SARS-CoV NendoU蛋白三维结构(PDB:2h85)中,质子化的H21与D18形成强盐键(在SARS-CoV-2和bat CoV RaTG13中则为Q21,无法形成盐键);D201、E203和D204形成一个负电斥力环(在SARS-CoV-2和bat CoV RaTG13中,该区域只有E202,无法形成负电斥力环);K266与D241形成强盐键(在SARS-CoV-2和bat CoV RaTG13中,对应的E264、E266与D267形成一个负电斥力环)。因此,与SARS-CoV相比,SARS-CoV-2 NendoU蛋白中电荷相互作用也发生了明显变化,这可能影响其构象和生物学功能。
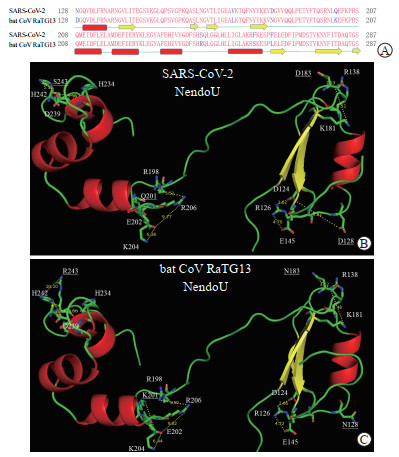
|
图 6 SARS-CoV-2和bat CoV RaTG13 NendoU蛋白中关键氨基酸变异及电荷相互作用 Fig 6 Key amino acid variations and electrostatic interactions in NendoU from SARS-CoV-2 and bat CoV RaTG13 A: Amino acid sequences shown in the three-dimensional structure. The variant amino acid residues are in blue. Green lines, yellow arrows and red bars indicate loops, β-folds and α-helixes, respectively. B, C: Partial three-dimensional structure of NendoU. S243, Q201, D183, D128, R243, K201, N183 and N128 are variant amino acid residues. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; bat CoV: Bat coronavirus; NendoU: Uridylate-specific endoribonuclease |
简而言之,与bat CoV RaTG13相比,SARS-CoV-2的RdRp、ExoN和NendoU 3个蛋白上的7处氨基酸发生变异(表 2),可能影响了这些RNA合成相关酶的构象和功能,从而改变病毒RNA复制的速率和保真性。
|
|
表 2 SARS-CoV-2中主要Nsp分子的氨基酸变异位点 Tab 2 Amino acid variation sites of Nsp from SARS-CoV-2 |
2.2.2 S蛋白的氨基酸变异对电荷相互作用的影响
S蛋白是冠状病毒中最关键的结构蛋白之一,由S1亚基和S2亚基组成。其中,S1的羧基端含有一个RBD,可以与宿主细胞表面受体如血管紧张素转化酶2(angiotensin-converting enzyme 2,ACE2)结合,从而改变蛋白构象,诱导S2亚基中的疏水融合肽暴露,介导病毒与宿主细胞发生膜融合[12]。因此,S蛋白是冠状病毒侵入宿主细胞的“钥匙”。在bat CoV RaTG13三聚化的S蛋白中,S1/S2切割位点处的E670和D672之间由于负电斥力保持8.2~8.4 Å距离(在3个单体中分别为8.20、8.23和8.38 Å)。而在SARS-CoV-2 S蛋白中,S680处(位于S2亚基的氨基端)插入了PRRA肽段,这一插入导致S1/S2切割位点处的E661和D663之间的距离明显缩短为6.8~7.0 Å(在3个单体中分别为6.79、6.81和6.98 Å),说明这2个氨基酸残基间的负电斥力明显增强,从而影响该区域的蛋白质构象,推测很可能会影响S1和S2亚基的剪切、ACE2受体的识别与结合或膜融合。
进一步对SARS-CoV-2和bat CoV RaTG13中S蛋白的氨基酸序列进行比对分析,发现二者的RBD共有19个氨基酸残基发生变异,其中有9个氨基酸残基涉及电荷变化。这些氨基酸变异中有1个位于受体结合位点的左侧,其他均位于右侧。根据三维结构中侧链基团氮氧原子间的距离判断:bat CoV RaTG13 S蛋白的RBD含有3个强盐键(D451-R453、D510-质子化的H514、D414-R417),且该区域含有较多的碱性氨基酸残基,因此这些盐键的作用力受正电斥力作用而增强。而在SARS-CoV-2 S蛋白的RBD,其受体结合位点右侧只形成了2个强盐键(R403-E406、D405-R408),而且由于含有的碱性氨基酸残基较少,盐键的正电排斥增强效应减弱;另外,其左侧则形成了一个全新的盐键(E516-质子化的H519)。由此可见,与bat CoV RaTG13相比,SARS-CoV-2 S蛋白受体结合位点两侧的盐键发生了明显变化,这必然影响其与受体的结合能力。已有研究报道,与SARS-CoV相比,SARS-CoV-2 S蛋白的受体结合位点处有多个氨基酸残基发生突变,但是受体结合的三维构象并没有改变[13]。最近的一项研究显示,SARS-CoV-2 S蛋白与ACE2受体的结合能力远高于bat CoV RaTG13(超过10倍)[15]。我们的分析结果也支持上述研究结果[15],提示SARS-CoV-2 S蛋白中3处关键的氨基酸变异可能影响了该病毒与受体的相互作用。
最近,有研究指出穿山甲冠状病毒与SARS-CoV-2的S蛋白高度同源(氨基酸序列一致性达到97%),推断SARS-CoV-2可能是这株穿山甲冠状病毒与bat CoV RaTG13基因组重组的结果[9, 14]。但是,穿山甲冠状病毒的S1/S2区域并无上述的PRRA短肽插入,而这个插入序列却曾在流感病毒和中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus,MERS-CoV)中出现[9, 14],暗示SARS-CoV-2 S蛋白的进化可能更为复杂,也许发生了不止一次的重组事件。
2.2.3 蛋白酶的氨基酸变异对电荷相互作用的影响PLpro是SARS-CoV及其他冠状病毒Orf1ab氨基端3个Nsp(Nsp1,Nsp2,Nsp3)剪切成熟所必需的蛋白水解酶[17]。对SARS-CoV-2和bat CoV RaTG13中PLpro蛋白进行氨基酸序列比对分析,发现二者之间有7个氨基酸变异,其中关键的一个变异位于第232位氨基酸残基上。根据PLpro蛋白三维结构中侧链基团氮氧原子间的距离判断,该变异位点附近的E203与R183可以形成一个较强的盐键。在SARS-CoV-2中,K232则通过正电斥力增强R183与E203间的盐键;在bat CoV RaTG13中,Q232则无增强作用。不过,盐键强弱的变化(甚至包括PLpro蛋白近羧基端的T-I变异)似乎并没有影响蛋白酶催化中心氨基酸残基的相互作用。因此,SARS-CoV-2 PLpro蛋白的氨基酸变异对蛋白酶构象及催化功能的影响可能并不明显。
3CLpro是对SARS-CoV及其他冠状病毒的Nsp进行成熟切割的主要蛋白酶,在Orf1ab上至少有11个切割位点[16]。从重建的高度保守的蛋白质三维结构来看,SARS-CoV-2与bat CoV RaTG13中3CLpro蛋白之间有2个氨基酸变异(P68T和N152T),它们不影响氨基酸残基之间的电荷相互作用,也不影响蛋白酶的活性中心。因此,这2个变异位点可能不会改变3CLpro蛋白酶的构象和功能。
总之,本研究通过分析SARS-CoV-2和bat CoV RaTG13之间主要蛋白质分子的氨基酸变异,发现SARS-CoV-2编码的RdRp、ExoN、NendoU和S蛋白上至少发生了10处关键的氨基酸变异,这些变异改变了相关氨基酸残基之间的电荷相互作用,可能影响到相应蛋白的空间构象及病毒的遗传表型(如病毒RNA复制校正功能、致病性、与细胞受体的亲和力及传染性等)。
| [1] |
Word Health Organization. Coronavirus disease (COVID-19) outbreak situation[EB/OL]. (2020-04-15)[2020-04-15]. https://www.who.int/emergencies/diseases/novel-coronavirus-2019.
|
| [2] |
WU F, ZHAO S, YU B, CHEN Y M, WANG W, SONG Z G, et al. A new coronavirus associated with human respiratory disease in China[J]. Nature, 2020, 579: 265-269. DOI:10.1038/s41586-020-2008-3 |
| [3] |
ZHOU P, YANG X L, WANG X G, HU B, ZHANG L, ZHANG W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 579: 270-273. DOI:10.1038/s41586-020-2012-7 |
| [4] |
LI Q, GUAN X, WU P, WANG X, ZHOU L, TONG Y, et al. Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia[J]. N Engl J Med, 2020, 382: 1199-1207. DOI:10.1056/NEJMoa2001316 |
| [5] |
LU R, ZHAO X, LI J, NIU P, YANG B, WU H, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus:implications for virus origins and receptor binding[J]. Lancet, 2020, 395: 565-574. DOI:10.1016/S0140-6736(20)30251-8 |
| [6] |
CHAN J F, YUAN S, KOK K H, TO K K, CHU H, YANG J, et al. A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission:a study of a family cluster[J]. Lancet, 2020, 395: 514-523. DOI:10.1016/S0140-6736(20)30154-9 |
| [7] |
HUANG C, WANG Y, LI X, REN L, ZHAO J, HU Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. Lancet, 2020, 395: 497-506. DOI:10.1016/S0140-6736(20)30183-5 |
| [8] |
LAM T, SHUM M, ZHU H, TONG Y, NI X, LIAO Y, et al. Identification of 2019-nCoV related coronaviruses in Malayan pangolins in southern China[J/OL]. bioRxiv, 2020. doi: 10.1101/2020.02.13.945485. 10.1101/2020.02.13.945485
|
| [9] |
XIAO K, ZHAI J, FENG Y, ZHOU N, ZHANG X, ZOU J, et al. Isolation and characterization of 2019-nCoV-like coronavirus from Malayan pangolins[J/OL]. bioRxiv, 2020. doi: 10.1101/2020.02.17.951335. 10.1101/2020.02.17.951335
|
| [10] |
HILGENFELD R, PEIRIS M. From SARS to MERS:10 years of research on highly pathogenic human coronaviruses[J]. Antiviral Res, 2013, 100: 286-295. DOI:10.1016/j.antiviral.2013.08.015 |
| [11] |
SNIJDER E J, DECROL E, ZIEBUHR J. The nonstructural proteins directing coronavirus RNA synthesis and processing[J]. Adv Virus Res, 2016, 96: 59-126. DOI:10.1016/bs.aivir.2016.08.008 |
| [12] |
LI F. Structure, function, and evolution of coronavirus spike proteins[J]. Annu Rev Virol, 2016, 3: 237-261. DOI:10.1146/annurev-virology-110615-042301 |
| [13] |
XU X, CHEN P, WANG J, FENG J, ZHOU H, LI X, et al. Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission[J]. Sci China Life Sci, 2020, 63: 457-460. DOI:10.1007/s11427-020-1637-5 |
| [14] |
WU A, NIU P, WANG L, ZHOU H, ZHAO X, WANG W, et al. Mutations, recombination and insertion in the evolution of 2019-nCoV[J/OL]. bioRxiv, 2020. doi: 10.1101/2020.02.29.971101. 10.1101/2020.02.29.971101
|
| [15] |
WRAPP D, WANG N, CORBETT K, GOLDSMITH J A, HSIEH C L, ABIONA O, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation[J]. Science, 2020, 367: 1260-1263. DOI:10.1126/science.abb2507 |
| [16] |
WATERHOUSE A, BERTONI M, BIENERT S, STUDER G, TAURIELLO G, GUMIENNY R, et al. SWISS-MODEL:homology modelling of protein structures and complexes[J]. Nucleic Acids Res, 2018, 46(W1): W296-W303. DOI:10.1093/nar/gky427 |
| [17] |
HILGENFELD R. From SARS to MERS:crystallographic studies on coronaviral proteases enable antiviral drug design[J]. FEBS J, 2014, 281: 4085-4096. DOI:10.1111/febs.12936 |
 2020, Vol. 41
2020, Vol. 41


