慢性乙型肝炎病毒(hepatitis B virus,HBV)感染是引发肝硬化、肝功能衰竭、肝癌的主要原因[1]。我国HBV感染者约有7 000万人,其中慢性乙型病毒性肝炎(chronic hepatitis B,CHB)患者有2 000万~3 000万人[2]。HBV感染的肝细胞内的共价闭合环状DNA(covalently closed circular DNA,cccDNA)是HBV前基因组RNA(pregenomic RNA,pgRNA)的唯一转录模板[3],pgRNA在病毒衣壳内反转录合成松弛环状DNA(relaxed circular DNA,rcDNA),同时病毒衣壳被包膜包裹形成完整病毒颗粒并释放到细胞外,然后再侵入感染新的肝细胞,在细胞核内rcDNA被修复为完整的cccDNA,开始新的一轮复制周期[4]。上述HBV的生命过程揭示肝内cccDNA的存在是HBV复制活跃的基础。临床研究发现,肝内能否检测到cccDNA是HBV是否被彻底治愈的重要指标[5]。通过肝内穿刺活组织检查可以定量检测cccDNA,但穿刺属于有创检查,不能作为临床常规检查手段。目前临床上已经常规应用一些血清学指标替代有创检测,如用HBsAg、HBcAb、HBV DNA等监测肝内HBV的复制水平,但这些指标均有其局限性。例如,HBV患者经过核苷类似物治疗后,血清HBV DNA水平可迅速降低到检测下限,但肝脏活组织检查仍然能检出cccDNA,此时若停药,复发的可能性将增大[6]。
研究显示血清中存在包含HBV pgRNA的类病毒颗粒,其滴度和经核苷类似物治疗停药后病毒的复发率具有相关性,有望成为新的临床参考指标[7]。但是目前尚没有公认的HBV RNA的检测方法。部分已发表文献中的检测方法是抽提样品总RNA,反转录后再做定量PCR[8],但RNA提取物中可能存在HBV DNA污染,从而导致定量结果偏高或假阳性。其他文献在反转录前使用DNA酶消化HBV DNA[9],但这一过程能否完全降解HBV DNA尚不明确,此外消化需要稀释RNA也可能降低检测灵敏度。本研究设计了一种新的HBV RNA检测方法,该方法不仅能够有效解决HBV DNA的污染问题,还可在一管反应中同时测定细胞培养上清中的HBV DNA和pgRNA,极大提高了检测效率,降低了人力、经济成本,具有潜在的临床应用价值。
1 材料和方法 1.1 引物与探针为保证本检测方法的通用性,从HBV database网站上下载了8条不同的HBV全基因组序列(HBV基因型A、B、C、D各2条),对比其共有保守序列设计引物和探针。本方法检测的是包括rcDNA和cccDNA在内的总HBV DNA,rcDNA相较于cccDNA在正链有部分缺失,因此避开rcDNA正链缺失部分,选择rcDNA和cccDNA共有的双链部分设计上、下游引物和探针,分别命名为AF、AR、PA,PCR产物seq A长度为173 bp,定位于HBV基因组的S区;HBV RNA包括0.7、2.1、2.4、3.5 kb 4种不同长度的5种RNA(图 1)[10-11]。为了特异性检测pgRNA,避免其他RNA的干扰,在pgRNA 5'端1 kb内的区间设计上、下游引物和探针(这段区间内的序列只有3.5 kb全长的转录本才有,其他较短的RNA转录本缺失此段序列,因此可以实现pgRNA的特异性检测),分别命名为BF、BR、PB,PCR产物seq B长度为118 bp,定位于HBV基因组的C区。引物、探针由生工生物工程(上海)股份有限公司合成,引物探针组A的上游引物AF(方向5'-3',下同)序列为GTG GTG GAC TTC TCT CAA TTT TCT AG,下游引物AR序列为TGA GGC ATA GCA GCA GGA TG,探针PA和修饰基团序列为FAM-ACG CCG CAG ACA CAT-MGB;引物探针组B的上游引物BF序列为CCC TAT CTT ATC AAC ACT TCC GGA,下游引物BR序列为AGA TCT TCT GCG ACG CGG,探针PB和修饰基团序列为Texas Red-CGT CTG CGA GGC GAG-MGB。
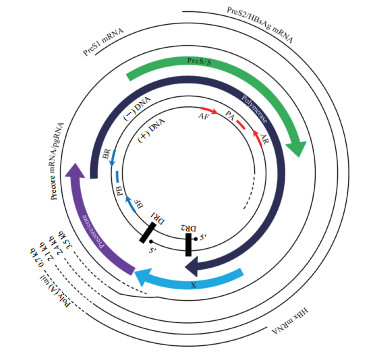
|
图 1 HBV rcDNA、基因、mRNA和引物、探针设计位点示意图(图片修改自文献[11]) Fig 1 Diagram of rcDNA, genes, mRNAs of HBV and sites of primers and probes (modified from reference[11]) HBV: Hepatitis B virus; rcDNA: Relaxed circular DNA; pgRNA: Pregenomic RNA; HBsAg: Hepatitis B surface antigen; HBx: Hepatitis B virus X; DR1: Direct repeat 1; DR2: Direct repeat 2; AF, AR: Forward and reverse primers of PCR (seq A); BF, BR: Forward and reverse primers of PCR (seq B); PA, PB: Probes for seq A and seq B, respectively; PCR: Polymerase chain reaction |
1.2 细胞系及其培养方法
人肝癌细胞系HepG2、SMMC-7721购自中国科学院细胞库;稳转含HBV全长基因组载体、表达类似HBV Dane颗粒[12]的人肝癌细胞系HepG2.2.15由中国医学科学院基础医学研究所陈坤博士惠赠。HepG2、SMMC-7721细胞常规培养于含10% FBS(美国Gibco公司)的DMEM培养液(美国Gibco公司)。HepG2.2.15细胞常规培养于含10% FBS的MEM培养液(法国Biowest公司)。
1.3 载体质粒pHBV1.3含有1.3倍全长HBV基因组(基因分型为B型,GenBank登录号: JN406371.1),载体骨架为pBluescript Ⅱ KS+,基因组插入载体的酶切位点分别为EcoR Ⅰ和Sal Ⅰ[13],由中国医学科学院基础医学研究所陈坤博士惠赠。
1.4 样本中RNA、核酸提取及反转录采用柱式法(RNAfast200总RNA急速抽提试剂盒,上海飞捷生物技术有限公司)提取细胞总RNA,采用柱式法(血清RNA柱式法提取试剂盒,常州百代生物科技有限公司)提取细胞培养上清中的RNA,采用磁珠法(核酸提取或纯化试剂盒,广州达安基因股份有限公司)提取细胞或培养上清中的总核酸。用反转录试剂盒(日本Toyobo公司)将RNA反转录合成cDNA,按照试剂盒说明书常规操作。
1.5 双重定量PCR体系采用qRT-PCR试剂盒(常州百代生物科技有限公司)进行双重定量PCR,总反应体系为20 μL,其中10 μmol/L引物、探针各取0.33 μL,检测样本1 μL,其余试剂用量同试剂盒说明书。采用LightCycler 96型自动化荧光定量PCR仪(瑞士Roche公司)进行检测,荧光通道分别选择FAM检测探针PA和Texas Red检测探针PB。反应条件为95 ℃预变性10 min,95 ℃变性12 s、60 ℃退火延伸25 s,40个循环。定量方法为绝对定量,标准品为pHBV1.3载体。使用NanoDrop分光光度计(美国ThermoFisher公司)测定浓度并计算拷贝数后,用DNA稀释液[生工生物工程(上海)股份有限公司]按照10倍梯度稀释。最终稀释成2.13×107、2.13×106、2.13×105、2.13×104、2.13×103拷贝/μL 5个浓度,作为绝对定量标准品。为了比较双重定量PCR和常规定量PCR结果的一致性,分别使用常规、双重定量PCR检测已知浓度为2.13×105拷贝/μL的标准品,记录以下数据(每组重复10次):(1)常规定量PCR扩增seq A的拷贝数;(2)常规定量PCR扩增seq B的拷贝数;(3)双重定量PCR同时扩增seq A和seq B二者各自的拷贝数。用统计学方法将检测值与实际浓度进行比较。
1.6 DNA凝胶电泳观察PCR产物条带按2.5%浓度在1×TAE缓冲液中加入琼脂糖粉末,用微波炉加热煮沸溶解,冷却至40 ℃后按1︰10 000比例加入核酸染料(上海天能科技有限公司),倒入加样模具凝固成胶块,将胶块放入盛有1×TAE缓冲液的电泳槽中。在DNA样品中加入适量6×电泳上样缓冲液(日本TaKaRa公司)配制成工作浓度,加入胶块加样孔中,100 V电泳30 min,于凝胶成像系统(上海天能科技有限公司)下紫外线曝光拍照。
1.7 验证HepG2.2.15细胞培养上清中含有HBV pgRNA设计实验如下:(1)磁珠法抽提HepG2.2.15细胞培养上清中的总核酸,用基因引物BR反转录或者不处理(在等量核酸提取物中加入水,使总体积和反转录组相同),然后分别用定量PCR测定seq B的初始拷贝数和反转录后拷贝数,比较二者有无差异;(2)用RNA酶A(RNase A)消化总核酸或不处理,再用BR反转录,分别通过定量PCR测定RNase A处理和不处理组的seq B拷贝数,比较二者有无差异。RNase A消化过程如下:用水将10 mg/mL的RNase A(美国CST公司)原液配制成100 μg/mL质量浓度,取1 μL加入9 μL核酸提取物中,使RNase A达到10 μg/mL的工作浓度,37 ℃消化30 min,随后加入1 μL重组RNA酶抑制剂(日本TaKaRa公司)终止消化。对照组不处理,在9 μL核酸提取物中加水2 μL至总体积11 μL。
1.8 验证通过反转录后拷贝数相减法检测细胞培养上清中HBV pgRNA的可行性用双重定量PCR检测细胞培养上清核酸提取物中seq A和seq B初始拷贝数,比较二者有无差异;使用BR对核酸提取物反转录或不处理,分别检测seq A的初始拷贝数和反转录后拷贝数,比较二者有无差异。
1.9 验证本方法检测细胞中HBV pgRNA的可行性抽提HepG2.2.15细胞的总核酸,并用双重定量PCR检测seq A和seq B的初始拷贝数,比较二者有无差异。用基因特异性引物AR对核酸提取物进行反转录并再次测定seq A与seq B拷贝数,比较反转录前后seq A和seq B增加的拷贝数有无差异。
1.10 检验本方法测定浓度和实际浓度的相关性将HepG2.2.15细胞培养上清用新鲜培养液进行2倍和4倍稀释,然后分别提取培养上清原液、2倍稀释培养上清、4倍稀释培养上清、新鲜培养液(阴性对照)中的总核酸,用双重定量PCR测定HBV DNA、pgRNA的拷贝数,考察不同稀释倍数和检测值之间的相关性。
1.11 统计学处理应用GraphPad Prism 5.0软件进行数据分析,呈正态分布的计量资料以x±s表示,两组间比较采用Student t检验。对于不同稀释倍数细胞培养上清和实测拷贝数之间的相关性比较,首先将稀释倍数和实测拷贝数均作对数变换,即变成以2为底的稀释倍数或拷贝数的对数,然后对二者进行Pearson相关性分析。检验水准(α)为0.05。
2 结果 2.1 双重定量PCR体系的建立 2.1.1 最佳的退火、延伸温度为60 ℃DNA凝胶电泳显示,当退火温度较低时反应体系会产生118 bp和173 bp目的条带以外的非特异性条带,随着温度升高非特异性扩增减少,60 ℃时非特异性条带完全消失(图 2A);而在60 ℃、62 ℃、64 ℃、66 ℃等更高退火、延伸温度下,随着温度升高特异性条带减弱,说明60 ℃具有最佳的扩增效率,上述温度下HepG2和SMMC-7721 cDNA模板均未见扩增条带(图 2B);在60 ℃的退火、延伸温度下,常规定量PCR和双重定量PCR条带位置一致,并且与173 bp的seq A和118 bp的seq B大小相符(图 2C)。
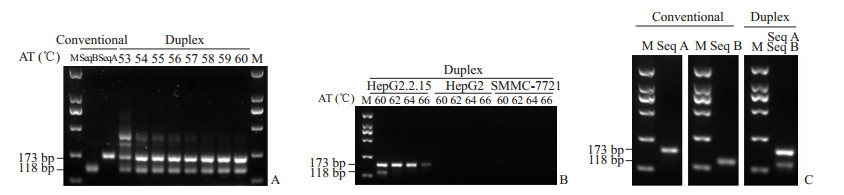
|
图 2 PCR产物的DNA凝胶电泳图 Fig 2 DNA gel electrophoresis images of PCR products A: Conventional and duplex PCR were performed at 53-60 ℃ annealing temperature (AT) with templates of HepG2.2.15 cDNA; B: Duplex PCR was performed at 60-66 ℃ with templates of HepG2.2.15, HepG2, and SMMC-7721 cDNA; C: Conventional PCR of seq A, conventional PCR of seq B and duplex PCR of seq A and seq B were performed at 60 ℃. M: Marker; PCR: Polymerase chain reaction |
2.1.2 双重定量PCR的标准曲线具有较好的扩增效率
把浓度的常用对数作为x、Ct值作为y绘制标准曲线,seq A的线性回归方程为y=-3.352x+37.329,扩增效率为99%,R2=0.997 6;seq B的线性回归方程为y=-3.443x+37.341,扩增效率为95%,R2=0.996 6。
2.1.3 双重定量PCR与常规定量PCR的定量结果均与实际值一致检测已知浓度为2.13×105拷贝/μL(拷贝数用常用对数变换后为5.328拷贝/μL)的标准品,检测结果以拷贝数的常用对数表示。常规定量PCR检测seq A的拷贝数为(5.353±0.058)拷贝/μL,双重定量PCR检测seq A的拷贝数为(5.343±0.040)拷贝/μL,二者差异无统计学意义(P>0.05,n=10);常规定量PCR检测seq B的拷贝数为(5.355±0.060)拷贝/μL,双重定量PCR检测seq B的拷贝数为(5.312±0.048)拷贝/μL,二者差异也无统计学意义(P>0.05,n=10)。上述结果与实际值进行比较,差异均无统计学意义(P均>0.05)。
2.2 验证HepG2.2.15细胞培养上清中含有HBV pgRNA使用基因特异性引物BR反转录后,细胞培养上清核酸提取物中的seq B拷贝数为(4 013±319)拷贝/μL;未经反转录处理的对照组seq B初始拷贝数为(2 354±156)拷贝/μL,二者差异有统计学意义(P<0.01,n=5)。使用RNase A消化细胞培养上清核酸提取物,经过BR反转录后测定的seq B拷贝数为(2 340±287)拷贝/μL;未经RNase A消化的对照组,经过BR反转录后测定的seq B拷贝数为(3 776±516)拷贝/μL,二者差异有统计学意义(P<0.05,n=5)。
2.3 验证通过反转录后拷贝数相减法检测细胞培养上清中HBV pgRNA的可行性细胞培养上清核酸提取物中seq A的初始拷贝数为(2 323±108)拷贝/μL,seq B的初始拷贝数为(2 354±156)拷贝/μL,二者差异无统计学意义(P>0.05,n=5);使用BR反转录后,seq A的拷贝数为(2 298±159)拷贝/μL,与seq A的初始拷贝数相比,二者差异无统计学意义(P>0.05,n=5)。
2.4 本方法不适用于检测细胞中HBV pgRNA的水平HepG2.2.15细胞核酸提取物中seq A的初始拷贝数为(3 528±166)拷贝/μL,seq B的初始拷贝数为(2 746±165)拷贝/μL,二者差异有统计学意义(P<0.01,n=5);使用AR反转录后,seq A增加的拷贝数为(54 180±1 115)拷贝/μL,seq B增加的拷贝数为(3 796±383)拷贝/μL,二者差异有统计学意义(P<0.01,n=5)。
2.5 本方法的测定结果与细胞培养上清中实际抽提HBV总核酸的含量呈现较好的对应关系对检测结果进行以2为底的对数变换,HepG2.2.15细胞培养上清原液中HBV DNA的拷贝数为(13.052±0.066)拷贝/μL,HBV pgRNA拷贝数为(11.556±0.270)拷贝/μL;2倍稀释培养上清中HBV DNA拷贝数为(11.850±0.125)拷贝/μL,HBV pgRNA拷贝数为(10.911±0.520)拷贝/μL;4倍稀释培养液中HBV DNA拷贝数为(11.023±0.123)拷贝/μL,HBV pgRNA拷贝数为(9.834±0.319)拷贝/μL;阴性对照样本均未检出HBV DNA或pgRNA。对对数转换后的稀释倍数和实测拷贝数进行相关性分析,稀释倍数和HBV DNA拷贝数的相关系数r=-0.986 1,R2=0.972 4(P<0.01,n=15),表明稀释倍数与HBV DNA拷贝数具有较强的负相关性;稀释倍数和HBV pgRNA拷贝数的相关系数r=-0.870 2,R2=0.757 2(P<0.01,n=15),表明稀释倍数与HBV pgRNA拷贝数也具有较强的负相关性。
3 讨论本研究设计了一种新的HBV RNA的检测方法,首先抽提包含DNA和RNA在内的总核酸,然后使用基因特异性引物反转录部分HBV pgRNA,通过双重荧光定量PCR同时定量HBV基因组上未经反转录段的HBV DNA拷贝数和经过反转录段的HBV DNA+pgRNA拷贝数,将二者相减间接求得HBV pgRNA的拷贝数。PCR-DNA凝胶电泳结果显示,本研究设计的双重定量PCR体系的最佳退火温度是60 ℃,此时具有较好的特异性和灵敏度。用2.13×107~2.13×103拷贝/μL 5个不同浓度的标准品绘制双重定量PCR标准曲线,2条标准曲线均有较高的扩增效率(≥95%),将标准品进一步稀释并测量,发现2条标准曲线的线性范围均可低至2.13×102拷贝/μL(结果未显示),说明本检测体系具有较高的灵敏度。
为了保证本检测方法的准确性,需要满足以下2个条件:(1)反转录前seq A和seq B的初始拷贝数相等;(2)反转录过程对seq A(即HBV DNA)的拷贝数无明显影响。本实验结果表明,在以HepG2.2.15细胞培养上清作为样本时满足上述条件,因此该方法用于测定细胞培养上清中的HBV DNA和pgRNA具有可行性;但是在检测细胞样本时发现seq A初始拷贝数>seq B,表明在胞内有整合于细胞基因组的HBV DNA。有研究者对HepG2.2.15细胞系进行了基因组和RNA水平的二代测序,结果显示HepG2.2.15基因组中整合了HBV DNA并且高表达相应的mRNA[14],这和本实验测定结果一致。由于细胞内整合HBV DNA的存在,seq A和seq B的初始拷贝数不同,因此通过拷贝数相减法间接计算HBV pgRNA可能会引起偏差。综上所述,本实验建立的检测体系适合用于测定细胞培养上清中的HBV DNA和pgRNA,不适用测定细胞样本,这也是下一步需要改进的问题。
分别检测HepG2.2.15细胞培养上清原液、2倍稀释培养上清、4倍稀释培养上清中HBV DNA和pgRNA的水平,结果显示稀释倍数和实测DNA和pgRNA拷贝数均存在较强的负相关性,说明本实验的检测值能够较为客观地反映样本中HBV DNA和pgRNA的实际浓度,具有较高的准确性。
研究发现血清HBV DNA和RNA的综合水平对于判断HBV感染阶段、疾病预后具有重要意义[15],因此临床实践中往往需要同时检测HBV DNA和RNA。本研究设计的检测方法通过双重荧光定量PCR,可一次检出HBV DNA和pgRNA的水平,整个过程均可通过全自动化仪器完成,耗时在2 h以内(已发表文献抽提总RNA测定HBV RNA的方法中,DNA酶消化步骤需要额外的人力,而且会降低检测的灵敏度[16],依据笔者经验需额外耗时40 min,如另外检测HBV DNA所需时间将更长)。本实验提出的方法相比于分别检测HBV DNA和pgRNA所需的样本量更少(理论上只需后者的一半),同时耗费的检测试剂及采样管等其他的医疗资源也会更少。
本实验建立的检测体系能够有效避免HBV DNA污染对结果的影响,可同时检测出HBV DNA和pgRNA,提高了检测效率,具有操作简单、节省材料和人力等优点,临床实用性较强。该方法的局限性是整合于细胞基因组的HBV DNA可能会对测定结果造成影响,因此理论上不适用于细胞检测,更适用于测定细胞培养上清或血清中的HBV DNA和pgRNA。
| [1] |
SCHWEITZER A, HORN J, MIKOLAJCZYK R T, KRAUSE G, OTT J J. Estimations of worldwide prevalence of chronic hepatitis B virus infection:a systematic review of data published between 1965 and 2013[J]. Lancet, 2015, 386: 1546-1555. DOI:10.1016/S0140-6736(15)61412-X |
| [2] |
王贵强, 王福生, 庄辉, 李太生, 郑素军, 赵鸿, 等.慢性乙型肝炎防治指南(2019年版)[J/CD].中华实验和临床感染病杂志(电子版), 2019, 13: 441-466.
|
| [3] |
鲁凤民, 曾婉嘉, 文夏杰, 廖昊, 邹军, 王雷婕, 等. 慢性乙型肝炎抗病毒治疗相关新型标志物及其临床应用[J]. 肝脏, 2019, 24: 483-486. DOI:10.3969/j.issn.1008-1704.2019.05.001 |
| [4] |
WANG J, HUANG H, LIU Y, CHEN R, YAN Y, SHI S, et al. HBV genome and life cycle[J]. Adv Exp Med Biol, 2020, 1179: 17-37. |
| [5] |
NASSAL M. HBV cccDNA:viral persistence reservoir and key obstacle for a cure of chronic hepatitis B[J]. Gut, 2015, 64: 1972-1984. DOI:10.1136/gutjnl-2015-309809 |
| [6] |
鲁凤民, 窦晓光, 张文宏, 王福生. 慢性乙型肝炎患者血清HBV RNA检测的临床意义[J]. 临床肝胆病杂志, 2018, 34: 934-938. DOI:10.3969/j.issn.1001-5256.2018.05.005 |
| [7] |
WANG J, SHEN T, HUANG X, KUMAR G R, CHEN X, ZENG Z, et al. Serum hepatitis B virus RNA is encapsidated pregenome RNA that may be associated with persistence of viral infection and rebound[J]. J Hepatol, 2016, 65: 700-710. DOI:10.1016/j.jhep.2016.05.029 |
| [8] |
WU L, WANG W, ZHANG X, ZHAO X, YU G. Anti-HBV activity and mechanism of marine-derived polyguluronate sulfate (PGS) in vitro[J]. Carbohydr Polym, 2016, 143: 139-148. DOI:10.1016/j.carbpol.2016.01.065 |
| [9] |
BUTLER E K, GERSCH J, MCNAMARA A, LUK K, HOLZMAYER V, DE MEDINA M, et al. Hepatitis B virus serum DNA and RNA levels in nucleos(t)ide analog-treated or untreated patients during chronic and acute infection[J]. Hepatology, 2018, 68: 2106-2107. DOI:10.1002/hep.30082 |
| [10] |
MITRA B, THAPA R J, GUO H, BLOCK T M. Host functions used by hepatitis B virus to complete its life cycle:implications for developing host-targeting agents to treat chronic hepatitis B[J]. Antiviral Res, 2018, 158: 185-198. DOI:10.1016/j.antiviral.2018.08.014 |
| [11] |
SEEGER C, MASON W S. Molecular biology of hepatitis B virus infection[J]. Virology, 2015, 479/480: 672-686. DOI:10.1016/j.virol.2015.02.031 |
| [12] |
SELLS M A, CHEN M, ACS G. Production of hepatitis B virus particles in Hep G2 cells transfected with cloned hepatitis B virus DNA[J]. Proc Natl Acad Sci USA, 1987, 84: 1005-1009. DOI:10.1073/pnas.84.4.1005 |
| [13] |
YUE X, WANG H, ZHAO F, LIU S, WU J, REN W, et al. Hepatitis B virus-induced calreticulin protein is involved in IFN resistance[J]. J Immunol, 2012, 189: 279-286. DOI:10.4049/jimmunol.1103405 |
| [14] |
HU X, JIANG J, NI C, XU Q, YE S, WU J, et al. HBV integration-mediated cell apoptosis in HepG2.2.15[J]. J Cancer, 2019, 10: 4142-4150. DOI:10.7150/jca.30493 |
| [15] |
HUANG H, WANG J, LI W, CHEN R, CHEN X, ZHANG F, et al. Serum HBV DNA plus RNA shows superiority in reflecting the activity of intrahepatic cccDNA in treatment-naïve HBV-infected individuals[J]. J Clin Virol, 2018, 99/100: 71-78. DOI:10.1016/j.jcv.2017.12.016 |
| [16] |
GAO M, FENG C, YING R, NIE Y, DENG X, ZHU Y, et al. A novel one-step quantitative reverse transcription PCR assay for selective amplification of hepatitis B virus pregenomic RNA from a mixture of HBV DNA and RNA in serum[J]. Arch Virol, 2019, 164: 2683-2690. DOI:10.1007/s00705-019-04372-0 |
 2020, Vol. 41
2020, Vol. 41


