2. 海军军医大学(第二军医大学)药学院药理学教研室, 上海 200433;
3. 海军军医大学(第二军医大学)基础医学院细胞生物学教研室, 上海 200433
2. Department of Pharmacology, School of Pharmacy, Naval Medical University(Second Military Medical University), Shanghai 200433, China;
3. Department of Cell Biology, College of Basic Medical Sciences, Naval Medical University(Second Military Medical University), Shanghai 200433, China
成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeat,CRISPR)及其相关蛋白9(CRISPR-associated protein 9,Cas9)介导的新一代基因编辑技术是近年来备受关注的高效且精准的基因编辑工具。CRISPR/Cas9系统是一种原核生物的适应性免疫防御系统,可以用来抵抗外来遗传物质的入侵,比如噬菌体病毒感染等[1]。当病毒首次入侵时,细菌能够将病毒的遗传物质片段整合到自身遗传信息的CRISPR序列中,CRISPR序列可转录成CRISPR RNA(crRNA)并能够与具有核酸内切酶活性的Cas9蛋白相结合;类似于哺乳动物的二次免疫反应,当与crRNA匹配的DNA片段再次出现时,Cas9利用其内切酶活性剪切这些DNA片段,防止病毒再次入侵[2-3]。正是因为这种精确的靶向功能,众多研究将CRISPR/Cas9应用于细胞的基因编辑[1, 4]。
人胚胎干细胞(human embryonic stem cell,hESC)是来自人类囊胚内细胞团的多能干细胞(pluripotent stem cell,PSC),具有分化为成年个体任何一种细胞的潜能。通过诱导hESC在体外的定向分化可以大量获得具有重要临床应用价值的功能细胞,如心肌细胞和神经元。PSC的体外分化过程通常忠实地模拟细胞的体内发育历程,因此人PSC也是在体外模拟和研究人类细胞早期发育的重要细胞工具。通过CRISPR/Cas9技术对hESC特定基因进行编辑可以在体外揭示相关基因对PSC定向分化的影响及其功能。
叉头框G1(forkhead box G1,FOXG1)是叉头框基因家族的转录因子。在目前研究的所有脊椎动物中,FOXG1是神经板端脑区域中首先表达的转录因子之一,它在前脑祖细胞中表达并持续到成年期。研究发现,人类FOXG1基因突变会导致发生先天性神经退行性疾病Rett综合征[5]。小鼠FOXG1基因敲除会导致脑部发育异常,引起严重小头畸形[6]。有文献报道,FOXG1的缺失会抑制端脑祖细胞的增殖而促进其分化,导致皮质神经元提前发生,阻碍晚期皮质神经元的产生[6-7]。本研究拟通过CRISPR/Cas9技术构建FOXG1基因缺失的hESC,并探究FOXG1基因在hESC早期神经诱导过程中的作用。
1 材料和方法 1.1 hESC的培养本实验采用由美国威斯康辛大学J. Thomson建立的H1系hESC(代次40~50),用E8培养基培养于2%基质胶包被的细胞培养板。细胞每天换液,待生长至70%~80%融合时用胰蛋白酶(美国Invitrogen公司)消化为单细胞,按1︰10传代。传代当天加入5 μmol/L非肌肉肌球蛋白Ⅱ型ATP酶抑制剂blebbistatin[美国Selleck生物科技有限公司(上海分公司)]防止细胞凋亡,blebbistatin于第2天换液后去除。
1.2 向导RNA(guide RNA,gRNA)的设计、载体构建及hESC的转染与克隆鉴定利用美国麻省理工学院张峰实验室的在线工具(https://zlab.bio/guide-design-resources)设计了2条针对人FOXG1基因(NM005249)的gRNA序列FOXG1-gRNA1(5’-TCG CGT GGT GGT TGT CGT TC-3’)和FOXG1-gRNA2(5’-GTC CGC TTT AGC CCC GTC CA-3’),并根据其实验室克隆策略将gRNA克隆到pX459载体(美国Addgene公司)[8]。
按照3×105/孔的密度将hESC接种至6孔板,第2天使用FuGENE HD转染试剂(美国Promega公司)将pX459 FOXG1-gRNA1和pX459 FOXG1-gRNA2质粒共转染hESC。转染24 h后接种至基质胶包被的10 cm培养皿,转染48 h开始使用嘌呤霉素0.5 μg/mL筛选2~3 d。接着去除嘌呤霉素继续培养约10 d,挑单克隆至基质胶包被的24孔培养板,再继续培养1周后按1︰2比例接种至2块24孔培养板。接种48 h后,用50 mmol/L的NaOH裂解其中一板细胞,作为基因组鉴定的模板;另一板细胞冻存,待鉴定后挑取并扩增阳性克隆。FOXG1基因的鉴定引物见表 1。hESC克隆基因型初始鉴定使用引物P1+和P1-组合,基因组PCR采用PrimerSTAR HS高保真PCR酶结合GC缓冲液[宝生物工程(大连)有限公司]对目的条带进行扩增,从中挑选出发生大片段敲除的单克隆,并对克隆基因型进行测序验证;P2+、P2-和P3-用于阳性克隆神经诱导后FOXG1基因表达的反转录聚合酶链反应(reverse transcription polymerase chain reaction,RT-PCR)鉴定。
|
|
表 1 用于hESC FOXG1基因型鉴定的PCR引物序列 Tab 1 Primer sequences for genotyping FOXG1 gene in hESCs by PCR |
1.3 hESC的神经诱导
本实验利用改进的双重Smad抑制[9]进行hESC的神经诱导。神经诱导的基础培养基为DMEM/F12、1×N2和1×B27;一组添加2 μmol/L的SB431542(nodal/activing/Smad2/3信号通路抑制剂)[美国Selleck生物科技有限公司(上海分公司)]和1 μmol/L的DMH1(骨形态发生蛋白受体抑制剂)[美国Selleck生物科技有限公司(上海分公司)],此诱导条件记为SD;另一组在SB431542和DMH1的基础上再添加0.1 μmol/L的LGK974(豪猪酶抑制剂,可以阻断WNT蛋白的分泌,抑制因WNT自分泌导致的神经后端化)[美国Selleck生物科技有限公司(上海分公司)],此诱导条件记为SDL。将hESC在上述2种诱导条件下连续培养7 d,一部分细胞用TRIzol试剂裂解用于RNA的提取和qRT-PCR分析,一部分细胞经多聚甲醛溶液固定进行细胞免疫荧光染色。
1.4 qRT-PCR分析用PrimeScript反转录试剂盒[宝生物工程(大连)有限公司]将RNA反转录为cDNA,使用SYBR Green预混试剂(美国Bio-Rad公司)和ABI7900定量PCR仪进行qRT-PCR,引物序列见表 2。使用相对Ct值分析法计算神经外胚层核心转录因子配对框基因6(paired box 6,PAX6)、神经干细胞核心转录因子性别决定区Y框蛋白2(sex-determining region Y-box 2,SOX2)、前脑特异的转录因子正小齿同源物2(orthodenticle homeobox 2,OTX2)mRNA的相对表达水平,以GAPDH为内参照。实验重复3次,结果以x±s形式表示。
|
|
表 2 qRT-PCR引物序列 Tab 2 Primer sequences for qRT-PCR |
1.5 细胞免疫荧光染色
细胞经4%多聚甲醛溶液室温固定10 min,用PBST(含0.2% Triton X-100的PBS)清洗3次,每次5 min;加入含有5%驴血清的PBST封闭液室温破膜封闭30 min,加一抗4 ℃过夜孵育;然后用PBST清洗3次,加入Alexa Fluor555和Alexa Fluor488标记的相应二抗(稀释1 000倍,美国Invitrogen公司)室温孵育1 h,DAPI复染细胞核后使用荧光显微镜(型号:ECLIPSE TE2000-U,日本Nikon公司)进行拍摄。使用的一抗包括兔抗FOXG1多克隆抗体(稀释500倍,英国Abcam公司)、兔抗PAX6多克隆抗体(稀释400倍,美国Proteintech公司)、小鼠抗SOX2单克隆抗体(稀释200倍,美国Santa Cruz公司)、小鼠抗八聚体结合转录因子4(octamer-binding transcription factor 4,OCT4)单克隆抗体(稀释200倍,美国Santa Cruz公司)。
1.6 细胞总蛋白的提取及蛋白质印迹分析将细胞用含0.5%苯甲基磺酰氟[生工生物工程(上海)有限公司]的RIPA蛋白裂解液(上海碧云天生物技术有限公司)裂解;13 800×g离心5 min后取蛋白上清,加入1/4体积的蛋白上样缓冲液,100 ℃水浴中加热10 min后上样到SDS-PAGE胶[雅酶生物科技(上海)有限公司]中,80 V恒压电泳100 min,然后将蛋白质转移到硝酸纤维素薄膜(美国Millipore公司)上。将膜用含5%脱脂牛奶的TBST(含0.1% Tween-20的Tris-HCl缓冲盐溶液)室温封闭120 min。加一抗4 ℃过夜孵育;然后用TBST洗3次后加入相应二抗(稀释20 000倍)室温孵育1 h,TBST洗3次后在膜上完全覆盖化学发光液(美国ThermoFisher公司),立即使用Amersham Imager 680化学发光成像仪(英国GE公司)拍摄。使用的一抗包括兔抗FOXG1单克隆抗体(稀释5 000倍,英国Abcam公司)、小鼠抗β-actin单克隆抗体(稀释1 000倍,美国Proteintech公司),使用的二抗包括HRP标记的山羊抗兔IgG、山羊抗小鼠IgG(美国Jackson ImmunoResearch公司)。
2 结果 2.1 FOXG1基因gRNA的设计和载体构建FOXG1基因不含内含子,mRNA长度为3 491 bp,其中494~1 963 bp为FOXG1基因蛋白编码序列(coding sequence, CDS),本实验设计的FOXG1-gRNA1(580~599 bp)和FOXG1-gRNA2(840~859 bp)均位于蛋白编码区,Cas9在2条gRNA的编辑位点同时发生编辑将导致基因组产生大约260 bp的片段敲除。FOXG1-gRNA1和FOXG1-gRNA2及基因型鉴定引物在FOXG1基因序列上的对应位置如图 1所示。
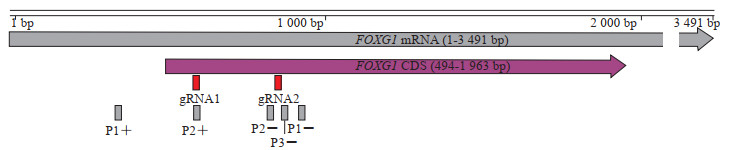
|
图 1 gRNA及基因型鉴定引物在FOXG1 mRNA序列中的位置示意图 Fig 1 Schematic illustration of gRNAs and genotyping primers in the sequence of FOXG1 mRNA gRNA: Guide RNA; FOXG1: Forkhead box G1; CDS: Coding sequence |
2.2 利用CRISPR/Cas9技术建立FOXG1基因大片段敲除的hESC
共挑取21个单克隆进行基因型PCR鉴定。使用的PCR引物P1+和P1-可以扩增涵盖2条gRNA所在区域的FOXG1基因组片段,未发生大片段敲除的hESC的基因组PCR产物大小为600 bp,而FOXG1的2个等位基因均发生了大片段敲除的hESC的基因组PCR产物大小约340 bp。PCR结果显示克隆9(C9)的基因组PCR产物为340 bp,可能FOXG1的2个等位基因均发生大片段敲除;6个克隆(C4、C10、C11、C13、C15、C20)的基因组PCR产物为600 bp,提示这些克隆可能仅发生单gRNA编辑或未发生编辑;12个克隆(C1、C3、C5、C6、C7、C8、C12、C16、C17、C18、C19、C21)基因组PCR产物同时含有340 bp和600 bp的条带,提示这些克隆可能仅在1条染色体发生了大片段敲除,或克隆不纯;1个克隆(C2)的基因组PCR产物小于340 bp,提示该克隆的1条染色体发生了更大片段的敲除(图 2A)。克隆C9的PCR产物测序结果表明存在2种基因型,基因型1缺失了266 bp,导致FOXG1基因在ATG后第31个密码子提前终止;基因型2缺失了260 bp,导致FOXG1基因在ATG后第33个密码子提前终止(图 2B)。
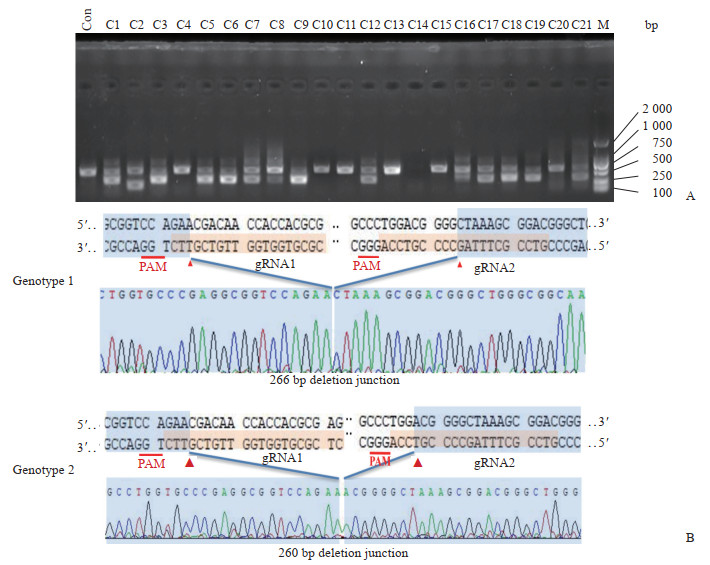
|
图 2 用CRISPR/Cas9技术建立FOXG1基因大片段敲除的hESC Fig 2 Establishment of hESCs with FOXG1 gene large fragment knockout by CRISPR/Cas9 A: Twenty-one colonies (C1-C21) of hESCs were genotyped by PCR after pX459 FOXG1-gRNA1 and pX459 FOXG1-gRNA2 co-transfection and subsequential puromycin selection; B: The genotypes of clone 9 (C9) were further confirmed by sequencing. CRISPR: Clustered regularly interspaced short palindromic repeat; Cas9: CRISPR-associated protein 9; FOXG1: Forkhead box G1; hESC: Human embryonic stem cell; Con: Control (hESCs without FOXG1 gene large fragment knockout); M: Marker; gRNA: Guide RNA; PAM: Protospacer adjacent motif |
2.3 FOXG1基因缺失不影响hESC的多能性和早期神经诱导
在体外,C9细胞仍然维持了hESC正常的克隆样生长,细胞免疫荧光染色显示C9细胞仍然表达人PSC的标志分子OCT4和SOX2(图 3A)。接下来我们利用C9细胞研究FOXG1敲除后对人PSC体外神经诱导的影响。我们采用2种神经诱导条件(SD条件和SDL条件)诱导C9细胞和对照hESC(H1细胞,pX459空载体转染H1后挑取的对照克隆)的神经分化。将C9细胞和对照H1细胞在SD和SDL条件下诱导7 d后,细胞免疫荧光染色表明诱导前的H1细胞不表达FOXG1,H1细胞诱导后有明显FOXG1的表达;C9细胞诱导后无FOXG1的表达,再次验证FOXG1基因被成功敲除(图 3A、3B)。但是PAX6和SOX2的表达在C9细胞与对照H1细胞间无明显差异(图 3A、3B)。蛋白质印迹分析结果证明FOXG1在C9细胞中被敲除(图 3C)。
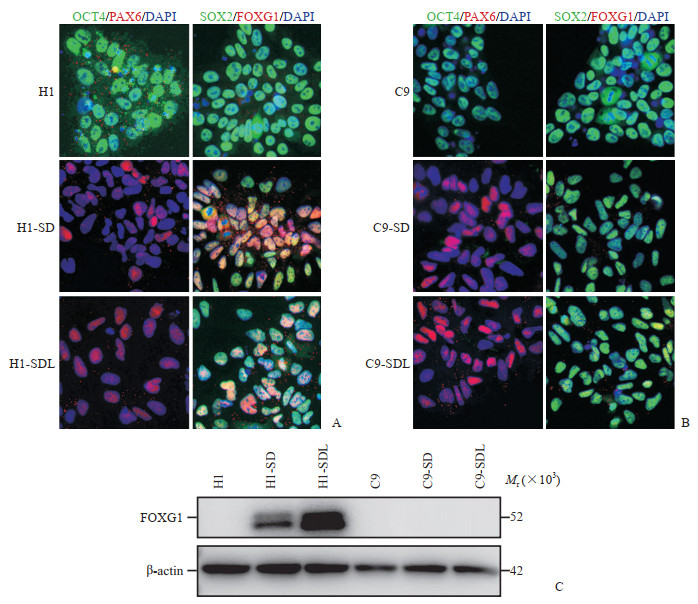
|
图 3 C9细胞和对照hESC(H1细胞)神经诱导前后相关标志物的细胞免疫荧光染色及蛋白质印迹分析结果 Fig 3 Immunofluorescence staining and Western blotting analysis of related markers in C9 cells and control hESCs (H1 cells) before and after neural induction A: Expression of OCT4, PAX6, SOX2 and FOXG1 was analyzed by immunofluorescence staining in H1 cells before and after neural induction; B: Expression of OCT4, PAX6, SOX2 and FOXG1 was analyzed by immunofluorescence staining in C9 cells before and after neural induction; C: Expression of FOXG1 protein was analyzed by Western blotting in H1 cells and C9 cells after neural induction. C9: Clone 9; hESC: Human embryonic stem cell; OCT4: Octamer-binding transcription factor 4; SOX2: Sex-determining region Y-box 2; PAX6: Paired box 6; DAPI: 4', 6-diamidino-2-phenylindole; FOXG1: Forkhead box G1; SD: SB431542+DMH1; SDL: SB431542+DMH1+LGK974. Original magnification: ×200 (A, B) |
qRT-PCR分析结果(图 4)证明FOXG1敲除并未显著影响神经诱导后PAX6、SOX2和OTX2 mRNA的表达,表明FOXG1基因敲除可能并不影响人PSC的神经发生。
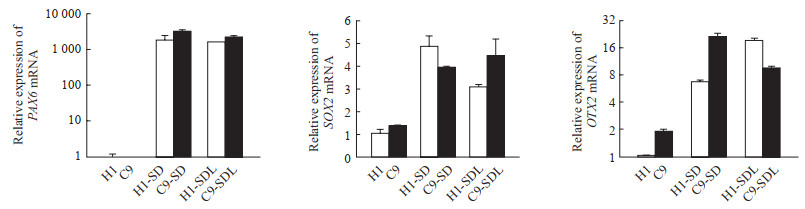
|
图 4 C9细胞和对照hESC(H1细胞)神经诱导前后相关标志物的qRT-PCR分析 Fig 4 qRT-PCR analysis of related markers in C9 cells and control hESCs (H1 cells) before and after neural induction C9: Clone 9; hESC: Human embryonic stem cell; qRT-PCR: Quantitative real-time polymerase chain reaction; PAX6: Paired box 6; SOX2: Sex-determining region Y-box 2; OTX2: Orthodenticle homeobox 2; SD: SB431542+DMH1; SDL: SB431542+DMH1+LGK974. n=3, x±s |
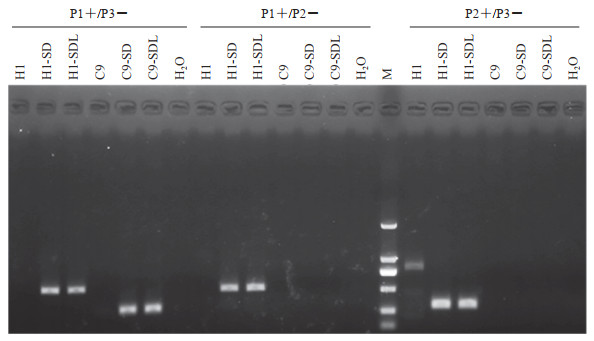
进一步使用3对引物对诱导后细胞FOXG1基因表达进行了RT-PCR检测,引物位置见图 1。引物组合P1+/P3-在gRNA敲除片段的两侧,RT-PCR结果显示诱导前的C9细胞和对照hESC(H1细胞)中均不表达FOXG1;而神经诱导后均可以检测到FOXG1的表达,H1细胞的PCR产物为550 bp,但C9细胞FOXG1的扩增片段显著截短,PCR产物大小为240 bp。引物P2+和P2-均位于2条gRNA的编辑区域内,使用引物组合P1+/P2-和P2+/P3-进行RT-PCR,仅能在神经诱导后的H1细胞中检测到FOXG1的表达条带,大小分别为500 bp和300 bp(图 5)。结果进一步证明C9克隆发生了FOXG1基因的大片段剪切编辑。
|
图 5 C9细胞和对照hESC(H1细胞)神经诱导前后FOXG1基因表达的RT-PCR分析 Fig 5 RT-PCR analysis of FOXG1 gene expression in C9 cells and control hESCs (H1 cells) before and after neural induction C9: Clone 9; hESC: Human embryonic stem cell; FOXG1: Forkhead box G1; RT-PCR: Reverse transcription polymerase chain reaction; M: Marker; SD: SB431542+DMH1; SDL: SB431542+DMH1+LGK974 |
3 讨论
利用CRISPR/Cas9系统进行基因编辑时,既可以采用单个gRNA进行编辑,也可以用多个gRNA进行片段敲除。我们的实践表明,使用多个gRNA共转染进行大片段的敲除编辑可以更方便、快速地建立基因敲除的人PSC系。使用单个gRNA进行基因编辑时,被编辑的碱基数通常较少,无法通过PCR扩增条带的大小筛选发生编辑的克隆;需要对PCR产物变性、退火后,再经T7E1内切酶识别并切割错配的杂合DNA双链来鉴定是否发生基因编辑;该方法也无法确定2个等位基因是否均发生编辑,需要对大量T7E1切割阳性的克隆进行测序验证[8]。而使用2个gRNA共转染进行基因编辑,因可诱导靶位点的大片段序列敲除,基因编辑情况可以方便地通过PCR产物大小进行判断,可以直接筛选出等位基因同时被编辑的克隆。通过该方法,我们可以高度可控地在4周内获得特定基因敲除的人PSC系。
既往研究发现FOXG1基因敲除小鼠出生后即死亡,大脑半球发育异常、体积较野生型小鼠显著减小,尤其是腹侧前脑几乎缺失[6]。本研究借助人PSC研究FOXG1基因在体外神经诱导和分化中的作用,利用改进的双重Smad抑制诱导人PSC的神经分化,该方法可以在7 d内高度均一地将人PSC诱导分化为类似于发育过程中的神经板细胞,但是结果显示FOXG1基因敲除并没有显著影响SOX2、PAX6和CTX2等基因的表达。SOX2既是PSC的标志分子,也是早期神经发生的关键基因;转录因子PAX6是人神经外胚层最早表达的核心分子[10];OTX2是前中脑的标志分子。发生这种体外和体内基因缺失后表型不吻合的可能原因是体外二维的神经诱导环境无法忠实地模拟体内三维的组织发生过程,因此有必要利用PSC的神经类器官分化体系进一步研究FOXG1对三维条件下神经诱导的影响。此外,FOXG1基因敲除是否会影响后期神经板细胞在神经发育前后轴和背腹轴上的区域化及神经元的分化有待进一步研究。
| [1] |
JINEK M, CHYLINSKI K, FONFARA I, HAUER M, DOUDNA J A, CHARPENTIER E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337: 816-821. DOI:10.1126/science.1225829 |
| [2] |
GARNEAU J E, DUPUIS M È, VILLION M, ROMERO D A, BARRANGOU R, BOYAVAL P, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J]. Nature, 2010, 468: 67-71. DOI:10.1038/nature09523 |
| [3] |
BARRANGOU R, FREMAUX C, DEVEAU H, RICHARDS M, BOYAVAL P, MOINEAU S, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315: 1709-1712. DOI:10.1126/science.1138140 |
| [4] |
CONG L, RAN F A, COX D, LIN S, BARRETTO R, HABIB N, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339: 819-823. DOI:10.1126/science.1231143 |
| [5] |
ARIANI F, HAYEK G, RONDINELLA D, ARTUSO R, MENCARELLI M A, SPANHOL-ROSSETO A, et al. FOXG1 is responsible for the congenital variant of Rett syndrome[J]. Am J Hum Genet, 2008, 83: 89-93. DOI:10.1016/j.ajhg.2008.05.015 |
| [6] |
XUAN S, BAPTISTA C A, BALAS G, TAO W, SOARES V C, LAI E. Winged helix transcription factor BF-1 is essential for the development of the cerebral hemispheres[J]. Neuron, 1995, 14: 1141-1152. DOI:10.1016/0896-6273(95)90262-7 |
| [7] |
HANASHIMA C, LI S C, SHEN L, LAI E, FISHELL G. Foxg1 suppresses early cortical cell fate[J]. Science, 2004, 303: 56-59. DOI:10.1126/science.1090674 |
| [8] |
RAN F A, HSU P D, WRIGHT J, AGARWALA V, SCOTT D A, ZHANG F. Genome engineering using the CRISPR-Cas9 system[J]. Nat Protoc, 2013, 8: 2281-2308. DOI:10.1038/nprot.2013.143 |
| [9] |
CHAMBERS S M, FASANO C A, PAPAPETROU E P, TOMISHIMA M, SADELAIN M, STUDER L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling[J]. Nat Biotechnol, 2009, 27: 275-280. DOI:10.1038/nbt.1529 |
| [10] |
ZHANG X Q, HUANG C T, CHEN J, PANKRATZ M T, XI J J, LI J, et al. Pax6 is a human neuroectoderm cell fate determinant[J]. Cell Stem Cell, 2010, 7: 90-100. DOI:10.1016/j.stem.2010.04.017 |
 2020, Vol. 41
2020, Vol. 41


