2. 海军军医大学(第二军医大学)长海医院感染科, 上海 200433;
3. 海军军医大学(第二军医大学)长海医院呼吸与危重症医学科, 上海 200433
2. Department of Infectious Diseases, Changhai Hospital, Naval Medical University (Second Military Medical University), Shanghai 200433, China;
3. Department of Respiratory and Critical Care Medicine, Changhai Hospital, Naval Medical University (Second Military Medical University), Shanghai 200433, China
新型冠状病毒肺炎(coronavirus disease 2019,COVID-19)于2019年12月被报道[1-3],并迅速发展为全球大流行。国际病毒分类委员会将引起此次疫情的新型冠状病毒命名为严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)。中国科学院武汉病毒研究所石正丽团队[1]从早期的5例患者样本中获得了该病毒的全基因组序列,来自5例患者的病毒序列相似性达到99.9%。尽管该病毒全基因组与严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus,SARS-CoV)/BJ01株的序列一致性为79.5%,但被作为冠状病毒分类标准的开放阅读框(open reading frame,ORF)1ab中的7个保守的复制酶结构域与SARS-CoV氨基酸序列一致性高达94.6%,提示该新型冠状病毒与SARS-CoV属于SARS相关冠状病毒(SARS-related coronavirus,SARSr-CoV)家族。SARS-CoV-2基因组序列与该实验室之前从中华菊头蝠分离到的1株蝙蝠冠状病毒毒株RaTG13基因组的序列一致性为96.2%,这提示其储存宿主可能为蝙蝠。SARS-CoV-2侵入宿主细胞所依赖的受体与SARS-CoV相同,也是血管紧张素转化酶Ⅱ(angiotensin-converting enzyme 2,ACE2)。由于ACE2的表达分布,这2种冠状病毒主要感染有纤毛的支气管上皮细胞和人肺泡Ⅱ型上皮细胞。多个报道提示穿山甲可能为SARS-CoV-2的中间宿主,但可能不是唯一的中间宿主[4-5]。
COVID-19疫情严重威胁人类健康及社会、经济发展,虽然目前中国国内疫情已得到控制,但中东、欧洲、北美地区的疫情仍呈暴发态势。面对COVID-19全球暴发的严峻态势,建立SARS-CoV-2感染的细胞模型及动物模型对于加快防治产品的研发进程、研判未来疫情进展及长期结局非常迫切。从感染者标本中分离、培养出病毒对于研究病毒的病原学特征、感染与致病机制、抗病毒药物与疫苗研发至关重要。为此,在我校生物安全三级(P3)实验室获得SARS-CoV-2实验活动资质以后,我们迅速开展了SARS-CoV-2的分离、鉴定,成功从2例COVID-19确诊患者鼻/咽拭子样本中分离出SARS-CoV-2,为后续研究提供了重要支撑。
1 材料和方法 1.1 细胞、试剂、抗体兔抗SARS-CoV-2刺突蛋白(spike protein,S蛋白)S1亚基单克隆抗体、核衣壳蛋白(nucleocapsid protein,N蛋白)单克隆抗体为北京义翘神州科技有限公司产品,SARS-CoV-2(ORF1ab/N基因)荧光探针定量试剂盒为上海伯杰生物医药有限公司产品,青链霉素双抗、Alexa Fluor®488标记抗兔及抗人IgG为美国Thermo Fisher公司产品,Vero E6细胞由复旦大学张荣研究员馈赠。
1.2 临床样本鼻/咽拭子样本来源于2020年1月至2月海军军医大学(第二军医大学)长海医院收治的COVID-19疑似患者,由本实验室用qRT-PCR法从送检的COVID-19疑似患者鼻/咽拭子样本中检测出SARS-CoV-2核酸阳性样本3份。3份用于SARS-CoV-2分离的样本以每管200 μL分装,冻存于-80 ℃冰箱中。2份COVID-19患者恢复期血清样本分别取自江苏大学附属镇江第三人民医院收治的2例确诊COVID-19患者(在患者治疗康复出院3周后采集)。健康成年人血清取自本实验室人员。所有样本提供者均签署知情同意书。
1.3 病毒分离病毒分离全过程均在海军军医大学(第二军医大学)P3实验室中进行。病毒分离方法参考复旦大学上海医学院张荣研究员及中国科学院武汉病毒研究所石正丽研究员的相关方法[1, 6]。Vero E6细胞采用含10% FBS的DMEM培养液(含1%青链霉素双抗)于5% CO2、37 ℃、饱和湿度的孵箱中培养待用。
将Vero E6接种于96孔细胞培养板,待细胞汇合度为90%左右时加入鼻/咽拭子样本进行病毒分离。将冻存于-80 ℃冰箱的鼻/咽拭子样本取出,置于4 ℃冰箱融化,然后取出置于冰上,加入等体积的DMEM培养液(含2 μg/mL TPCK胰酶、1%青链霉素双抗和0.2%牛血清白蛋白),再置于37 ℃孵箱,1 h后吸取鼻/咽拭子上清,加入接种有Vero E6细胞的96孔板,每孔50 μL。将96孔板置于孵箱中,2 h后吸除上清,用PBS轻轻洗孔3次,随后加入100 μL含适量TPCK胰酶的无血清DMEM培养液,置于孵箱中继续培养。24 h后每天观察细胞是否出现细胞病变效应(cytopathic effect,CPE),待超过50%细胞出现CPE并从孔底面脱落时,吸取上清,避免碰触细胞残骸,然后高速离心,小心吸取10 μL上清传代感染接种于24孔板中的Vero E6细胞,取20 μL上清用qRT-PCR法检测SARS-CoV-2核酸。
1.4 SARS-CoV-2核酸检测用磁珠法全自动核酸提取仪(杭州奥盛仪器有限公司)从细胞培养上清中抽提病毒RNA,溶解于50 μL无核酸酶水,然后用SARS-CoV-2(ORF1ab/N基因)荧光探针定量试剂盒检测SARS-CoV-2核酸。
1.5 SARS-CoV-2 S基因受体结合区(receptor binding domain,RBD)区的扩增和测序取5 μL抽提的病毒RNA,用高保真反转录试剂(美国Promega公司)进行反转录反应,反转录引物用随机引物。得到的cDNA用高保真DNA聚合酶(日本TaKaRa公司)进行PCR扩增,引物序列:S773 5′-GGACAGCTGGTGCTGCAGC-3′,S1718 5′-GTAGTGTCAGCAATGTCTCTGC-3′。扩增产物经琼脂糖凝胶电泳,切出分子大小与预期946 bp相符的DNA条带,回收后测序。引物合成和PCR产物测序均由上海博尚生物技术有限公司完成。
1.6 免疫荧光检测与细胞病变观察将病毒分离第1代细胞上清接种于24孔板中的Vero E6细胞,超过50%细胞出现CPE时,取细胞培养上清,高速离心去除细胞残骸,再取4 μL连续2倍稀释,接种于96孔板中的Vero E6细胞。待最高剂量病毒感染孔超过50%细胞出现CPE时,将96孔板浸泡于甲醇中,置于-20 ℃冰箱灭活病毒并固定细胞30 min。随后取出96孔板,显微镜下观察细胞并拍照,然后用免疫荧光法检测病毒蛋白。一抗包括兔抗SARS-CoV-2 S蛋白S1亚基单克隆抗体、兔抗SARS-CoV-2 N蛋白单克隆抗体和2份COVID-19患者恢复期血清,均以1:200稀释。用健康成年人血清作为COVID-19患者恢复期血清的阴性对照。荧光二抗使用Alexa Fluor®488标记的抗兔及抗人IgG。荧光抗体反应以后,用DAPI进行细胞核染色。用全自动细胞成像及分析系统(BioTek Cytation 5 Imaging Reader,美国BioTek公司)进行观察、拍照。
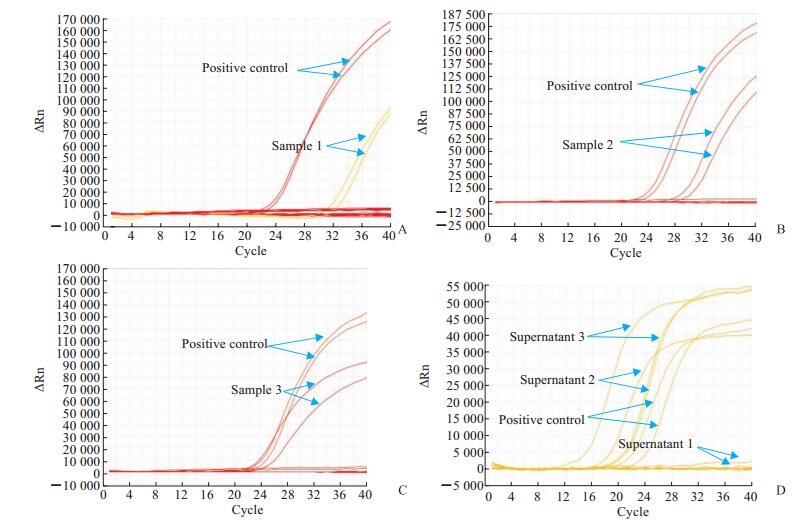
2 结果 2.1 病毒分离培养的核酸检测将3份鼻/咽拭子样本分别接种Vero E6细胞,接种24 h后每隔12 h观察细胞CPE,2号和3号样本接种孔的Vero E6细胞分别在接种后60 h和72 h出现明显CPE,大量细胞脱落。1号样本接种孔及阴性对照孔细胞未观察到明显CPE。至接种后的84 h和96 h,2号和3号样本接种孔中仅有少量贴壁细胞。分别取20 μL上清抽提核酸,用qRT-PCR法检测SARS-CoV-2 ORF1ab/N基因。如图 1所示,2号和3号样本接种孔分别在PCR第14.8个和第10.5个循环时开始出现ORF1ab基因扩增曲线[即循环阈值(cycle threshold,Ct)分别为14.8、10.5],N基因扩增曲线起峰稍晚;而1号样本接种孔和阴性对照孔均未出现扩增曲线。这与细胞上清中的病毒核酸载量高低相符。
|
图 1 鼻/咽拭子样本及鼻/咽拭子样本接种的Vero E6培养上清中SARS-CoV-2 ORF1ab/N基因的qRT-PCR扩增曲线 Fig 1 qRT-PCR amplification curves of SARS-CoV-2 ORF1ab/N sequence of nasal/throat swab samples and cell culture supernatants of Vero E6 cells cultured with nasal/throat swab samples A-C: Amplification curves of nasal/throat swab samples 1, 2 and 3, respectively; D: Amplification curve of cell culture supernatants of Vero E6 cells inoculated with nasal/throat swab samples 1, 2 and 3. For each sample tested, the first emerging peak curve is the ORF1ab gene amplification curve, and the next is the N gene amplification curve. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; ORF: Open reading frame; qRT-PCR: Quantitative real-time polymerase chain reaction; ΔRn: Baseline-corrected normalized reporter |
2.2 病毒分离培养的细胞病变观察及免疫荧光检测
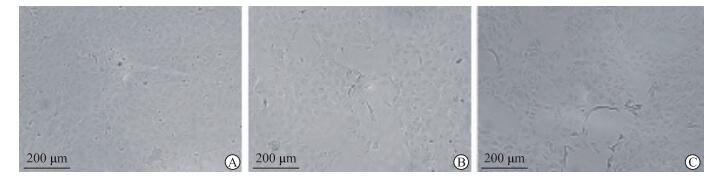
将经接种于24孔板的Vero E6细胞再次扩增的病毒上清连续2倍稀释感染接种于96孔板中的Vero E6细胞,待最高剂量病毒感染孔超过50%细胞出现CPE时,用甲醇灭活病毒并固定细胞,于显微镜下观察细胞形态,并拍照记录细胞病变。如图 2所示,从2号和3号2份鼻/咽拭子样本分离出的病毒在感染后12 h即可致使细胞出现明显CPE。分别以2种单克隆抗体及2份COVID-19患者恢复期患者血清为检测抗体,用免疫荧光法检测SARS-CoV-2蛋白。这些抗体均可与从2份鼻/咽拭子样本中分离出的病毒感染的Vero E6细胞结合,不与未感染病毒的Vero E6细胞结合,健康成人血清不与病毒感染的Vero E6细胞结合(图 3为从2号鼻/咽拭子样本中分离出的病毒的免疫荧光检测结果)。由此可以确定,从2号和3号鼻/咽拭子样本中分别分离出了SARS-CoV-2,下文分别称为S2、S3株SARS-CoV-2。
|
图 2 SARS-CoV-2感染的Vero E6细胞的细胞病变效应 Fig 2 The cytopathic effect of Vero E6 cells infected with SARS-CoV-2 A: Mock (cell supernatant from normal Vero E6 cells) infected cells; B: Cells infected by virus from nasal/throat swab sample 2; C: Cells infected by virus from nasal/throat swab sample 3. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2 |

|
图 3 免疫荧光检测SARS-CoV-2蛋白 Fig 3 Assay of SARS-CoV-2 proteins using immunofluorescence technique A: Convalescent serum 1; B: Convalescent serum 2; C: Serum of a healthy adult; D: Rabbit anti-N monoclonal antibody; E: Rabbit anti-S1 monoclonal antibody. The left panel shows Vero E6 cells infected with SARS-CoV-2 isolated from nasal/throat swab sample 2, and the right panel shows cells without virus infection. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; N: Nucleocapsid protein; S1: Spike protein S1 subunit |
2.3 病毒S基因RBD区测序
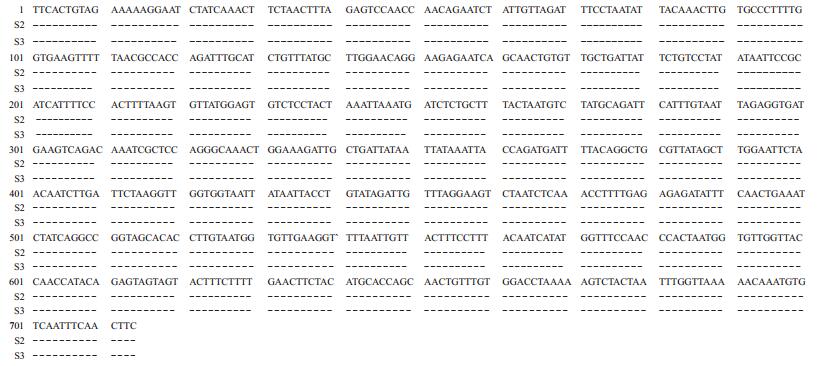
用反转录PCR分别扩增S2、S3株SARS-CoV-2 S基因的RBD区序列,测序结果如图 4所示,2株病毒的S基因RBD区序列与最早完成基因组测序的SARS-CoV-2病毒株(GenBank登录号:NC_045512)RBD区序列一致。
|
图 4 2株SARS-CoV-2与最早测序的SARS-CoV-2病毒株(GenBank登录号:NC_045512)S基因RBD区序列的比对 Fig 4 Sequence alignment of S gene RBD between two SARS-CoV-2 strains and the earliest sequenced SARS-CoV-2 strain (GenBank accession: NC_045512) S2 and S3: SARS-CoV-2 isolated from nasal/throat swab sample 2 and 3. SARS-CoV-2: Severe acute respiratory syndrome coronavirus 2; S: Spike protein; RBD: Receptor binding domain |
3 讨论
冠状病毒属于套式病毒目(Nidovirales)冠状病毒科(Coronaviridae)冠状病毒属(Coronavirus),是一类有包膜的单股正链RNA病毒,基因组长约30 kb,是基因组最长的RNA病毒[7]。根据冠状病毒基因组结构和系统发生学分析,冠状病毒科可分为α、β、γ和δ 4个属,α和β属的冠状病毒一般感染哺乳类动物和人,γ和δ属的冠状病毒主要感染鸟类。除SARS-CoV-2外,尚有6种可感染人类的冠状病毒:人冠状病毒(human coronavirus,HCoV)-229E、HCoV-OC43、HCoV-NL63、HCoV-HKU1、SARS-CoV和中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus,MERS-CoV)[8-9]。HCoV-229E、HCoV-OC43、HCoV-NL63和HCoV-HKU1致病性较低,一般仅引起轻微的呼吸道症状。而SARS-CoV、MERS-CoV及引起本次疫情的SARS-CoV-2均为高致病性冠状病毒,能够引起人类严重下呼吸道感染[8-9]。该3种病毒均属于β属冠状病毒。
冠状病毒膜表面的S蛋白以三聚体形式存在,可分为S1和S2两个结构域。S1介导病毒与宿主细胞膜表面受体的结合,S2介导病毒包膜与宿主细胞之间的膜融合。在游离的病毒颗粒表面,S蛋白并未被加工为S1和S2两个独立亚基,而是一条完整的多肽[10-12]。在病毒侵入宿主细胞时,S蛋白被宿主细胞表面的蛋白酶切割为S1和S2两个亚基,病毒的感染性显著增强[10-12]。这一特性对于病毒的分离培养具有重要意义,细胞培养液中的动物血清可抑制宿主细胞蛋白酶活性,从而影响病毒的细胞侵入效率。
我们将3份鼻/咽拭子样本分别接种Vero E6细胞,从病毒核酸载量较高的2号和3号鼻/咽拭子样本中成功分离出SARS-CoV-2,而1号鼻/咽拭子样本接种的Vero E6细胞经过5 d培养及上清盲传2代均未检测到病毒复制。其原因除了与1号鼻/咽拭子样本病毒载量相对较低有关以外,还可能与1号鼻/咽拭子的采集和处理方式与2号、3号鼻/咽拭子样本不同有关。1号鼻/咽拭子样本使用干脱脂棉拭子采集,用含10% FBS的DMEM培养液对鼻/咽拭子进行吹洗,以洗脱鼻/咽拭子上的黏膜上皮细胞和病毒颗粒。而2号和3号鼻/咽拭子样本则使用无血清病毒采样管进行采集。3个样本的运输和保存条件一致。我们推测,1号样本中含有的FBS抑制了Vero E6细胞表面的蛋白酶活性,从而不利于病毒的细胞侵入,这也可能与病毒分离失败有关。我们在病毒培养过程中发现,相对于无血清培养基,在含有5% FBS的培养基中病毒复制速度明显降低,引起CPE的时间也明显延迟。这也提示鼻/咽拭子样本中存在的FBS不利于病毒分离。
我们通过用qRT-PCR检测病毒核酸、观察病毒引起的细胞病变、用免疫荧光方法检测病毒蛋白、对病毒S基因RBD区进行扩增和测序等方法,确认从2例COVID-19患者鼻/咽拭子样本中分别分离出SARS-CoV-2。这为开展SARS-CoV-2病毒学、感染与致病机制研究及检测、预防、治疗技术与产品的研发提供了重要支撑。
志谢 感谢复旦大学上海医学院张荣研究员、易志刚副研究员为病毒分离提供宝贵建议并馈赠Vero E6细胞,感谢中国疾病预防控制中心谭文杰研究员为病毒分离提供宝贵建议,感谢在本实验室学习和工作过的朱分禄博士和王路博士提供个人生物防护帮助。
| [1] |
ZHOU P, YANG X L, WANG X G, HU B, ZHANG L, ZHANG W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 579: 270-273. DOI:10.1038/s41586-020-2012-7 |
| [2] |
ZHU N, ZHANG D, WANG W, LI X, YANG B, SONG J, et al. A novel coronavirus from patients with pneumonia in China, 2019[J]. N Engl J Med, 2020, 382: 727-733. DOI:10.1056/NEJMoa2001017 |
| [3] |
WU F, ZHAO S, YU B, CHEN Y M, WANG W, SONG Z G, et al. A new coronavirus associated with human respiratory disease in China[J]. Nature, 2020, 579: 265-269. DOI:10.1038/s41586-020-2008-3 |
| [4] |
KANDEEL M, IBRAHIM A, FAYEZ M, AL-NAZAWI M. From SARS and MERS CoVs to SARS-CoV-2: moving toward more biased codon usage in viral structural and nonstructural genes[J/OL]. J Med Virol, 2020. doi: 10.1002/jmv.25754.
|
| [5] |
ZHANG T, WU Q, ZHANG Z. Probable pangolin origin of SARS-CoV-2 associated with the COVID-19 outbreak[J/OL]. Curr Biol, 2020, 30: 1346-1351.e2. doi: 10.1016/j.cub.2020.03.022.
|
| [6] |
张荣, 易志刚, 王玉燕, 滕峥, 徐巍, 宋武慧, 等. 新型冠状病毒上海株(nCoV-SH01)的分离和鉴定[J]. 微生物与感染, 2020, 15: 16-21. DOI:10.3969/j.issn.1673-6184.2020.01.004 |
| [7] |
BRIAN D A, BARIC R S. Coronavirus genome structure and replication[J]. Curr Top Microbiol Immunol, 2005, 287: 1-30. |
| [8] |
FORNI D, CAGLIANI R, CLERICI M, SIRONI M. Molecular evolution of human coronavirus genomes[J]. Trends Microbiol, 2017, 25: 35-48. DOI:10.1016/j.tim.2016.09.001 |
| [9] |
闻玉梅. 冠状病毒的致病性及防控[J]. 微生物与感染, 2020, 15: 3-6. |
| [10] |
SCHOEMAN D, FIELDING B C. Coronavirus envelope protein: current knowledge[J/OL]. Virol J, 2019, 16: 69. doi: 10.1186/s12985-019-1182-0.
|
| [11] |
LI F. Structure, function, and evolution of coronavirus spike proteins[J]. Annu Rev Virol, 2016, 3: 237-261. DOI:10.1146/annurev-virology-110615-042301 |
| [12] |
BELOUZARD S, MILLET J K, LICITRA B N, WHITTAKER G R. Mechanisms of coronavirus cell entry mediated by the viral spike protein[J]. Viruses, 2012, 4: 1011-1033. DOI:10.3390/v4061011 |
 2020, Vol. 41
2020, Vol. 41


