2. 重庆医科大学附属儿童医院儿科学重庆市重点实验室, 重庆 400014;
3. 重庆医科大学附属儿童医院泌尿外科, 重庆 400014;
4. 重庆医科大学附属儿童医院儿童发育疾病研究教育部重点实验室, 重庆 400014;
5. 重庆医科大学附属儿童医院儿童发育重大疾病国家国际科技合作基地, 重庆 400014;
6. 重庆医科大学附属儿童医院国家儿童健康与疾病临床医学研究中心, 重庆 400014
2. Chongqing Key Laboratory of Pediatrics, Children's Hospital of Chongqing Medical University, Chongqing 400014, China;
3. Department of Urology, Children's Hospital of Chongqing Medical University, Chongqing 400014, China;
4. Key Laboratory of Child Development and Disorders of Ministry of Education, Children's Hospital of Chongqing Medical University, Chongqing 400014, China;
5. China International Science and Technology Cooperation Base of Child Development and Critical Disorders, Children's Hospital of Chongqing Medical University, Chongqing 400014, China;
6. National Clinical Research Center for Child Health and Disorders, Children's Hospital of Chongqing Medical University, Chongqing 400014, China
Argonaute家族的Piwi(P-element-induced wimpy testis)蛋白主要在各种生物的生殖干细胞或胚胎源性/性腺肿瘤组织中广泛表达,其与Piwi互作RNA(Piwi-interacting RNA,piRNA)相互作用,维持生殖细胞中关键保守基因的稳定[1]。Piwi家族蛋白Piwi样蛋白(P-element-induced wimpy testis like,Piwil)2和Piwil4及piRNA已被证实在多种肿瘤组织中存在差异表达,但是不同肿瘤中piRNA的表达水平及分布不同[2-4],发现更多piRNA并明确其在肿瘤中的表达情况有助于肿瘤的诊断和治疗[5]。
恶性肿瘤的发生、耐药和转移均与肿瘤组织中存在少量肿瘤干细胞(cancer stem cell,CSC)有关[6-7]。目前的研究一般是基于干细胞标志物从实体肿瘤中分离获得CSC,但在CSC的提取和培养技术上仍存在相当大的困难。本课题组前期通过Piwil2基因重编程人成纤维细胞(fibroblast,FB)建立了肿瘤样干细胞[8]。本研究以该肿瘤样干细胞作为体外培养的CSC模型,通过高通量测序技术筛选Piwil2调控的差异表达piRNA并预测其靶基因,分析差异表达的piRNA及其靶基因在儿童肾母细胞瘤中的表达情况及临床意义,为临床诊断及治疗肾母细胞瘤提供新的分子标志物。
1 材料和方法 1.1 主要仪器与试剂CO2培养箱和超速离心机(美国Thermo公司),恒温水浴箱(上海医疗器械厂),细胞培养皿(美国HyClone公司),移液器(德国Eppendorf公司),荧光显微镜(日本Nikon公司)。FBS(美国HyClone公司),F-12培养液(美国Gibco公司),嘌呤霉素(美国Sigma公司),TRIzol试剂(美国Thermo公司),miRNA反转录第一链试剂盒和miRNA qRT-PCR试剂盒[天根生化科技(北京)有限公司],mRNA反转录第一链试剂盒(日本TaKaRa公司),mRNA qRT-PCR试剂盒(德国Qiagen公司)。
1.2 细胞培养本课题组前期将携带Piwil2-GFP标记的慢病毒及空载目的基因的GFP标记的慢病毒分别感染原代培养的小儿包皮FB,分别命名为Piwil2-FB、GFP-FB,经细胞行为学及体外细胞染色体核型分析证实Piwil2-FB为Piwil2诱导的肿瘤样干细胞[8],并长期储存于液氮中。将Piwil2-FB、GFP-FB从-80 ℃冰箱中取出,快速在37 ℃水浴箱中复温,将细胞悬液移至10 mL EP管中,加入2 mL F-12培养液,离心清洗后分别培养于含10% FBS的F-12培养液中(10 cm培养皿),在37 ℃、5%CO2培养箱中培养。细胞每隔48 h进行换液,待生长至80%以上融合度时用于实验。
1.3 临床标本收集重庆医科大学附属儿童医院泌尿外科2015年6月至2019年6月入院治疗的肾母细胞瘤患儿的手术标本,均为首次行肿瘤切除术并经病理科2位医师证实为肾母细胞瘤组织。本研究获得重庆医科大学附属儿童医院医学伦理委员会批准,且标本均在患儿监护人知情同意后获取。标本来自34例儿童患者,男15例、女19例,年龄为4.0~104.0(29.9±24.2)个月;依照美国国家肾母细胞瘤研究组(National Wilms’Tumor Study Group,NWTS)-5标准[9]:组织分化良好27例、分化不良7例,分期Ⅰ期7例、Ⅱ期9例、Ⅲ期11例、Ⅳ期1例、Ⅴ期6例。取每例患儿的肿瘤组织作为肿瘤组,并取其癌旁组织(距离肿瘤≥3 cm的正常肾脏组织)作为对照组织,Ⅴ期双侧肿瘤则取肿瘤同侧的正常肾脏组织。
1.4 高通量测序及靶基因预测送检将两组细胞样本(Piwil2-FB、GFP-FB)送上海惠研生物科技有限公司进行高通量测序,Piwil2-FB作为实验组、GFP-FB作为对照组。cDNA文库构建及高通量测序由上海惠研生物科技有限公司完成,差异基因的筛选标准为P<0.05且差异倍数≥1.5,通过miRanda算法(http://www.microrna.org/microrna/getDownloads.do)对这些差异基因进行靶基因预测,并通过David(https://david.ncifcrf.gov/home.jsp)对靶基因进行基因本体(gene ontology,GO)功能分析。
1.5 总RNA提取、反转录及qRT-PCR采用TRIzol法提取细胞及组织的总RNA。将10 cm培养皿中的细胞用1 mL TRIzol试剂消化10 min后吸入1.5 mL去酶EP管中(组织加100 μg/mL TRIzol后用电动匀浆器充分匀浆1~2 min),加入200 μL氯仿震荡混匀后室温放置15 min,4 ℃ 12 000×g离心15 min,吸取上清加入500 μL异丙醇混匀,室温静置10 min,4 ℃ 12 000×g离心10 min后弃上清,加入1 mL 75%乙醇温和震荡,4 ℃ 8 000×g离心5 min后弃上清,即得总RNA。将总RNA真空干燥5~10 min后加入50 μL DEPC水溶解。利用NanoDrop ND-1000分光光度仪对总RNA进行质量检验,D260/D280为1.8~2.0;进一步行琼脂糖凝胶电泳,可见28S、18S 2条亮带。使用miRNA反转录试剂盒和mRNA反转录试剂盒将总RNA反转录为cDNA,采用miRNA qRT-PCR试剂盒和mRNA qRT-PCR试剂盒对组织进行检测。piRNA及其靶基因的引物由生工生物工程(上海)股份有限公司设计合成,引物序列和退火温度见表 1。
|
|
表 1 piRNA和靶基因的引物序列 Tab 1 Primers of piRNAs and target genes |
1.6 统计学处理
应用SPSS 21.0及Graphpad Prism7.0软件进行统计学分析和作图。非正态分布的数据描述采用中位数(下四分位数,上四分位数)表示,通过Wilcoxon检验比较配对样本中目的基因的表达差异,采用Mann-Whitney检验比较目的基因在不同年龄、性别、临床分期和病理分型的肿瘤组织中的差异。检验水准(α)为0.05。
2 结果 2.1 Piwil2-FB中piRNA表达谱及差异piRNA从Piwil2-FB中筛选出230个差异表达piRNA,对比The piRNA database数据库(http://www.regulatoryrna.org/database/piRNA/),其中180个为已发现且报道过的piRNA(46个在人类基因中有过报道,134个在非人类生物中有过报道),50个为未曾报道的新piRNA。230个差异表达piRNA中,106个表达量在Piwil2-FB中显著上调(其中22个未知,上调未知用NU表示),124个表达量显著下调(其中28个未知,下调未知用ND表示)。50个差异表达的未知piRNA的聚类分析见图 1。
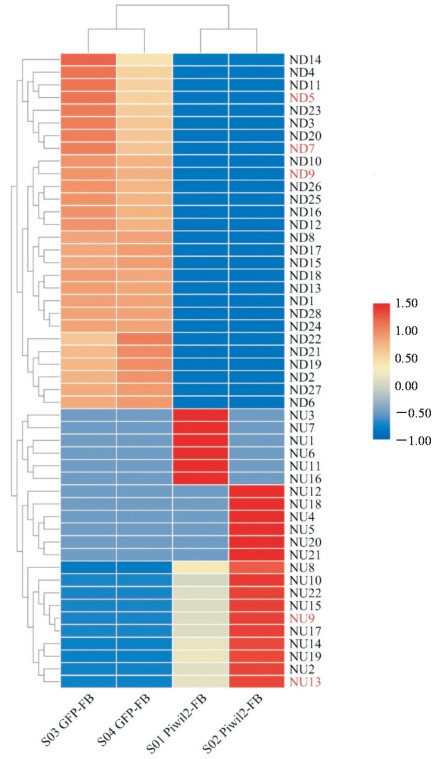
|
图 1 Piwil2诱导的肿瘤样干细胞中50个差异表达的未知piRNA的聚类分析图 Fig 1 Heatmap of 50 differentially expressed unknown piRNAs in Piwil2-induced cancer stem-like cells Piwil2: Piwi like 2; piRNA: Piwi-interacting RNA; Piwi: P-element-induced wimpy testis; NU: Unknown upregulated piRNA; ND: Unknown downregulated piRNA; S01/S02 Piwil2-FB: Piwil2-green fluorescent protein reprogramming human fibroblast; S03/S04 GFP-FB: Green fluorescent protein reprogramming human fibroblast |
2.2 piRNA靶基因预测及富集分析
通过miRanda算法对230个差异表达基因进行靶基因预测,共61个piRNA预测出靶基因,预测出的靶基因共43个。对这43个靶基因进行GO分析(P<0.05)(https://david.ncifcrf.gov/home.jsp),发现其参与的生物学过程主要为调节细胞钙离子浓度,分子功能主要为参与ATP酶的活动及多聚A尾RNA结合(图 2)。通过String数据库(www.string-db.org)对其进行蛋白互作网络分析(互作分数≥0.15),其中肌球蛋白IXA(myosin IXA,MYO9A)有8个相互作用蛋白,成蛋白2(formin 2,FMN2)有7个互作蛋白,蛋白酪氨酸磷酸酶D型受体(protein tyrosine phosphatase receptor-type delta,PTPRD)有6个互作蛋白,NOP56核糖核蛋白(NOP56 ribonucleoprotein,NOP56)有4个互作蛋白,40S核糖体蛋白S8(40S ribosomal protein S8,RPS8)有3个互作蛋白,相互作用综合分数最高的组合是NOP56和RPS8(0.994)。见图 3。靶基因MT-RNR2样蛋白1(MT-RNR2 like protein 1,MTRNR2L1)虽然没有在蛋白互作网络中,但是在差异表达量前10位的下调未知piRNA中有3个piRNA靶向它。
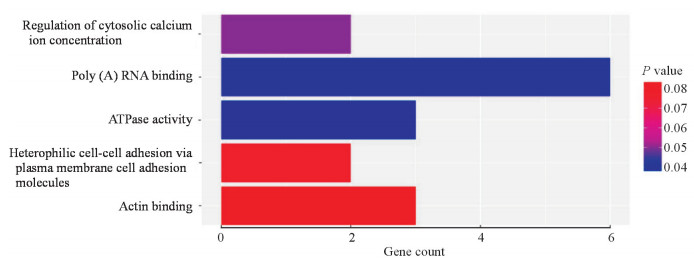
|
图 2 Piwil2诱导的肿瘤样干细胞中差异表达piRNA的预测靶基因的GO分析 Fig 2 GO functional classification of predicted target genes of differentially expressed piRNAs in Piwil2-induced cancer stem-like cells Piwil2: Piwi like 2; piRNA: Piwi-interacting RNA; Piwi: P-element-induced wimpy testis; GO: Gene ontology |
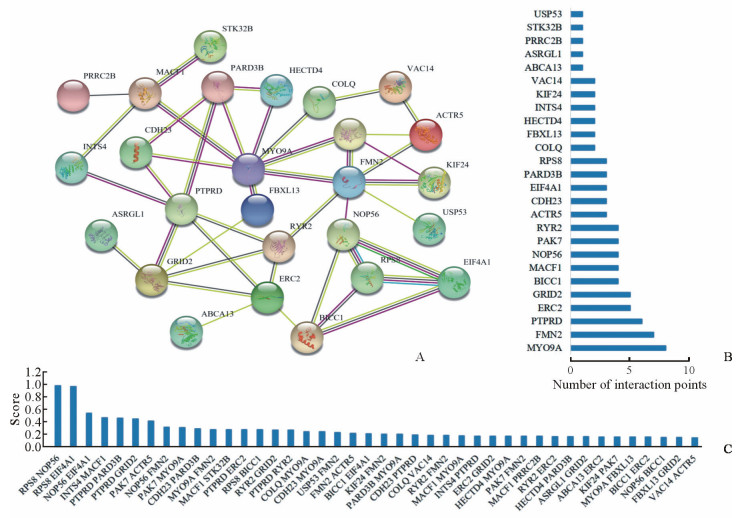
|
图 3 Piwil2诱导的肿瘤样干细胞中差异表达piRNA的预测靶基因的蛋白互作分析 Fig 3 Protein interaction analysis of predicted target genes of differentially expressed piRNAs in Piwil2-induced cancer stem-like cells A: Gene interaction network; B: Interaction points; C: Combined score. Piwil2: Piwi like 2; piRNA: Piwi-interacting RNA; Piwi: P-element-induced wimpy testis |
2.3 piRNA及其靶基因在肾母细胞瘤患儿肿瘤组织及癌旁组织中的表达
选择piRNA NU13(未知的上调差异表达piRNA第13位)及其靶基因NOP56,piRNA NU9(未知的上调差异表达piRNA第9位)及其靶基因RPS8,piRNA ND5、ND7、ND9(未知的下调差异表达piRNA第5、7、9位)及其靶基因MTRNR2L1,在34例肾母细胞瘤患儿组织中进行qRT-PCR检测。结果(图 4)显示,在肿瘤组织中,NU13表达量下调(P<0.01),其靶基因NOP56的表达与癌旁正常组织差异无统计学意义(P=0.58);NU9表达量下调(P<0.01),其靶基因RPS8的表达与癌旁正常组织差异无统计学意义(P=0.29);而ND5、ND7、ND9及其靶基因MTRNR2L1的表达量均下调(P均<0.01)。其中NOP56和RPS8在肿瘤组织中的表达量与患儿年龄和性别有关(P均<0.05),其余基因的表达量与患儿年龄、性别、肿瘤临床分期、病理分型并无相关性(P均>0.05)。见表 2。
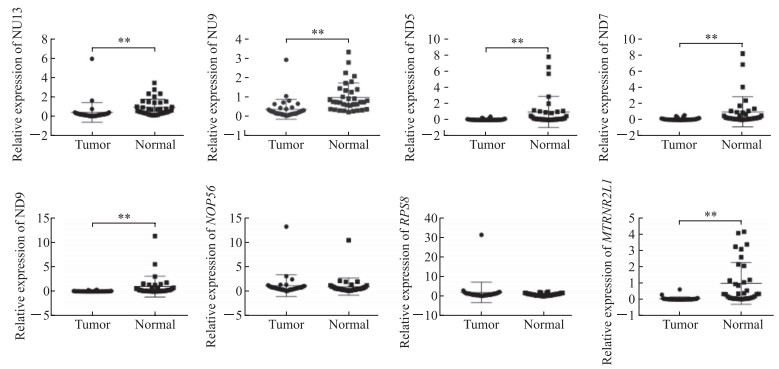
|
图 4 儿童肾母细胞瘤组织中piRNA及其预测靶基因的表达 Fig 4 Expression of piRNAs and their predicted target genes in childhood nephroblastoma piRNA: Piwi-interacting RNA; Piwi: P-element-induced wimpy testis; NU: Unknown upregulated piRNA; ND: Unknown downregulated piRNA; MTRNR2L1: MT-RNR2 like protein 1; NOP56: NOP56 ribonucleoprotein; RPS8: 40S ribosomal protein S8. **P < 0.01. n=34, median (lower quartile, upper quartile) |
|
|
表 2 儿童肾母细胞瘤组织中piRNA及其预测靶基因表达与临床病理参数的关系 Tab 2 Correlation between expression of piRNAs and their predicted target genes and clinicopathological parameters in childhood nephroblastoma |
3 讨论
CSC具有致癌性、多重分化、自我更新等特性,有学者认为其是肿瘤复发转移及抵抗放射治疗和化学治疗的根本原因[10],探究其起源及参与其发生过程中的标志物有助于更好地判断肿瘤预后及更精准地进行靶向治疗。本课题组前期用Piwil2基因重编程人FB建立了稳定表达Piwil2-GFP标记的Piwil2-FB,经细胞生物行为学测试及CSC标志物检测证实重编程的细胞为肿瘤样干细胞[8],可以作为理想的体外CSC细胞模型。
piRNA是长度为23~31 nt的小分子非编码RNA,特征是在5'端常见单磷酸的尿嘧啶,而在3'端有甲基化修饰[11]。piRNA与Piwi蛋白(包括Piwil2和Piwil4)结合后,通过乒乓效应抑制转座子的功能而起到稳定基因的作用,此外piRNA还可通过DNA甲基化与组蛋白修饰参与基因调节[12-14]。在Piwil2基因重编程FB构建的体外CSC模型中,我们推测Piwil2基因过表达所致的相关piRNA表达量及功能变化可能在肿瘤发生过程中具有重要作用,其差异表达的piRNA可能对肿瘤组织的发生及对肿瘤的诊治和预后判断有重要价值。
本实验通过高通量测序技术从Piwil2-FB筛选出差异表达piRNA 230个,通过miRanda算法预测出靶基因共43个。对筛选出的差异表达piRNA的靶基因进行GO分析发现其参与的生物学过程主要为调节细胞钙离子浓度,分子功能主要为参与ATP酶的活动及多聚A尾RNA结合。利用String数据库进行蛋白互作网络分析,结果显示靶基因中NOP56和RPS8的相互作用综合分数最高(0.994)且在网络中有较多相互作用蛋白(4个、3个),同时GO分析发现NOP56与RPS8也富集于多聚A尾RNA结合。靶基因MTRNR2L1虽然未出现于此蛋白互作网络中,但在表达量前10位的下调未知piRNA中有3个piRNA靶向它。
肾母细胞瘤作为儿童中最常见的恶性肿瘤,目前临床上还没有高特异性的诊断及预后相关标志物或治疗靶点[15-16]。通过生物信息学筛选,本研究将piRNA NU13及其靶基因NOP56,piRNA NU9及其靶基因RPS8,piRNA ND5、ND7、ND9及其靶基因MTRNR2L1在34例肾母细胞瘤患儿组织中进行qRT-PCR检测,结果显示在肿瘤组织中以上5个piRNA及靶基因MTRNR2L1的表达量下调(P均<0.01)。本研究检测的5个piRNA均在肿瘤组织中下调,虽然NU9与NU13的靶基因表达未见明显差异,但其依然可能通过其他多种途径参与肿瘤的发生。许多研究已报道了piRNA参与肿瘤发生的途径,最常见的是通过联合Piwi蛋白来调控转座子的功能从而影响肿瘤的发生[17]。此外,piRNA及piRNA/Piwi复合体也可通过靶向某些蛋白编码基因来调节肿瘤的生长,如招募DNA甲基转移酶(DNA methyltransferase,DNMT)来调节DNA的甲基化等[18]。因此,推测本实验中的piRNA表达量下调可能导致转座子功能失调,进而促进肿瘤的发生,但具体机制仍有待进一步研究。本研究结果表明piRNA NU9、NU13、ND5、ND7、ND9在肾母细胞瘤的肿瘤组织与癌旁正常组织中的表达差异显著,可以作为区分恶性肿瘤与正常组织的潜在标志物;然而它们的表达情况与肿瘤临床分期、病理分型之间并无相关性,是否可以作为肿瘤患者预后判断标志物还需进一步研究。
对43个靶基因进行蛋白互作网络分析,发现了其中位置较为重要且蛋白互作评分与可信度最高的互作蛋白NOP56与RPS8。NOP56参与核糖体RNA前体核糖甲基化修饰,是小分子核仁核糖核蛋白复合体的组成之一[19]。研究表明NOP56异常与Burkitt淋巴瘤的Myc基因突变有关[20],近期也有研究发现乳腺癌的发生与NOP56的上调有关[21]。RPS8作为40S核糖体亚基的组成部分,其确切功能尚未明确,有研究显示,RPS8与CDK11p46协同作用可以抑制cap-和IRES依赖的翻译过程,降低或过表达RPS8可通过抑制翻译使荧光素酶的活性降低[22]。String数据库显示RPS8参与有关翻译的通路,且其上调可促进NOP56的表达与功能。本研究中靶基因NOP56和RPS8的表达在肿瘤组织及癌旁正常组织中并无明显差异,但两者在肿瘤组织中的表达与年龄及性别有关,推测其功能可能与机体的发育相关,需要加大样本量且剔除年龄与性别因素才可能发现其在肿瘤中真实的表达情况。MTRNR2L1基因是编码humanin蛋白的MTRNR2基因的众多核拷贝之一,可能由线粒体DNA转录而来,有研究发现其与胰岛素抵抗有关[23],它所编码的humanin蛋白具有神经保护活性和抗凋亡作用,同时也可直接作用于线粒体促进ATP的生成[24-26]。许多研究显示肿瘤及其干细胞在能量代谢方面存在异常,糖酵解是肿瘤及其干细胞的主要产能方式,同时相比正常细胞其线粒体数量明显下降[27-28]。本研究结果显示MTRNR2L1在肾母细胞瘤的肿瘤组织中表达下调,推测CSC可能通过调控piRNA从而间接调控正常细胞中MTRNR2L1或其他MTRNR2样基因的表达,从而下调humanin蛋白来改变正常细胞的细胞周期及线粒体活动,使其向肿瘤细胞分化。另一种可能的原因是肾母细胞瘤肿瘤组织中的线粒体含量降低[29],导致线粒体相关基因的表达量都有所下调。
综上所述,本研究通过高通量测序对肿瘤样干细胞中piRNA及其靶基因的表达与功能进行了初步探索,初步证明差异表达的piRNA NU9、NU13、ND5、ND7、ND9对鉴别儿童肾母细胞瘤的肿瘤组织与正常肾组织具有参考价值,但这些piRNA是否真正调控其预测的靶向基因及这些piRNA在其他肿瘤中是否也有诊断价值,仍需进一步的细胞实验和更广的疾病谱进行验证。
| [1] |
TÓTH K F, PEZIC D, STUWE E, WEBSTER A. The piRNA pathway guards the germline genome against transposable elements[J]. Adv Exp Med Biol, 2016, 886: 51-77. |
| [2] |
LIU J, ZHANG S, CHENG B. Epigenetic roles of PIWI-interacting RNAs (piRNAs) in cancer metastasis (review)[J]. Oncol Rep, 2018, 40: 2423-2434. |
| [3] |
HIRAKATA S, SIOMI M C. piRNA biogenesis in the germline: from transcription of piRNA genomic sources to piRNA maturation[J]. Biochim Biophys Acta, 2016, 1859: 82-92. DOI:10.1016/j.bbagrm.2015.09.002 |
| [4] |
ZENG G, ZHANG D, LIU X, KANG Q, FU Y, TANG B, et al. Co-expression of Piwil2/Piwil4 in nucleus indicates poor prognosis of hepatocellular carcinoma[J]. Oncotarget, 2017, 8: 4607-4617. |
| [5] |
ZHANG H, REN Y, XU H, PANG D, DUAN C, LIU C. The expression of stem cell protein Piwil2 and piR-932 in breast cancer[J]. Surg Oncol, 2013, 22: 217-223. DOI:10.1016/j.suronc.2013.07.001 |
| [6] |
EUN K, HAM S W, KIM H. Cancer stem cell heterogeneity: origin and new perspectives on CSC targeting[J]. BMB Rep, 2017, 50: 117-125. DOI:10.5483/BMBRep.2017.50.3.222 |
| [7] |
PEIRIS-PAGÈS M, MARTINEZ-OUTSCHOORN U E, PESTELL R G, SOTGIA F, LISANTI M P. Cancer stem cell metabolism[J/OL]. Breast Cancer Res, 2016, 18: 55. doi: 10.1186/s13058-016-0712-6.
|
| [8] |
ZHANG D, WU X, LIU X, CAI C, ZENG G, ROHOZINSKI J, et al. Piwil2-transfected human fibroblasts are cancer stem cell-like and genetically unstable[J]. Oncotarget, 2017, 8: 12259-12271. |
| [9] |
GROSFELD J L, O'NEILL J A Jr, FONKALSRUD E W, CORAN A G.小儿外科学[M].6版.吴晔明, 译.北京: 北京大学医学出版社, 2008: 467-468.
|
| [10] |
SUN J, LI G, LIU Y, MA M, SONG K, LI H, et al. Targeting histone deacetylase SIRT1 selectively eradicates EGFR TKI-resistant cancer stem cells via regulation of mitochondrial oxidative phosphorylation in lung adenocarcinoma[J]. Neoplasia, 2020, 22: 33-46. DOI:10.1016/j.neo.2019.10.006 |
| [11] |
ROJAS-RÍOS P, SIMONELIG M. piRNAs and PIWI proteins: regulators of gene expression in development and stem cells[J/OL]. Development, 2018, 145. pii: dev161786. doi: 10.1242/dev.161786.
|
| [12] |
OZATA D M, GAINETDINOV I, ZOCH A, O'CARROLL D, ZAMORE P D. PIWI-interacting RNAs: small RNAs with big functions[J]. Nat Rev Genet, 2019, 20: 89-108. DOI:10.1038/s41576-018-0073-3 |
| [13] |
刘启鹏, 安妮, 岑山, 李晓宇. piRNA抑制基因转座的分子机制[J]. 遗传, 2018, 40: 445-450. DOI:10.3760/cma.j.issn.1673-4386.2018.06.002 |
| [14] |
YIN J, JIANG X Y, QI W, JI C G, XIE X L, ZHANG D X, et al. piR-823 contributes to colorectal tumorigenesis by enhancing the transcriptional activity of HSF1[J]. Cancer Sci, 2017, 108: 1746-1756. DOI:10.1111/cas.13300 |
| [15] |
陈天拓, 何晶晶, 苏义林, 朱凯. 肿瘤干细胞标志物CD133和CD44在肾母细胞瘤中的表达及意义[J]. 安徽医学, 2018, 39: 1194-1198. DOI:10.3969/j.issn.1000-0399.2018.10.007 |
| [16] |
SHUKRUN R, PODE-SHAKKED N, PLENICEANU O, OMER D, VAX E, PEER E, et al. Wilms' tumor blastemal stem cells dedifferentiate to propagate the tumor bulk[J]. Stem Cell Reports, 2014, 3: 24-33. DOI:10.1016/j.stemcr.2014.05.013 |
| [17] |
LIU Y, DOU M, SONG X, DONG Y, LIU S, LIU H, et al. The emerging role of the piRNA/piwi complex in cancer[J/OL]. Mol Cancer, 2019, 18: 123. doi: 10.1186/s12943-019-1052-9.
|
| [18] |
FU A, JACOBS D I, HOFFMAN A E, ZHENG T, ZHU Y. PIWI-interacting RNA 021285 is involved in breast tumorigenesis possibly by remodeling the cancer epigenome[J]. Carcinogenesis, 2015, 36: 1094-1102. |
| [19] |
LYKKE-ANDERSEN S, ARDAL B K, HOLLENSEN A K, DAMGAARD C K, JENSEN T H. Box C/D snoRNP autoregulation by a cis-acting snoRNA in the NOP56 pre-mRNA[J/OL]. Mol Cell, 2018, 72: 99-111.e5. doi: 10.1016/j.molcel.2018.08.017.
|
| [20] |
COWLING V H, TURNER S A, COLE M D. Burkitt's lymphoma-associated c-Myc mutations converge on a dramatically altered target gene response and implicate Nol5a/Nop56 in oncogenesis[J]. Oncogene, 2014, 33: 3519-3527. DOI:10.1038/onc.2013.338 |
| [21] |
曲杰, 林萍萍, 吕喜英, 吴练练, 李青山. NOP56在乳腺癌组织中的表达情况及对临床预后的意义[J]. 生物信息学, 2019, 17: 122-130. DOI:10.12113/j.issn.1672-5565.201806002 |
| [22] |
HAO Y, KONG X, RUAN Y, GAN H, CHEN H, ZHANG C, et al. CDK11p46 and RPS8 associate with each other and suppress translation in a synergistic manner[J]. Biochem Biophys Res Commun, 2011, 407: 169-174. DOI:10.1016/j.bbrc.2011.02.132 |
| [23] |
VEILLEUX A, MAYEUR S, BÉRUBÉ J C, BEAULIEU J F, TREMBLAY E, HOULD F S, et al. Altered intestinal functions and increased local inflammation in insulin-resistant obese subjects: a gene-expression profile analysis[J/OL]. BMC Gastroenterol, 2015, 15: 119. doi: 10.1186/s12876-015-0342-y.
|
| [24] |
BODZIOCH M, LAPICKA-BODZIOCH K, ZAPALA B, KAMYSZ W, KIEC-WILK B, DEMBINSKA-KIEC A. Evidence for potential functionality of nuclearly-encoded humanin isoforms[J]. Genomics, 2009, 94: 247-256. DOI:10.1016/j.ygeno.2009.05.006 |
| [25] |
XIAO J, KIM S J, COHEN P, YEN K. Humanin: functional interfaces with IGF-I[J]. Growth Horm IGF Res, 2016, 29: 21-27. DOI:10.1016/j.ghir.2016.03.005 |
| [26] |
殷博凯, 郭艳芹, 何治. Humanin及其衍生肽保护作用的研究进展[J]. 生命科学, 2019, 31: 940-944. |
| [27] |
PALORINI R, VOTTA G, BALESTRIERI C, MONESTIROLI A, OLIVIERI S, VENTO R, et al. Energy metabolism characterization of a novel cancer stem cell-like line 3AB-OS[J]. J Cell Biochem, 2014, 115: 368-379. DOI:10.1002/jcb.24671 |
| [28] |
AMINZADEH S, VIDALI S, SPERL W, KOFLER B, FEICHTINGER R G. Energy metabolism in neuroblastoma and Wilms tumor[J]. Transl Pediatr, 2015, 4: 20-32. |
| [29] |
FEICHTINGER R G, NEUREITER D, ROYER-POKORA B, MAYR J A, ZIMMERMANN F A, JONES N, et al. Heterogeneity of mitochondrial energy metabolism in classical triphasic Wilms' tumor[J]. Front Biosci (Elite Ed), 2011, 3: 187-193. |
 2020, Vol. 41
2020, Vol. 41


