2. 个体化药物治疗四川省重点实验室, 四川省医学科学院·四川省人民医院药学部, 成都 610072
2. Personalized Drug Therapy of Key Laboratory of Sichuan Province, Department of Pharmacy, Sichuan Academy of Medical Sciences·Sichuan Provincial People's Hospital, Chengdu 610072, Sichuan, China
加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)适用于复杂的多样本转录组数据,常用于研究不同器官、组织类型和不同阶段的发育调控、生物和非生物胁迫的不同时间点响应机制等。其依据基因在不同样品中的表达模式相似性可得到基因集合,还可以通过分析基因模块与样品或表型数据之间的相关性得到与某个样品或某表型高度相关的基因集合,最后通过模块内部基因关联分析得到模块内部的关键基因。WGCNA的分析流程为:构建基因表达相关性矩阵→根据基因表达相似性对基因进行层次聚类建树,并划分成不同的基因模块→根据基因模块的特征值与临床性状相关联→计算基因模块内部连接度,确定核心基因[1]。
乳腺癌的发病率和死亡率在我国女性恶性肿瘤中的排序分别为第1位和第5位,全国女性乳腺癌发病率为41.82/10万,虽然在全球水平中偏低,但增速却位列首位[2]。生物标志物有助于疾病诊断、判断疾病分期,也可用来评价新药或新疗法在目标人群中的安全性和有效性。本研究基于WGCNA对乳腺癌中最普遍的类型导管癌和小叶癌进行了疾病分期和诊断年龄的生物标志物挖掘,对利用数据挖掘发现新型生物标志物这一思路进行探索和验证。
1 材料和方法 1.1 数据来源及数据预处理本研究的乳腺癌基因表达谱数据及临床信息来源于美国国立癌症研究所(National Cancer Institute,NCI)和国家人类基因组研究所(National Human Genome Research Institute,NHGRI)共同创建的癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库。在分析之前需要对下载的数据进行预处理,包括提取样本信息、构建基因表达矩阵、将探针名转化为基因名,最终获得行名为样本名、列名为基因名的矩阵用于后续分析。
1.2 共表达网络的构建与模块识别安装R软件WGCNA包,为节省计算机运算消耗的内存,本研究选取表达量方差大于所有方差四分位数的基因。剔除离群样本并确保基因表达矩阵的样品号与临床信息的样品号一一对应。按照无尺度网络的标准选择合适的加权系数β,并用此系数将相关矩阵转化为邻接矩阵,此后通过拓扑重叠(topological matrix,TOM)计算基因间的关联,基于TOM值进行层次聚类建树。建树的方法采用动态混合剪切法,将相异度作为距离测度,设定最小模块尺寸为30,进行模块识别并绘制基因树状图。计算模块的特征值,即对模块内的所有基因进行主成分分析(principal component analysis,PCA)得到的第一主成分。
1.3 与临床信息相关模块及核心基因的识别基于样本的临床信息表对模块的性状进行关联分析,寻找和性状显著相关的模块用于后续分析。采用2种方式帮助识别相关性较高的模块:计算模块的特征值与表型的相关系数(即module eigengene E,ME值)、定义基因的显著性(gene significance,GS),表征基因和表型之间的相关性,取所有基因GS绝对值的平均数即模块显著性(module significance,MS)表示该模块与表型之间的相关性。
筛选出与表型高度相关的模块后,还需要对模块下的基因进行核心基因筛选。通过计算模块中每个基因与该模块的相关系数即模块隶属度(module membership,MM)并结合GS值筛选核心基因,设定GS绝对值>0.2且其归属模块MM绝对值>0.8为核心基因。
1.4 不同人种的交叉验证候选基因模块数量较多时本研究选用另一人种进行交叉验证。数据依然从TCGA数据库中下载,基因模块的筛选同1.2和1.3项。得到的显著性基因模块与之前的显著性基因模块中所有基因取交集,即为候选基因。
1.5 候选基因的功能富集为了解候选基因的生物学功能,本研究将候选基因映射至在线网站DAVID(http://david-d.ncifcrf.gov/)中,进行基因本体(gene ontology,GO)通路分析,并设置P<0.05的条目富集程度差异有统计学意义。
1.6 候选基因表达差异分析与生存分析为验证候选基因的生物学意义,本研究采用UALCAN癌症数据(http://ualcan.path.uab.edu/index.html)进行差异表达和生存期的验证。为减少运算,只取候选基因中的核心基因输入该网站中,分析其在正常组织和乳腺癌组织中的表达是否有差异以及是否影响乳腺癌患者的生存期。
2 结果 2.1 乳腺癌样本数据筛选亚洲人种女性导管癌和小叶癌基因表达数据从TCGA数据库下载。筛选条件为:乳腺癌→TCGA→TCGA-乳腺浸润性癌→female→Asian→disease type→ductal and lobular neoplasms。筛选得到的原始数据共有56个样本,其中含导管癌47例、小叶癌8例和正常对照1例。数据经过合并和id转换(只选择编码基因)生成19 755个基因和56个样本的表达矩阵。为使基因表达矩阵和临床信息的标本号相对应,需要剔除正常对照。为减少运算时计算机消耗的内存,选取基因表达量的方差大于所有方差四分位数的4 937个基因(即选取在各个样本中变化较大的基因)进行后面的运算。基因表达矩阵应进行缺失值处理(删除缺失值较多的基因)和离群样本的剔除。临床表型数据纳入诊断年龄和肿瘤分期。根据TCGA数据库资料,肿瘤分期包含8个:stageⅠ、stageⅠa、stageⅠb、stageⅡa、stageⅡb、stageⅢa、stageⅢb、stageⅢc。根据样本聚类的距离鉴定离群样本,剔除离群样本TCGAC8A1HJ和TCGAC8A1HE,最终有53个样本纳入后续分析。见图 1。
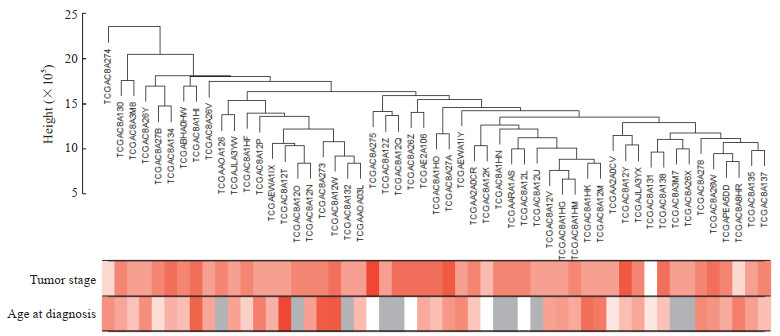
|
图 1 1纳入分析的53个样本肿瘤分期和诊断年龄的样本聚类分析热图 Fig 1 Fifty-three sample dendrogram and trait heatmap for tumor stage and age at diagnosis |
2.2 筛选软阈值
共表达网络符合无尺度网络,即出现连接度为k的节点的对数lgk与该节点出现的概率的对数lg [P(k)]呈负相关,且相关系数应>0.8。我们使用R软件WGCNA包进行构建权重共表达网络,使用分析包自动选择的软阈值计算得到软阈值β=12(图 2)。
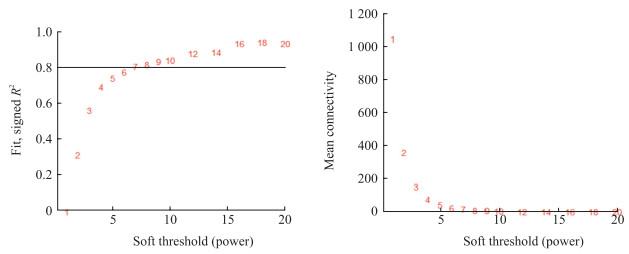
|
图 2 软阈值确定 Fig 2 Soft threshold determination |
2.3 划分基因模块
确定软阈值后,通过动态剪切树法进行模块初步识别并合并相似模块,设置每个基因网络模块最少的基因数目为30,最终得到19个模块,其中灰色模块是无法聚集到其他模块的基因集合。如图 3。
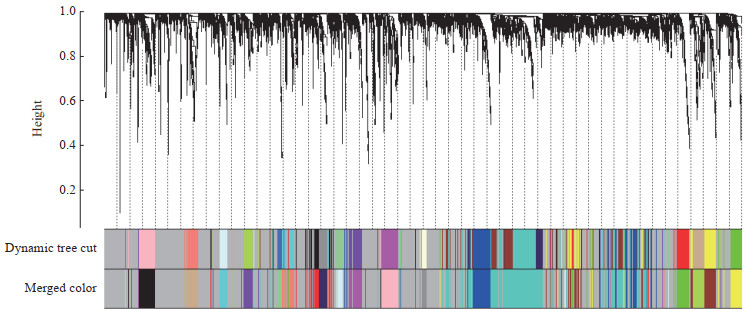
|
图 3 基因聚类树和模块划分 Fig 3 Gene cluster dendrograms and module detecting |
2.4 临床表型与基因模块的相关性
根据各个模块的特征向量,分别计算这些模块与2个表型(肿瘤分期和诊断年龄)的相关性,如图 4。结果显示,棕褐色、褐色、黄色、红色、黄绿色模块与肿瘤分期的相关性较高(P值分别为0.000 7、0.01、0.02、0.02、0.03),而粉红色、棕褐色2个模块与诊断年龄相关性较高(P值分别为0.03和0.05)。
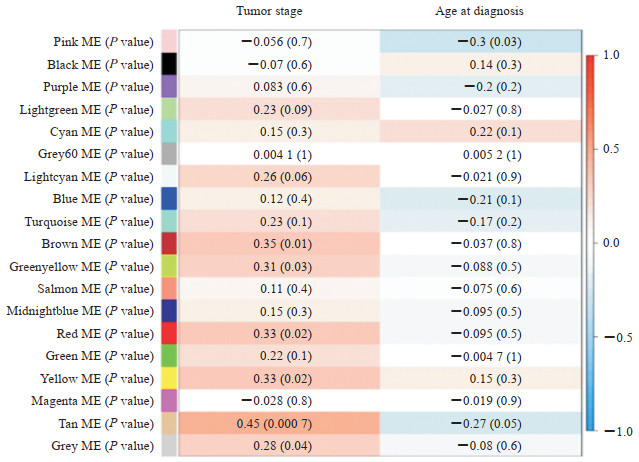
|
图 4 乳腺癌肿瘤分期和诊断年龄相关的基因模块鉴定 Fig 4 Gene module-trait relationships for cancer stage and age at diagnosis of breast cancer ME: Module eigengene E |
2.5 在非洲人种中进行重复验证
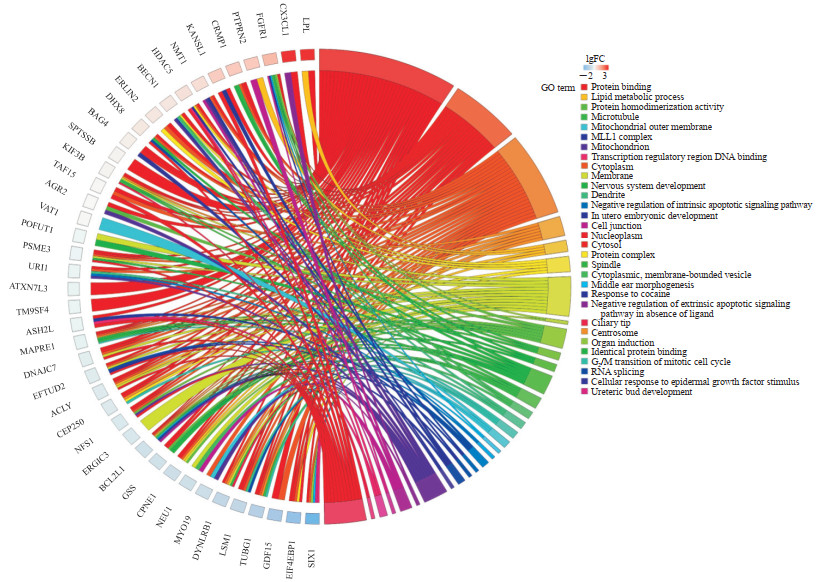
因筛选所得有显著相关的模块数量较多,我们选取另外一个人种进行交叉验证。设定人种选项选为非洲人种后下载得到156例数据,剔除正常对照和离群样本后有126例纳入分析。操作步骤同亚洲人群。分析得到3个模块skyblue1、skyblue2、saddlebrown与诊断年龄有关(P<0.05);skyblue2、mediumorchid、mediumpurple3与肿瘤分期有关(P<0.05)。将亚洲人种和非洲人种的11个差异有统计学意义的模块的基因取交集,得到42个基因。将这42个基因做GO富集发现,除了NXPH1未出现在任何通路上外,其余41个基因主要富集在分子功能(molecular function,MF)的蛋白质结合、细胞定位(cellular component,CC)的细胞质和生物过程(biological process,BP)的脂代谢中,如图 5。
|
图 5 41个候选基因GO富集弦图 Fig 5 GO enrichment analysis for 41 candidate genes GO: Gene ontology; FC: Fold change; MLL1: Mixed lineage leukemia 1 |
2.6 候选生物标志物验证
2个人群交叉验证得到的基因交集中只取模块内连接度排名前30的核心基因,得到以下9个基因:蛋白酶体激活亚单位3(proteasome activator subunit 3,PSME3)、N-肉豆蔻酰基转移酶1(N-myristoyltransferase 1,NMT1)、DEAH-box RNA解螺旋酶8(DEAH-box RNA helicase 8,DHX8)、共济失调蛋白7样3(ataxin 7-like 3,ATXN7L3)、延伸因子Tu-GTP结合域包含蛋白2(elongation factor Tu-GTP binding domain-containing protein 2,EFTUD2)、KAT8调控NSL复合物亚单位1(KAT8 regulatory NSL complex subunit 1,KANSL1)、复合素2(complexin 2,CPLX2)、内质网脂质筏相关蛋白2(endoplasmic reticulum lipid raft-associated protein 2,ERLIN2)、ASH2样组蛋白赖氨酸甲基转移酶复合物亚单位(ASH2-like histone lysine methyltransferase complex subunit,ASH2L),见表 1。
|
|
表 1 候选基因及所对应的模块、模块所属性状及是否为核心基因 Tab 1 Candidate genes and their corresponding modules, and hub gene or not |
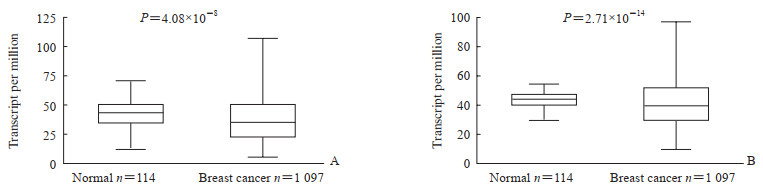
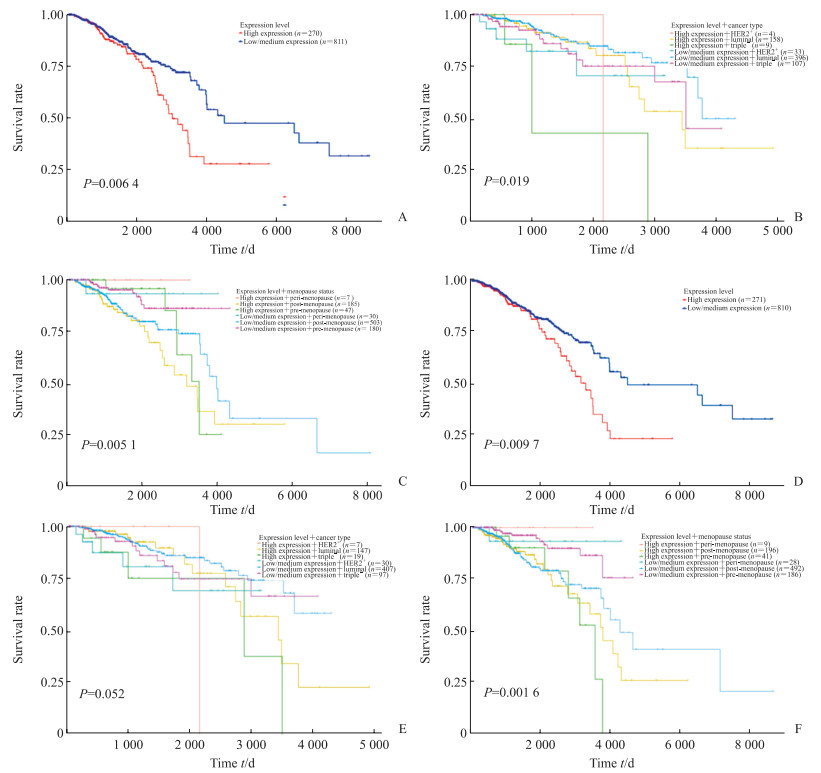
将9个基因名称分别输入UALCAN癌症数据(http://ualcan.path.uab.edu/index.html),查看该基因在正常组织和乳腺癌组织中的表达是否存在差异,以及表达的高低是否影响乳腺癌患者的生存时间。结果(图 6、图 7)显示,ERLIN2和ASH2L这2个基因不仅表达差异具有统计学意义(P<0.01),而且表达水平对生存期(包括疾病不同类型的分层)可能存在显著性影响(除图 7E的P=0.052外,其余P均<0.05),ERLIN2和ASH2L高表达人群生存期短于低表达人群, 可见ERLIN2和ASH2L低表达是保护因素。KANSL1无法被网站识别。其余6个基因表达差异虽然均有统计学意义,但对生存期的影响不显著或只对某种类型的乳腺癌或患者绝经情况有影响。由此可见,ERLIN2和ASH2L这2个基因可作为候选生物标志物用于后续大样本临床验证及机制探讨。
|
图 6 ERLIN2(A)和ASH2L(B)在正常组织和乳腺癌组织中的表达差异 Fig 6 Expression of ERLIN2(A) and ASH2L(B) in normal tissues and breast cancer tissues The data were derived from The Cancer Genome Atlas. ERLIN2: Endoplasmic reticulum lipid raft associated protein 2; ASH2L: ASH2-like histone lysine methyltransferase complex subunit. Median (lower quartile, upper quartile) |
|
图 7 ERLIN2(A~C)和ASH2L(D~F)表达水平对乳腺癌患者生存时间的影响 Fig 7 of ERLIN2 (A-C) andASH2L (D-F) expression on breast cancer patient survival The data were derived from The Cancer Genome Atlas. ERLIN2: Endoplasmic reticulum lipid raft associated protein 2; ASH2L: ASH2-like histone lysine methyltransferase complex subunit; HER2+: Human epidermal growth factor receptor 2 positive breast cancer; Triple-: Triple negative breast cancer |
3 讨论
传统生物标志物的研究一般分为3个阶段:筛选、验证和确认。筛选标志物时需要借助高通量的组学手段,对大规模的临床样本进行代谢组学或蛋白质组学测定,得到差异有统计学意义的代谢物或蛋白质,然后再将候选标志物放在更大的样本中验证[3]。随着大数据时代的到来,信息资源共享水平不断提升,利用公共数据挖掘寻找生物标志物成为可能。本研究即利用公共数据库TCGA采用WGCNA算法对乳腺癌的基因表达数据进行挖掘,筛选出生物标志物并对其生物学意义进行了验证,为生物标志物的新型筛选模式提供了一定的思路和流程。
已用于临床的乳腺癌相关分子生物标志物包括雌激素受体和孕激素受体,两者通常用于对乳腺癌进行分型、指导治疗及预后评价[4]。人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)的过表达预示着乳腺癌具有更强的浸润能力,是一种重要的预后标志物[5]。除此之外,一些新型生物标志物如长链非编码RNA、微RNA、外泌体等均与乳腺癌相关[6-8]。
WGCNA属于组学数据挖掘的高级分析,用于从高通量数据中挖掘模块信息。目前,通过WGCNA筛选核心基因,探索疾病的靶标和相关生物标志物取得了不小的进展[9-12]。本研究采用WGCNA筛选得到的核心基因ERLIN2编码内质网脂质转运相关蛋白2,属于与肿瘤分期相关性较高的基因模块,GO富集分析显示该基因的功能是蛋白相互作用。最初发现该基因与遗传性痉挛性截瘫相关[13],对其在乳腺癌中的生理功能和机制研究较少[14-17]。Zhang等[15]研究发现,该蛋白通过相互作用及稳定有丝分裂促进因子参与细胞周期的调控,并在侵袭性乳腺癌中高表达,研究显示ERLIN2的下调导致细胞周期停滞,可抑制乳腺癌的恶性增殖并增加乳腺癌细胞对抗癌药物的敏感性。这与本研究生存分析所显示的结果一致:ERLIN2 高表达乳腺癌人群生存期短于低表达人群。有研究显示微RNA-410可作用于ERLIN2使其表达下调,是一个重要的肿瘤抑制因子[14]。
ASH2L是一种特异性的H3K4甲基转移酶MLL的保守亚基,在胚胎的发育调控中扮演重要角色[18]。研究发现ASH2L在白血病来源的肿瘤细胞系中高表达,可能和血液疾病相关[19]。吴林霖[20]在体外实验中研究发现,ASH2L能够抑制胰腺癌细胞株PANC-1的凋亡,属于一种促癌基因。本研究通过数据挖掘也发现ASH2L 在乳腺癌中扮演着重要角色,值得进一步研究。
高通量技术的快速发展和成熟极大助推了生物标志物的研究。这些技术在整体层面上揭示了基因间的相互作用,展现了复杂疾病庞大的基因网络。这些基因虽然都参与疾病的发生、发展,但贡献可能非常微小,将各个网络的生物标志物代表筛选出来组成一个标志物组合,则可更加客观地反映复杂疾病的状态,是未来生物标志物筛选和检测的趋势[21]。本研究其余的6个基因虽然单独没有明显影响乳腺癌患者的生存期,但是有可能存在共同影响。总之,利用数据挖掘的方式可以省时、省力地挖掘到候选生物标志物,但差异有统计学意义并不代表有生物学意义,生物标志物是否能应用于临床还需要在实践中验证。
| [1] |
刘伟, 李立, 叶桦, 屠伟. 权重基因共表达网络分析在生物医学中的应用[J]. 生物工程学报, 2017, 33: 1791-1801. |
| [2] |
杜沛玲, 方佳英, 贾潇岳, 徐镇喜, 林昆. 1994~2013年中国女性乳腺癌流行病学特征[J]. 汕头大学医学院学报, 2016, 29: 124-126. |
| [3] |
吴昊.基于气相色谱-质谱联用技术的代谢组学研究方法在肝癌及消化道肿瘤诊断中的应用[D].上海: 复旦大学, 2010.
|
| [4] |
熊荣国, 田野, 田振. 乳腺癌预后分子生物标志物的研究进展[J]. 现代肿瘤医学, 2018, 26: 3150-3154. DOI:10.3969/j.issn.1672-4992.2018.19.039 |
| [5] |
张青, 甘淋. 乳腺癌生物标志物的研究进展[J]. 生命的化学, 2018, 38: 85-90. |
| [6] |
钟国斌, 韦薇, 周晓, 朱玲钰, 梅燕. 外泌体作为浸润性乳腺癌诊断标志物的研究进展[J]. 重庆医学, 2018, 47: 249-255. DOI:10.3969/j.issn.1671-8348.2018.02.035 |
| [7] |
刘夏, 浦春, 黄丽珠. 乳腺癌相关miRNA的研究进展[J]. 临床输血与检验, 2017, 19: 413-416. DOI:10.3969/j.issn.1671-2587.2017.04.030 |
| [8] |
宋宏伟, 丛辉. ADAMTS9-AS2在乳腺癌患者血浆中的表达及其意义[J]. 检验医学与临床, 2019, 16: 464-466,470. DOI:10.3969/j.issn.1672-9455.2019.04.009 |
| [9] |
LI Q, CHEN W, SONG M, CHEN W, YANG Z, YANG A. Weighted gene co-expression network analysis and prognostic analysis identifies hub genes and the molecular mechanism related to head and neck squamous cell carcinoma[J]. Cancer Biol Ther, 2019, 20: 750-759. DOI:10.1080/15384047.2018.1564560 |
| [10] |
MAIND A, RAUT S. Identifying condition specific key genes from basal-like breast cancer gene expression data[J]. Comput Biol Chem, 2019, 78: 367-374. DOI:10.1016/j.compbiolchem.2018.12.022 |
| [11] |
ZHANG Y, LI H, ZHANG W, CHE Y, BAI W, HUANG G. LASSO-based Cox-PH model identifies an 11-lncRNA signature for prognosis prediction in gastric cancer[J]. Mol Med Rep, 2018, 18: 5579-5593. |
| [12] |
LIU J, JING L, TU X. Weighted gene co-expression network analysis identifies specific modules and hub genes related to coronary artery disease[J/OL]. BMC Cardiovasc Disord, 2016, 16: 54. doi: 10.1186/s12872-016-0217-3.
|
| [13] |
RYDNING S L, DUDESEK A, RIMMELE F, FUNKE C, KRÜGER S, BISKUP S, et al. A novel heterozygous variant in ERLIN2 causes autosomal dominant pure hereditary spastic paraplegia[J/OL]. Eur J Neurol, 2018, 25: 943-e71. doi: 10.1111/ene.13625.
|
| [14] |
WU H, LI J, GUO E, LUO S, WANG G. MiR-410 acts as a tumor suppressor in estrogen receptor-positive breast cancer cells by directly targeting ERLIN2 via the ERS pathway[J]. Cell Physiol Biochem, 2018, 48: 461-474. DOI:10.1159/000491777 |
| [15] |
ZHANG X, CAI J, ZHENG Z, POLIN L, LIN Z, DANDEKAR A, et al. A novel ER-microtubule-binding protein, ERLIN2, stabilizes cyclin B1 and regulates cell cycle progression[J/OL]. Cell Discov, 2015, 1: 15024. doi: 10.1038/celldisc.2015.24.
|
| [16] |
WANG G, ZHANG X, LEE J S, WANG X, YANG Z Q, ZHANG K. Endoplasmic reticulum factor ERLIN2 regulates cytosolic lipid content in cancer cells[J]. Biochem J, 2012, 446: 415-425. DOI:10.1042/BJ20112050 |
| [17] |
WANG G, LIU G, WANG X, SETHI S, ALI-FEHMI R, ABRAMS J, et al. ERLIN2 promotes breast cancer cell survival by modulating endoplasmic reticulum stress pathways[J/OL]. BMC Cancer, 2012, 12: 225. doi: 10.1186/1471-2407-12-225.
|
| [18] |
DOU Y, MILNE T A, RUTHENBURG A J, LEE S, LEE J W, VERDINE G L, et al. Regulation of MLL1 H3K4 methyltransferase activity by its core components[J]. Nat Struct Mol Biol, 2006, 13: 713-719. DOI:10.1038/nsmb1128 |
| [19] |
WANG J, ZHOU Y, YIN B, DU G, HUANG X, LI G, et al. ASH2L:alternative splicing and downregulation during induced megakaryocytic differentiation of multipotential leukemia cell lines[J]. J Mol Med (Berl), 2001, 79: 399-405. DOI:10.1007/s001090100222 |
| [20] |
吴林霖.胰腺癌中ASH2L和KDM4B基因的功能分析及临床意义[D].上海: 复旦大学, 2013.
|
| [21] |
范月蕾, 陈大明, 于建荣. 生物标志物研究进展与应用趋势[J]. 生命的化学, 2013, 33: 344-351. |
 2019, Vol. 40
2019, Vol. 40


