2. 上海交通大学附属儿童医院儿童精准医学大数据工程技术研究中心, 上海 200040
2. Big Data Eengineering and Technology Research Center for Pediatric Precision Medicine, Children's Hospital of Shanghai Jiao Tong University, Shanghai 200040, China
骨龄是骨骼年龄的简称,它是将人体骨骼实际发育程度与标准发育程度进行比较所得的发育年龄。骨龄在很大程度上代表了人的真正发育水平[1],人类骨骼的发育过程具有连续性,因此,骨龄评估能准确反映个体的生长发育水平和成熟程度[2]。通过骨龄可以及早了解儿童的生长发育潜力和性成熟趋势[3]。骨龄评估还有助于儿科内分泌疾病的诊断,同时其对一些身材矮小的患者的治疗具有指导意义[4]。
传统的骨龄评估通常是对被测者的左手和腕部进行X线片检查,然后由医师对X线片进行解读。骨龄测定方法有简单计数法、图谱法、评分法和计算机骨龄评分系统等,目前临床上基于手腕骨X线图像的儿童骨龄评估方法主要包括G-P图谱法(Greulich and Pyle atlas method)和TW(Tanner Whitehouse)计分法[1]。G-P图谱法于1950年由Greulich和Pyle[2]发明。TW2计分法是基于检测20处感兴趣区(ROI)及其特征进行综合评分[5]。中国也先后制定了李果珍法[6]、CHN法、中华05标准[7]等评估方法。此外,有学者基于国际通用的TW3方法,以当代中国儿童为样本,提出了通过桡骨远端和尺骨(DRU)进行分类的骨龄评定标准[8],更符合目前中国儿童骨龄发育情况。但这类方法存在以下缺点:(1)标准图谱的准确度对检测结果影响较大;(2)医师对比影像和图谱时会由于个人疲劳程度、单个阅片时间限制等主观原因产生偏差,个体差异大;(3)从阅片、查阅图谱到产生相应结果的时间较长。
随着人工智能技术的发展,人工智能专家试图通过机器学习方法解决上述问题。机器学习方法被广泛用于医学图像分析,通过在给定数据集上训练模型完成对新数据的分类、识别和分割等,常用算法有支持向量机(SVM)算法、隐马尔科夫(HMM)算法、人工神经网络等。然而,以往基于图像识别的自动骨龄检测方法需利用先验知识从原始数据中人工提取特征,多采用图像分割分类、背景差法和人工标记特征等工程训练模型。尽管这些方法在许多领域达到了合理的准确率[9],但由于特征选取难度较大,模型可能存在过拟合问题,泛化能力难以保证;且传统模型难以适应大规模数据集,模型可扩展性差[10]。此外,在处理带有大量噪音的图像、5岁以上儿童的影像数据时,传统自动图像识别方法的表现明显下降[11]。随着深度卷积神经网络不断刷新计算机视觉物体识别ImageNet大赛的记录,深度学习技术在计算机图像处理中的发展迅速,同时为影像辅助诊断提供了新的技术思路[12]。
本研究结合了左手X线骨龄检查与计算机视觉深度学习技术,对左手X线图像进行识别,旨在提取左手X线图像的骨龄评估关键位置特征,并结合年龄和性别信息建立骨龄评估回归模型,取得了较为准确的检测结果,现报告如下。
1 材料和方法 1.1 数据来源本研究采集了2016年1月至12月上海市儿童医院医学图像存档及通信系统(picture archiving and communication system,PACS)收集的11 858条dicom数据,数据进行批量脱敏处理,包括去除涉及患者基本信息的姓名、身份识别号字段以及图像中的患者姓名拼音。该数据集中患者年龄为0~18岁,均使用G-P图谱法测定骨龄,每幅图像的尺寸为1 500×1 000像素大小。图 1为数据集中不同年龄分布的儿童手骨X线图像。随机选择总数据的20%作为验证集,用于探寻适用于模型的最佳参数;剩余80%作为训练集。

|
图 1 不同年龄段不同成像设备拍摄的左手X线图像 Fig 1 Left hand X-ray image from different age ranges and digital radiology (DR) systems A: 0-3 years old; B: 4-6 years old; C: 7-9 years old; D: 10-12 years old; E: 13-15 years old (white bonding boxes indicate region of interests); F: 16-18 years old |
1.2 基于目标检测技术的模型求解与评价
首先对原始dicom图像进行预处理,保证图像的灰度分布处于同一分布区间。通过直方图匹配和归一化操作对图像进行灰度和尺度归一化,通过dicom的头文件分析调整灰度分布,并调整图像为500×500的输入大小,以黑色为成像背景,灰阶展现骨骼,并将灰度值归一化为0~1。
运用基于区域的卷积神经网络(region-based convolutional neural network,RCNN)[13]定位关键位置,在图像上生成一个滑动窗口,通过滑动窗口沿图像移动获得潜在的目标区域,使用RCNN对目标区域进行标准特征提取,即根据卷积、池化等操作得到固定维度的输出。然后,对上一步的输出向量进行分类(需要根据特征训练分类器);其次通过边界回归(bounding-box regression)得到精确的目标区域,由于实际目标会产生多个子区域,需对完成分类的前景目标进行精确的定位与合并,避免多个检出。图 2展示了RCNN的网络结构。
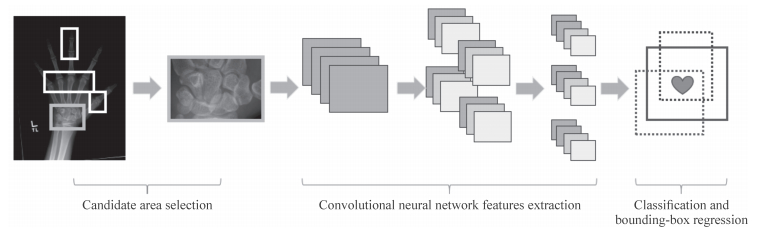
|
图 2 RCNN网络结构示意图 Fig 2 Network structure of region-based convolutional neural network (RCNN) |
边界回归训练过程中,输入数据为N个训练对{(Pi, Gi)},i=1, …, N,其中
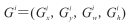
通过最小化损失函数,迭代生成多个(M个)弱模型,然后将每个弱模型的预测结果相加,后面的模型Fm+1(x)基于前面学习模型的Fm(x)的效果生成[14],初始化模型为常数值,其公式为:
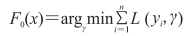
|
式中L为损失函数,γ为残差。通过迭代生成M个基分类器计算伪残差,其公式为:
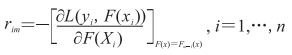
|
基于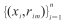
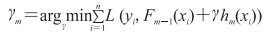
|
最后更新模型,得计算公式如下:
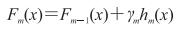
|
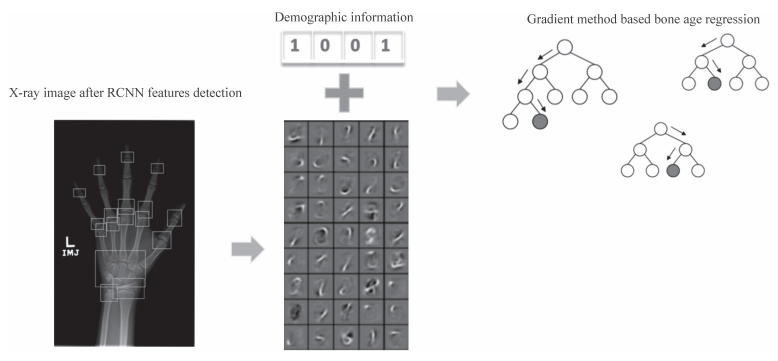
在骨龄的辅助检测中,需模拟影像科医师诊断关注的17个特征区域,首先检出左手X线图像上的17个关键区域,包括绕腕关节、腕骨关节、掌指关节和手指间关节,定位并提取这17个关键区域的特征,通过XGboost算法[15]训练骨龄评估模型,根据一阶和二阶导数迭代生成集学习器,相加更新学习器。图 3显示了骨龄回归路径和步骤。
|
图 3 骨龄回归实现步骤示意图 Fig 3 Schematic illustration of automatic bone age regression method Using faster-RCNN to train the model to get region of interests, 17 regions of interests are detected on left hand X-ray image, and each image generates a series of deep information, and then using dimension reduction and adding gender and age information, finally bone age can be regressed with gradient method. RCNN: Region-based convolutional neural network |
1.4 损失函数
本研究选取交叉熵损失函数作为分类任务的损失函数[16],交叉熵损失的计算公式如下:
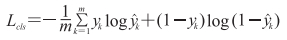
|
式中m为样本个数,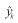
选取L2损失函数为XGboost回归边框任务的损失函数,L2损失函数的公式如下:
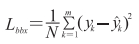
|
式中m为样本个数,
模型训练完毕后,另采集上海市儿童医院2018年2月至3月PACS中1 229例0~17岁骨龄检测的影像数据作为测试集,检测该模型的性能。基于G-P图谱法再次评估所有数据,并对过估、低估结果由2位以上儿科放射医师进行讨论以达成共识(商讨后的骨龄值)。将测试集数据经预处理后输入训练好的关注区域定位模型中,提取并平均化处理所关注特征区域的深度学习特征,再结合性别、年龄信息组成多模态的特征,输入到训练好的骨龄回归模型中预测骨龄,并与达成共识的骨龄进行比较。采用平均绝对误差(mean absolute error,MAE)评价骨龄回归模型的骨龄评估准确性[17],计算公式如下:
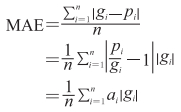
|
式中,n为样本总量,gi和pi为2种方法分别对第i个样本的观察结果。
模型训练完毕后分别计算其评估验证集和测试集所有数据的MAE值,并与已有文献进行对比分析。同时,由于加入了性别和年龄信息,将验证集结果按年龄和性别分别统计并比较MAE。
1.6 实验环境本实验基于深度学习框架Tensorflow 1.4,在2块TitanX显卡上完成,并使用python 3.5和jupyter完成数据的统计和分析。
图像预处理:使用python 3.5的pydicom工具包将dicom格式的影像转换为jpg格式图片。使用opencv直方图均一匹配方法将所有灰度分布匹配到同一灰度分布。使用算法工具将灰度值归一化转换为0~1。
训练数据标注:由1位经验丰富的放射科医师使用labelImg工具对图像进行标注,并由1位医师进行审核,使用包围盒(bounding box)在骨龄图像上对腕骨、掌骨远端、近节指骨近端、远节指骨近端、桡骨、尺骨共17个区域进行标注。
建立数据集:将数据集按8:2的比例随机分为训练集和验证集,对图像进行翻转、旋转等数据增强操作。
建立关键区域特征提取模型:选取RESNET101作为faster RCNN的基础网络[13],选取focal loss作为损失函数,同时提取左手X线图像中17个关键区域的特征。每个关键区域生成2 048维特征,基于图像共产生17×2 048维特征;对同一类型的特征区域进行加权平均处理后,降维至6×2 048维特征。此外,儿童年龄、性别为骨龄判定的重要标准之一,输入信息中增加数据。通过XGboost算法[15],使用贪心策略对max_depth,min_child_weight,gamma,subsample,scale_pos_weight等超参数例进行最优调试,得到的整体MAE与医师阅片结果进行最小化最优回归,建立最优骨龄检测模型。
学习率从0.001开始每20 000步减小10倍,直到为0.000 01。RPN_ANCHOR_RATIOS(锚定区域区域比例)为[0.5, 1, 2],每个GPU batch为2,faster-RCNN基础网络为RESNET101,输入图像的最短边为800,包括腕骨、掌骨远端、近节指骨近端、远节指骨近端、尺骨和背景的分类,总共12个类别。
2 结果 2.1 预测结果对比本研究的训练集、验证集和测试集数据分布情况如表 1所示。训练集、验证集和测试集的数据分布总体相同,男童和女童的数据分布均不呈高斯分布,在男童和女童3岁时均出现第1个高峰,之后男童人数第2个高峰出现在13岁,而女童人数在11岁时达到最大值。
|
|
表 1 训练、验证和测试数据集的年龄段分布 Tab 1 Distribution of different real ages in training, validation and test data |
基于包括1 229例X线图像和相应的骨龄数据的分析,本研究检测得到的骨龄MAE为0.455,处理每张图像平均耗时0.4 s,其与各类骨龄检测方法的比较结果如表 2所示。随着数据量的增加,MAE总体呈下降趋势,且深度学习方法[18]所得的MAE低于传统图像处理[19]、降维算法[11]、模糊算法[10]等非深度学习方法。此外,与深度学习端到端的整体图像学习方法[18]相比,本模型通过关注特征区域以及性别和年龄进行骨龄检测,所得MAE进一步下降。
|
|
表 2 各类方法的数据量、MAE对比 Tab 2 Comparison of MAE and amount of data between our experiment and other methods |
2.2 各年龄段分布对比
比较本研究检测不同年龄段男童和女童骨龄的MAE,结果如表 3所示。男童的MAE为0.445,女童为0.474。按年龄段分类,男童和女童的MAE最小值均出现在0~2岁,MAE最大值均出现在17~18岁。2~16岁区间内,男童和女童的MAE均在0.308~0.668间分布。
|
|
表 3 各年龄段患儿的MAE Tab 3 MAE result among different age ranges |
3 讨论
相较于目前医院普遍运用的G-P图谱法,全自动影像分析一直是计算机视觉和放射学研究的目标。与传统机器学习方法相似,大多数现有方法都是利用计算机分割或定位算法定位ROI。本方法使用RCNN能达到较高的骨龄预测精度,说明左手的掌指骨、腕骨、尺骨和桡骨区域对于骨龄检测有重要作用。
早有学者将机器学习方法引入骨龄检测,如Michael和Nelson[21]实现的计算机辅助骨龄自动评估系统。Pietka等[3]在2001年通过对指骨区域分割,实现了基于TW2计分法的骨龄自动评估。Bocchi等[22]通过Gabor变换和多尺度滤波方法,完成了对X线图像中各类骨骼的预处理,并利用神经网络实现了基于TW2计分法的骨龄自动评估。Gertych等[19]将骨龄评估细分为3个步骤,即腕骨自动分割、形态学特征提取和模糊分类,以保证骨龄自动评估的性能。Lee等[23]提出了基于深度学习方法的骨龄评估方法,实现了对整个手腕区域所反映骨龄值的回归预测。
本方法优势在于提取了关键区域的深度特征,因此准确率较高。一些传统方法使用的是不同的数据集,且未详细标明具体数据集类型,因此将本研究与传统方法相比缺乏公平性。Spampinato等[18]首先使用了深度学习的方法,通过端到端的回归方法实现了基于X线图像的深度学习骨龄回归,其MAE与传统方法相比明显提升,然而端到端的方法忽略了医师临床诊断需要的领域专业知识,也没有添加性别等人口学信息,其MAE高于本研究方法,表明在图像中提供医师的专业知识作为附加信息可以提高模型的精度。此外,本模型在验证集和测试集上均取得了比既往研究更小的MAE,并且在验证集和测试集上的误差较小,提示本研究方法具有较好的稳定性,且引入性别和年龄变量可以更准确描述生长发育中骨骼发育的变化。
由于本研究直接采用医院的真实数据,儿童的数据在各个年龄段不符合高斯分布。医院影像科数据分布体现了儿童集中到医院就诊的年龄分布,第1个数据高峰与儿童生长发育的动态变化呈正相关[24],第2个高峰与青春期发育有关,因此出现在男童和女童的青春期前后。数据分布差异导致了相应的MAE差异和模型回归精度的不平衡[25]。因此,在训练集数量较小的高年龄段中,模型的MAE大于数据量较大的年龄段。
本研究发现对于<24个月的骨龄X线影像,G-P图谱法的年龄间隔为3个月;对于<5岁的骨龄X线影像,G-P图谱法的年龄间隔为6个月。由于学习图像的误差较小,<5岁的儿童的MAE与G-P图谱法的分辨率精度呈正相关,而对于6~14岁的骨龄图像,训练样本量越大的区域MAE更小,与既往深度学习样本量越多模型越准确的结论一致。同时,由于>15岁且≤18岁的儿童训练样本、测试样本均较少,需增加样本量对模型进行进一步优化。
骨龄回归依赖于定位结果,因为定位算法的准确度问题和影像质量不高(影像曝光程度、拍摄模糊、倾斜度较大等)会导致定位不准确,从而影响骨龄回归结果。
与传统的端到端方法相比,本研究中依据G-P图谱法以及ROI所提取的重点关注区域可以进一步提升骨龄检测结果,然而实际骨龄不仅仅由17个特征区域决定,其他位置的特征也会影响骨龄结果。进一步研究可就以下几个方面展开:(1)在数据量增加的情况下训练端到端的网络,并通过观察神经网络关注的区域进行热力图指导相应研究;(2)本方法中第2步骨龄回归采用了一维特征进行分类,可能会丢失特征的空间相关信息,有待进一步研究设计更准确的骨龄回归模型;(3)重点收集15~18岁青少年的数据,增加训练集中相应年龄段的数据分布。
综上,本研究提出的基于深度学习的骨龄检测方法通过学习X线图像数据和临床医学经验,使用RCNN的特征提取网络提取左手骨龄图像中骨骼发育的重要区域特征,通过区域学习对高维度特征进行降维,并进一步添加年龄和性别信息,使用基于梯度的算法对数据进行降维,构建了骨龄回归模型。该模型可达到令人满意的骨龄检测准确度,为儿童生长发育提供了数据参考依据。
| [1] |
沈勋章. 手腕部骨龄鉴定方法的研究进展[J]. 中国医药科学, 2011, 1: 9-12. |
| [2] |
GERTYCH A, ZHANG A, SAYRE J, POSPIECH-KURKOWSKA S, HUANG H K. Bone age assessment of children using a digital hand atlas[J]. Comput Med Imaging Graph, 2007, 31: 322-331. DOI:10.1016/j.compmedimag.2007.02.012 |
| [3] |
PIETKA E, GERTYCH A, POSPIECH S, CAO F, HUANG H K, GILSANZ V. Computer-assisted bone age assessment:image preprocessing and epiphyseal/metaphyseal ROI extraction[J]. IEEE Trans Med Imaging, 2001, 20: 715-729. DOI:10.1109/42.938240 |
| [4] |
REN S, HE K, GIRSHICK R, SUN J. Faster R-CNN:towards real-time object detection with region proposal networks[J]. IEEE Trans Pattern Anal Mach Intell, 2017, 39: 1137-1149. DOI:10.1109/TPAMI.2016.2577031 |
| [5] |
BULL R K, EDWARDS P D, KEMP P M, FRY S, HUGHES I A. Bone age assessment:a large scale comparison of the greulich and pyle, and tanner and whitehouse (Tw2) methods[J]. Arch Dis Child, 1999, 81: 172-173. DOI:10.1136/adc.81.2.172 |
| [6] |
新瑜, 李瑞珍. 单纯性肥胖儿童骨龄与体质量指数及性激素水平的相关性[J]. 实用儿科临床杂志, 2011, 26: 487-488. DOI:10.3969/j.issn.1003-515X.2011.07.008 |
| [7] |
朱翔宇, 欧阳斌, 罗冬梅, 刘杰. 基于中华05骨龄标准的桡骨骨龄等级计算机评定系统的设计[J]. 体育科学, 2016, 36: 59-65. DOI:10.3969/j.issn.1004-3624.2016.05.012 |
| [8] |
MALINA R M, BEUNEN G P. Assessment of skeletal maturity and prediction of adult height (Tw3 method)[J]. Amer J Human Biol, 2002, 14: 788-789. DOI:10.1002/(ISSN)1520-6300 |
| [9] |
THODBERG H H, KREIBORG S, JUUL A, PEDERSEN K D. The BoneXpert method for automated determination of skeletal maturity[J]. IEEE Trans Med Imaging, 2009, 28: 52-66. DOI:10.1109/TMI.2008.926067 |
| [10] |
HSIEH C W, JONG T L, TIU C M. Bone age estimation based on phalanx information with fuzzy constrain of carpals[J]. Med Biol Eng Comput, 2007, 45: 283-295. DOI:10.1007/s11517-006-0155-9 |
| [11] |
THODBERG H H. An automated method for determination of bone age[J]. J Clin Endocrinol Metab, 2009, 94: 2239-2244. DOI:10.1210/jc.2008-2474 |
| [12] |
GREENSPAN H, GINNEKEN B V, SUMMERS R M. Guest editorial deep learning in medical imaging:overview and future promise of an exciting new technique[J]. IEEE Trans Med Imaging, 2016, 35: 1153-1159. DOI:10.1109/TMI.2016.2553401 |
| [13] |
HE K, ZHANG X, REN S, SUN J. Deep residual learning for image recognition[C]. Proceedings of IEEE Conference on Computer Vision and Pattern Recognition, 2016:770-778.
|
| [14] |
FRIEDMAN J H. Greedy function approximation:a gradient boosting machine[J]. Annals Statistics, 2001, 29: 1189-1232. |
| [15] |
CHEN T, GUESTRIN C. Xgboost:a scalable tree boosting system[C]//the 22nd Acm Sigkdd International Conference on Knowledge Discovery and Data Mining. ACM, 2016:785-794.
|
| [16] |
RUBINSTEIN R Y, KROESE D P. Preliminaries[M]//The Cross-Entropy Method. New York, NY:Springer, 2004:1-28.
|
| [17] |
WILLMOTT C J, MATSUURA K. Advantages of the mean absolute error (MAE) over the root mean square error (RMSE) in assessing average model performance[J]. Climate Res, 2005, 30: 79-82. DOI:10.3354/cr030079 |
| [18] |
SPAMPINATO C, PALAZZO S, GIORDANO D, ALDINUCCI M, LEONARDI R. Deep learning for automated skeletal bone age assessment in X-ray images[J]. Med Image Anal, 2017, 36: 41-51. DOI:10.1016/j.media.2016.10.010 |
| [19] |
GERTYCH A, ZHANG A, SAYRE J, POSPIECH-KURKOWSKA S, HUANG H K. Bone age assessment of children using a digital hand atlas[J]. Comput Med Imaging Graph, 2007, 31(4/5): 322-331. |
| [20] |
GIORDANO D, KAVASIDIS I, SPAMPINATO C. Modeling skeletal bone development with hidden Markov models[J]. Comput Methods Programs Biomed, 2016, 124: 138-147. DOI:10.1016/j.cmpb.2015.10.012 |
| [21] |
MICHAEL D J, NELSON A C. HANDX:a model-based system for automatic segmentation of bones from digital hand radiographs[J]. IEEE Trans Med Imaging, 1989, 8: 64-69. DOI:10.1109/42.20363 |
| [22] |
BOCCHI L, FERRARA F, NICOLETTI I, VALLI G. An artificial neural network architecture for skeletal age assessment[C/OL]. Proceedings of International Conference on Image Processing, 2003, 1:I-1077-80. doi: 10.1109/ICIP.2003.1247153.
|
| [23] |
LEE H, TAJMIR S, LEE J, ZISSEN M, YESHIWAS B A, ALKASAB T K, et al. Fully automated deep learning system for bone age assessment[J]. J Digit Imaging, 2017, 30: 427-441. DOI:10.1007/s10278-017-9955-8 |
| [24] |
刘慧娟, 高丽, 李桂玲, 王霞, 雷平冲, 薛莉, 等. 郑州市0~3岁儿童生长发育动态变化分析[J]. 中华实用诊断与治疗杂志, 2015, 29: 989-991. |
| [25] |
HUANG C, LI Y, CHEN C L, TANG X. Learning deep representation for imbalanced classification[C/OL]. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), 2016:5375-5384. doi: 10.1109/CVPR.2016.580.
|
 2018, Vol. 39
2018, Vol. 39


