2. 上海交通大学生命科学技术学院上海交大-耶鲁大学生物统计与数据科学联合中心,上海 200240;
3. 上海交通大学附属儿童医院儿童精准医学大数据工程技术研究中心, 上海 200040;
4. 上海交通大学附属儿童医院儿童保健科, 上海 200040
2. SJTU-Yale Joint Center of Biostatistics and Data Science, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China;
3. Big Data Engineering and Technology Research Center for Pediatric Precision Medicine, Children's Hospital of Shanghai Jiao Tong University, Shanghai 200040, China;
4. Department of Child Healthcare, Children's Hospital of Shanghai Jiao Tong University, Shanghai 200040, China
罕见病是指发病率极低且仅在极少数人身上发生的疾病或病变,因此又被称为孤儿病。有些罕见病即便在婴儿出生时没有显示出明显症状,也会随着时间推移慢慢出现且病变越来越严重,大部分症状会伴随患者终身。许多罕见病在患者生命早期发病,约有30%的罕见病儿童会在5岁前死亡[1]。较为常见的罕见病有苯丙酮尿症、Wilson病、黏多糖症、高血氨症和有机酸血症等。罕见病目前没有统一并被广泛接受的定义,世界各国对罕见病的界定存在差异。根据世界卫生组织的定义,罕见病是患病人数占总人口65/100 000到1/100 000的疾病或病变。美国在《2002年罕见病法案》中将患病人口保持在20万以内或者患病率为1/1 500的疾病定义为罕见病。欧盟对罕见病的定义进行了进一步细化,他们从罕见病的流行范围、发病时长及严重程度对罕见病进行了定义。欧盟将流行范围很小(一般低于1/2 000)、发病时间长或伴随终生、严重威胁患者生命或患者因此变得相当虚弱、需要多种专门治疗手段的疾病定义为罕见病。我国将患病率低于1/500 000或新生儿发病率低于1/10 000的疾病定义为罕见病[2]。按照美国研究机构的估计,全球大约有7 000种罕见病,共影响约3亿的人口[3]。按照欧洲研究机构的估计,大约有5 000~7 000种罕见病,影响6%~8%的人口[4]。据文献报道,中国的罕见病患者人数约有1 000万,但考虑到庞大的人口基数,这一数字可能被严重低估[5]。约75%的罕见病在出生时或儿童期即可发病,并且部分罕见病即便被及时诊断也缺乏有效的治疗或干预手段,因此罕见病是儿科学中一项亟待突破的关键研究领域[6-7]。
目前罕见病在临床上面临3大难题:(1)罕见病难以诊断,经常漏诊、误诊;(2)罕见病即便被成功诊断,也很难进行临床干预,在约7 000种罕见病中,目前只有大约400种罕见病具备治疗手段[8];(3)罕见病知识文献更新速度快,临床工作者难以及时跟进最新的罕见病研究与诊疗进展。当前全球罕见病专家的共识在于越早诊断越有利于预防病情恶化、治疗干预和减少家庭负担。对于大部分可防、可治且相对比较常见的罕见病,如代谢性疾病通过新生儿筛查即可进行及时有效的干预。上海市儿童医院早在1985年就成立了新生儿筛查中心,截至2010年底已经筛查了超过100万新生儿,确诊了700多例罕见代谢疾病,通过及时的随访治疗,超过99%的患者生活正常,其中还有全国化学竞赛的获奖者[9]。然而,可通过代谢谱筛查出来的罕见病只是一小部分。据统计,目前全球25%的罕见病患者需反复就医5~30年才能被正确诊断,延误了病情的控制治疗,给患者及其家庭造成了沉重的经济和精神负担[10]。
1 人工智能技术为罕见病诊疗提供机会造成罕见病诊断困难的原因很多,究其根本是罕见病的特殊性。首先,罕见病包括约7 000种不同疾病,且这些疾病散布于不同科室,各科室的临床医师很少见到真实案例,造成临床医师无法获得诊断经验,在日常诊疗中很难将罕见病与常见相似疾病区分。而不准确的临床诊断形成后往往难以纠正,导致患者反复就医。其次,罕见病的诊断方法大多基于基因检测和遗传分析技术,相关技术和知识在大部分医疗机构中尚未普及,即便临床形成了罕见病的预判,获得了基因检测结果,由于罕见病的复杂性和基因检测结果的复杂性,临床医师也很难在数千种疾病中准确诊断。
临床决策支持系统(clinical decision support system,CDSS)充分利用了海量的医学知识和人工智能分析引擎,并且整合结构化、半结构化或非结构化医学信息,通过人机交互的方式改善和提高了医疗决策效率。CDSS一直是人工智能领域的热门话题,也是人工智能方法最有价值的应用领域。2011年IBM公司推出的Watson系统即对医疗领域产生了重大影响,Watson已与纪念斯隆―凯特琳癌症中心等国际著名医疗机构及国内的一些医院合作尝试进行临床辅助诊疗并取得了重大的成果。基于图像识别的深度学习框架也在医疗图像诊断识别中取得了令人惊叹的成果[11-15],如糖尿病性视网膜病变诊断[16]和皮肤癌诊断[17]等。近年来,上海市儿童医院在人工智能和儿童医疗健康领域开展了大量工作:药剂科基于药物基因组学技术开发了儿童精准用药系统,目前已整合进入临床流程,受到美国医疗信息与管理系统学会(Healthcare Information and Management Systems Society,HIMSS)7级评审专家组的好评;放射科将儿科X线图像与深度学习引擎结合,开发了自动化儿童骨龄智能评估系统,该系统目前正在临床推广验证。机器学习和人工智能技术在罕见病的诊断中展现了巨大潜力,一方面,现代医疗知识库系统具有海量疾病信息的高效存储、管理和访问能力,方便临床医师快速检索和查询已知罕见病及其研究进展;另一方面,得益于人工智能算法模型的进步,罕见病临床智能辅助决策引擎可以快速综合、归档、识别和辨别罕见病所需信息,对患者症状进行初步评估,减少了临床医师重复烦琐的工作,以便其将更多精力分配到遗传病的辨别、诊断和治疗中。上海市儿童医院在罕见病智能辅助诊断系统领域深耕多年,研发了针对儿童罕见病的智能辅助决策系统,经过初期测试评估其精度达到了同类世界先进水平[18]。
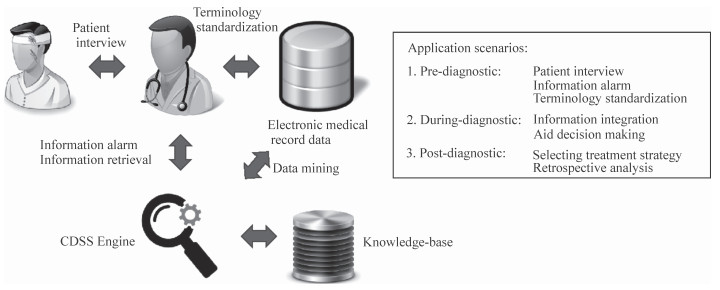
根据罕见病辅助诊断的临床应用场景,可以将罕见病CDSS分为3类(图 1)。(1)诊断前系统:诊断前系统是医患交流期间使用的系统,一般用于医师的问诊环节,其主要功能在于辅助医师进行病史采集、对疑似罕见病的病例进行风险预警及推荐疑似病例确诊所需的检查和检验,该系统应当具备疾病知识库和疾病表型知识库的交互式检索能力,对推荐的信息给出足够的支持证据,减少临床医师的误判。(2)诊断中系统:诊断中系统专门用于临床表型、疾病知识和分子遗传等信息的综合评估,并给出潜在罕见病的候选列表,该系统的主要功能在于综合患者的疾病表型和基因型信息,对已有的7 000多种罕见病进行评估打分,辅助临床医师准确诊断。(3)诊断后系统:罕见病确诊后,CDSS需从知识库调取该罕见病的参考治疗方案,供临床医师选择。同时,罕见病CDSS还能对既往病例进行回溯,根据历史罕见病病例评估当前病患。此外,利用医院(甚至区域医疗信息系统)的电子病历(electronic medical record,EMR)数据,基于EMR的疾病表型识别技术和罕见病CDSS分析引擎,可挖掘并识别反复就医、具备特殊疾病表型的患者,建立高风险疾病队列,并给予特殊的临床指导和干预。目前国际上主要的罕见病CDSS主要集中于第1类和第2类,本文列举了一些有代表性的工具资源(表 1)。
|
图 1 罕见病临床决策支持系统(CDSS)的临床应用场景 Fig 1 Clinical application scenarios of clinical decision support system (CDSS) for rare disease |
|
|
表 1 目前主要的罕见病辅助诊断工具及知识库资源 Tab 1 Current tools and resources for diagnosis of rare diseases |
2 罕见病CDSS中的人工智能技术
典型的CDSS至少应该包含3个主要部分:知识库、推理引擎和人机交互系统。罕见病CDSS是典型的强知识库型CDSS,知识库系统是其核心组件,本文将从罕见病中涉及的人工智能技术和知识库系统2个部分简单介绍罕见病的CDSS。
2.1 疾病表型分析系统在传统的罕见病诊断过程中,临床医师主要依靠人工加互联网搜索的方式对患者进行疾病表型的描述。但在临床实践中,因为诸多原因往往很难获得患者精准、全面的表型描述。罕见病CDSS的第1个应用场景就是辅助临床医师进行精准、全面的患者表型注释。除利用CDSS的交互式检索能力外,近年来基于人工智能技术还发展出了一些新兴的疾病表型分型技术,这些技术均可被称为“下一代疾病表型分型技术”。如基于图像等高维数据的疾病表型分型技术,face2gene软件可利用患者的脸部照片对面部特征进行量化,利用图像识别技术、人工智能算法与参考数据库进行比对给出罕见病的诊断参考,可取得不错的效果[19];高度自动化的临床病例数据表型化技术可利用自动化、高通量的疾病表型标准系统,分析患者的历史诊疗记录,对患者的疾病发生、发展谱进行详细的分析和全面的疾病表型刻画,为基于人工智能系统的决策引擎提供全面、丰富的特征信息[20]。
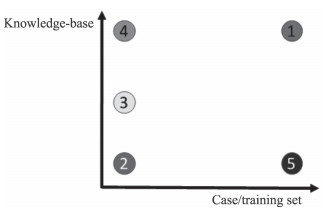
2.2 罕见病CDSS的推理引擎依据当前CDSS决策引擎的特点,大致可以将CDSS分为临床路径驱动型决策系统、医疗规则驱动型决策系统、医学知识驱动型决策系统、基于机器学习的决策系统。基于临床路径和医疗规则的决策系统因其对知识库和决策引擎的要求较低,且最贴近临床的日常需求,在目前医院信息系统(hospital information system,HIS)中最为常见。由于目前机器学习的“黑箱”问题,基于机器学习的CDSS在临床应用中还不常见,但其发展速度迅猛。由于医学知识库体系的复杂性,基于大型医学知识库和智能推理引擎的决策系统发展较为缓慢,但对于罕见病智能辅助诊断,这类CDSS的架构最适合,因为罕见病及其疾病表型和基因型的描述和表示需要庞大的知识库体系作为支撑。此外,罕见病病例散发且稀少,并不能充分发挥机器学习方法的“暴力求解”能力。见图 2。目前本课题组也在测试和优化研发的基于医学知识库的罕见病CDSS。当前用于驱动罕见病表型知识库系统的推理引擎大多是基于本体结构的语义学方法,其中比较典型的有Resink[21]、Lin[22]、graph IC measure[23]等,也有少量基于搜索引擎技术的方法,如FindZebra[24]、MalaCards[25]等。就当前发展趋势而言,各种罕见病CDSS都在尝试综合疾病表型知识库和疾病基因知识库,希望能建立统一的推理引擎框架。目前世界各国都已启动罕见病的疾病队列研究,如果能积累足够病例,无疑将会突破罕见病CDSS推理引擎目前的局限。
|
图 2 4类临床决策支持系统(CDSS)与知识库系统和训练数据的依赖关系 Fig 2 Relationships between four different types of CDSS and knowledge-base and training data 1: Ideal CDSS; 2: Work-flow CDSS; 3: Rule based CDSS; 4: Knowledge-base CDSS; 5: Machine-learning based CDSS. CDSS: Clinical decision support system |
3 罕见病医学知识库体系
任何人工智能系统离不开知识库体系的支持,知识库系统是人工智能系统的真正“大脑”,而算法引擎只是驱动的“心脏”。如通用智能问答系统Watson系统,它的知识库存储了数百万量级的图书、文献和资料。与通用知识库、知识图谱系统不同,医疗系统的知识库具有其专业性和特殊性,特别是在罕见病领域,它有着非常特别的知识库体系,以下将从疾病知识库、表型知识库和基因知识库3类知识库作简要介绍。
3.1 罕见病疾病知识库罕见病种类较多,症状较一般疾病复杂,且缺乏相关资料,因此需要建立罕见病相关数据库,为患者、医师及研究人员提供信息检索、资讯及学习等服务。Orphanet(https://www.orpha.net/consor/cgi-bin/index.php)、OMIM(http://omim.org/)和DECIPHER(database of chromosomal imbalance and phenotype in humans using ensembl resources,https://decipher.sanger.ac.uk/)是罕见病研究领域比较重要的数据库。Orphanet收录了近6 000种罕见病相关信息,是目前最全面的罕见病数据库。不仅如此,Orphanet还收录了大量与罕见病治疗相关的药物(即孤儿药物)信息及资料,为患者、医师及研究人员提供了宝贵的罕见病信息。美国Johns Hopkins大学医学院Victor A. McKusiek教授主编的《人类孟德尔遗传(Mendelian Inheritance in Man)》是医学遗传学领域最权威的百科全书,甚至被誉为医学遗传学界的“圣经”。OMIM(online Mendelian inheritance in man)是《人类孟德尔遗传》在线版,它收录了所有已知的遗传病、性状及其基因,除了简略描述各种疾病的临床特征、鉴别诊断、治疗与预防外,OMIM还提供致病基因的连锁关系、染色体定位、功能和动物模型等资料,以及大量与疾病研究相关的重要参考文献。DECIPHER是目前分子遗传学中最重要的数据库之一。DECIPHER收集了一系列遗传疾病相关信息,包括变异位点、临床表型等,为罕见病的诊断提供了大量信息。最为重要的是,DECIPHER还收录了超过200家研究中心上传的超过10 000例的真实临床案例,为临床医师诊断罕见病提供了参考。除了Orphanet、OMIM和DECIPHER外,基因位点数据库,如1000 Genomes、HGMD、dbSNP、ClinVar、ExAC等也为罕见病的诊断和研究提供了重要帮助。
3.2 罕见病表型知识库疾病症状(表型)的标准化是构建罕见病辅助诊断系统的前提。有学者提出应用计算机科学中的本体论概念来实现疾病表型的标准化。本体论的思想是对医学文献信息进行总结归纳,提取常用的疾病表型词汇并建立词汇间结构化的分层关系,使用统一的语言描述疾病,从而实现表型的标准化。其中,最具代表性的是由Robinson等[26]于2008年提出的人类表型本体(human phenotype ontology,HPO)。HPO从表型异常、遗传模式、临床调节因素和衰老/死亡4个方面对疾病表型进行了系统整理,根据医学文献和专家意见将表型症状进行分层划分。因此,可以使用HPO作为语言对疾病进行系统化及标准化的描述。到目前为止,HPO已经完成了对OMIM中7 000多种遗传病、Orphanet中3 000多种罕见病的统一标注。2016年,国内成立的中文人类表型标准用语联盟基本完成了对人类表型本体中词汇的汉化翻译,增加了HPO在国内罕见病辅助诊断领域的实用性。
HPO系统化描述了罕见疾病患者的表型特征,很多罕见病辅助诊断工具利用HPO作为标准化表型描述工具。通过对罕见病标准化的HPO表型注释和参考罕见病的HPO表型注释,可识别可能罕见病,也可以进行致病突变的识别排序。HPO体系已被很多大型研究计划所接受,如美国国立卫生研究院的Undiagnosed Diseases Program and Network、英国政府10万人基因组计划、英国Sanger研究中心的DECIPHER、英国Sanger研究中心的Deciphering Developmental Disorders计划和欧洲遗传病参考协作网(European Reference Networks for Rare Diseases),以及我国的国家罕见病注册登记系统。
3.3 罕见病基因知识库罕见病与遗传病的界限经常被模糊,然而罕见病并不完全等同于遗传病,只有约80%的罕见病可能与遗传变异相关[8]。对于这些与遗传变异相关的罕见病,可以依据遗传病的分类标准进行分类:(1)染色体病,即染色体数目或结构异常所致的疾病,染色体病发病率通常较高,且多数为新发突变,如唐氏综合征;(2)单基因遗传病,即由1对等位基因控制的遗传性疾病,按照传递方式分为常染色体显性、隐性和性连锁遗传性疾病;(3)多基因遗传病,即由2对及以上等位基因发生变异而导致的遗传性疾病,同时该类疾病的形成还可能受环境因素的影响;(4)线粒体病,即线粒体DNA异常导致的遗传性疾病,该疾病为母系遗传。
目前常见的收录了罕见遗传疾病的基因知识库非常多,主要有:(1)基因功能数据库,如NCBI refGene、Ensemble ensGene、UCSC knownGene等,这类数据库提供了基因组中基因与基因间区信息等相关信息;(2)基因位点功能数据库,如ClinVar、HGMD、dbSNP等,这类数据库的数据主要来源于文献报道的基因位点及该位点的致病信息,为罕见遗传病的诊断提供了直接诊断依据;(3)基因位点人群频率数据库,如1000 Genomes、ExAC、Esp6000等,这类数据库的数据主要来源于大规模正常人群队列研究,基因位点的人群频率信息可以有效排除基因组中的非致病性位点,为罕见遗传病诊断中假阳性位点的排除提供依据;(4)基因位点功能预测数据库CAD Gene(https://ghr.nlm.nih.gov/gene/CAD)、M-CAP[27]、REVEL等,这类数据库主要来源于对已有数据库进行深度挖掘,发现致病性位点可能的特性,从而对基因组中所有可能的致病性位点进行识别,在一定程度上弥补了现有数据库数据量少的缺点。
4 小结自20世纪70年代全世界第1个CDSS——斯坦福大学的MYCIN系统出现以来,医疗信息领域已经积累一定的CDSS研发和应用经验。一个成功的CDSS至少需包含2项关键要素:(1)CDSS需要伴随临床诊疗流程,方便临床医师随时调取使用,且需要与现有医疗信息系统深度整合,孤立的CDSS很难成功。(2)CDSS不能只提供“yes/no”式的答案,而需要提供一系列的临床选择候选,提供评估打分及相应的支撑证据链,并与临床医师的诊疗逻辑深度耦合,才能被临床接受[28]。罕见病CDSS为一种比较新的CDSS,在其发展过程中同样需要认真考虑这些经验。此外,罕见病CDSS研究者应共同努力,推进建立用于评估和测试罕见疾病诊断精度的开放标准参考病例数据集,促进本领域技术和方法的发展。
| [1] |
Wikipedia. Rare diease[EB/OL]. [2017-12-08]. https://en.wikipedia.org/wiki/Rare_disease.
|
| [2] |
陈静. 可治性罕见病[M]. 上海: 上海交通大学出版社, 2017: 前言.
|
| [3] |
Rarelist: globalgenes; 2018[EB/OL]. [2018-06-28]. https://globalgenes.org/rarelist/.
|
| [4] |
EURORDIS Rare Diseases Europe. Rare diseases: understanding this public health priority[EB/OL]. [2018-06-28]. https://www.eurordis.org/IMG/pdf/princeps_document-EN.pdf.
|
| [5] |
WANG J B, GUO J J, YANG L, ZHANG Y D, SUN Z Q, ZHANG Y J. Rare diseases and legislation in China[J]. Lancet, 2010, 375: 708-709. DOI:10.1016/S0140-6736(10)60240-1 |
| [6] |
FUNG K W, RICHESSON R, BODENREIDER O. Coverage of rare disease names in standard terminologies and implications for patients, providers, and research[J]. AMIA Annu Symp Proc, 2014, 2014: 564-572. |
| [7] |
GONG S, JIN S. Current progress in the management of rare diseases and orphan drugs in China[J]. Intractable Rare Dis Res, 2012, 1: 45-52. |
| [8] |
SANFILIPPO A, LIN J. Rare diseases:diagnosis, therapies and hope[M]. St. Louis, MO: Rare Genomics Institute, 2017: 6.
|
| [9] |
上海市儿童医院新生儿筛查中心[EB/OL]. [2018-06-28]. http://www.shchildren.com.cn/ks/yj/ks_xsrsczx/contents/358/2470.html.
|
| [10] |
SCHIEPPATI A, HENTER J I, DAINA E, APERIA A. Why rare diseases are an important medical and social issue[J]. Lancet, 2008, 371: 2039-2041. DOI:10.1016/S0140-6736(08)60872-7 |
| [11] |
LEE J H, KIM K G. Applying deep learning in medical images:the case of bone age estimation[J]. Healthc Inform Res, 2018, 24: 86-92. DOI:10.4258/hir.2018.24.1.86 |
| [12] |
JEON J P, KIM C, OH B D, KIM S J, KIM Y S. Prediction of persistent hemodynamic depression after carotid angioplasty and stenting using artificial neural network model[J]. Clin Neurol Neurosurg, 2018, 164: 127-131. DOI:10.1016/j.clineuro.2017.12.005 |
| [13] |
YATES E J, YATES L C, HARVEY H. Machine learning "red dot":open-source, cloud, deep convolutional neural networks in chest radiograph binary normality classification[J]. Clin Radiol, 2018, 73: 827-831. DOI:10.1016/j.crad.2018.05.015 |
| [14] |
HA R, CHANG P, KARCICH J, MUTASA S, FARDANESH R, WYNN R T, et al. Axillary lymph node evaluation utilizing convolutional neural networks using MRI dataset[J/OL]. J Digit Imaging, 2018. doi: 10.1007/s10278-018-0086-7.[Epubaheadofprint].
|
| [15] |
LI Z, HE Y, KEEL S, MENG W, CHANG R T, HE M. Efficacy of a deep learning system for detecting glaucomatous optic neuropathy based on color fundus photographs[J]. Ophthalmology, 2018, 125: 1199-1206. DOI:10.1016/j.ophtha.2018.01.023 |
| [16] |
GULSHAN V, PENG L, CORAM M, STUMPE M C, WU D, NARAYANASWAMY A, et al. Development and validation of a deep learning algorithm for detection of diabetic retinopathy in retinal fundus photographs[J]. JAMA, 2016, 316: 2402-2410. DOI:10.1001/jama.2016.17216 |
| [17] |
ESTEVA A, KUPREL B, NOVOA R A, KO J, SWETTER S M, BLAU H M, et al. Dermatologist-level classification of skin cancer with deep neural networks[J]. Nature, 2017, 542: 115-118. DOI:10.1038/nature21056 |
| [18] |
GONG X, JIANG J, DUAN Z, LU H. A new method to measure the semantic similarity from query phenotypic abnormalities to diseases based on the human phenotype ontology[J/OL]. BMC Bioinformatics, 2018, 19(Suppl 4): 162. doi: 10.1186/s12859-018-2064-y.
|
| [19] |
LIEHR T, ACQUAROLA N, PYLE K, ST-PIERRE S, RINHOLM M, BAR O, et al. Next generation phenotyping in Emanuel and Pallister-Killian syndrome using computer-aided facial dysmorphology analysis of 2D photos[J]. Clin Genet, 2018, 93: 378-381. DOI:10.1111/cge.2018.93.issue-2 |
| [20] |
BANDA J M, SENEVIRATNE M, HERNANDEZ-BOUSSARD T, SHAH N H. Advances in electronic phenotyping:from rule-based definitions to machine learning models[J]. Annu Rev Biomed Data Sci, 2018, 1: 53-68. DOI:10.1146/annurev-biodatasci-080917-013315 |
| [21] |
RESNIK P. Using information content to evaluate semantic similarity in a taxonomy[C]//the 14th International Joint Conference on Artificial Intelligence (IJCAI-95). Morgan Kaufmann Publishers Inc., San Francisco, CA, USA, 1995: 448-453.
|
| [22] |
LIN D. An information-theoretic definition of similarity[C]. Proceedings of the Fifteenth International Conference on Machine Learning, 1998: 296-304.
|
| [23] |
PESQUITA C, FARIA D, BASTOS H, FALCO A, COUTO F M. Evaluating GO-based semantic similarity measures[C]. Proceedings of 10th Annual Bio-ontologies Meeting, 2007, 37: 38.
|
| [24] |
DRAGUSIN R, PETCU P, LIOMA C, LARSEN B, JORGENSEN H L, COX I J, et al. FindZebra:a search engine for rare diseases[J]. Int J Med Inform, 2013, 82: 528-538. DOI:10.1016/j.ijmedinf.2013.01.005 |
| [25] |
RAPPAPORT N, NATIV N, STELZER G, TWIK M, GUAN-GOLAN Y, STEIN T I, et al. MalaCards: an integrated compendium for diseases and their annotation[J/OL]. Database (Oxford), 2013, 2013: bat018. doi: 10.1093/database/bat018.
|
| [26] |
ROBINSON P N, KOHLER S, BAUER S, SEELOW D, HORN D, MUNDLOS S. The Human Phenotype Ontology:a tool for annotating and analyzing human hereditary disease[J]. Am J Hum Genet, 2008, 83: 610-615. DOI:10.1016/j.ajhg.2008.09.017 |
| [27] |
JAGADEESH K A, WENGER A M, BERGER M J, GUTURU H, STENSON P D, COOPER D N, et al. M-CAP eliminates a majority of variants of uncertain significance in clinical exomes at high sensitivity[J]. Nat Genet, 2016, 48: 1581-1586. DOI:10.1038/ng.3703 |
| [28] |
WIKIPEDIA. Clinical decision support system[EB/OL]. [2017-12-08]. https://en.wikipedia.org/wiki/Clinical_decision_support_system.
|
 2018, Vol. 39
2018, Vol. 39


