近几十年来,医学影像技术如计算机断层扫描(computed tomograhy,CT)、磁共振成像(magnetic resonance imaging,MRI)、正电子发射断层成像(positron emission tomography,PET)超声、X线等在疾病的检测、诊断和治疗中起着重要作用[1]。医学影像判读主要由放射科医师和临床医师等进行。然而,医师的经验存在较大的不稳定性,故希望能通过机器学习技术得到改进,使医师受益于计算机辅助。
在医学影像分析应用机器学习的过程中,有意义的特征提取是目标任务成功完成的核心。传统意义上或任务相关的特征大多基于目标领域中的人类专家知识。因此,非专家使用机器学习技术进行研究具有挑战性,而深度学习技术通过吸收学习过程中的特征工程步骤可有助于清除这一障碍[2]。也就是说人工提取特征,如需要再进行一定的预处理,然后输入数据和学习目标,深度学习技术就可以通过自学习的方式找到解决方案[3]。因此,特征提取工程的负担已从人转移到计算机,使非机器学习专家能有效利用深度学习技术进行医学影像等领域研究或应用。
深度学习技术能取得惊人成功得益于中央处理单元(CPU)和图形处理单元(GPU)计算能力的进步、大量数据的获得以及学习算法的发展[4]。从技术角度看,深度学习可以看作是通过建立两层以上的网络来改进传统的人工神经网络。研究表明,在深层神经网络中发现分层特征表示,从而可以从低层特征中提取高层特征[4]。由于具有从数据中学习分层特征的优良特性,深度学习已在各种人工智能应用中获得了优异的性能[5-6]。特别是计算机视觉领域的巨大进步启发其在医学图像分析中的应用,如图像分割[7-8]、图像配准[9]、图像融合[10]、图像标注[11]、辅助诊断和预后[12-13]、病变检测[14-15]和显微成像分析[16]。本文简要概述了几种深度学习方法,然后介绍了医学影像学中深度学习技术的相关应用研究,并讨论了深度学习技术在医学成像中的局限性。
1 深度学习方法概述深度学习是一类机器学习算法,它是利用多个级联的非线性处理单元进行特征提取和转换。每个连续层使用前一层的输出作为输入;学习(如分类)和(或)无监督(如模式分析)在监视环境下的行为;学习是与不同抽象层次相对应的多层次表示;概念的层次结构形成;反向传播训练并采用梯度下降法。深度学习的层次包括人工神经网络的隐含层和一组命题公式。它们也可能包括深生成模型中的潜在变量,如深信任网络中的节点和深度玻尔兹曼机。现有的深度学习技术包括前馈神经网络[17]、深度置信网络[18]、深度玻尔兹曼机[19]、深度卷积神经网络[20]等。
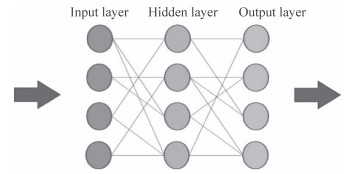
1.1 前馈神经网络前馈神经网络(图 1)是最简单的一种神经网络[17],其神经元分层排列,每个神经元只接收前一层相连神经元的输出,并输出给下一层,各层之间没有反馈。前馈神经网络的研究始于20世纪60年代,目前其理论研究和实际应用均已达到成熟阶段。对于前馈神经网络结构设计,一般采用直接定型法、修剪法和生长法。直接定型法是设计一个实际网络。修剪法由于要求从一个足够大的初始网络开始,所以整个过程复杂漫长,学习网络的训练只是最速下降优化过程,而对于超大初始网络,它不能保证一定收敛到全局最优解或足够好的局部最优解,因此,修剪法并不总是有效。生长法更符合人认识事物、积累知识的过程,具有自组织的特点,更具发展潜力。
|
图 1 前馈神经网络结构示意图 Fig 1 Schematic diagram of feedforward neural network structure |
前馈神经网络结构简单、应用广泛,能以任意精度逼近任意连续函数和平方可积函数,还可以精确实现任意有限训练样本集。从系统观点看,前馈神经网络是一种静态非线性映射,通过简单非线性处理单元的复合映射可获得复杂的非线性处理能力;从计算观点看,大部分前馈神经网络都是学习网络,其分类和模式识别能力一般强于反馈网络。
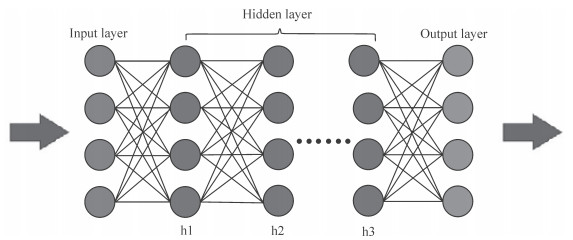
1.2 深度置信网络深度置信网络(图 2)既可用于非监督学习,类似于自编码机;也可用于监督学习,即作为分类器。当用于非监督学习时,其目的是尽可能保留原始特征的特点,同时降低特征的维度[18]。当用于监督学习时,其目的在于尽量降低分类错误率。深度置信网络的神经元组成元件是受限玻尔兹曼机。若干个受限玻尔兹曼机“串联”起来就构成了一个深度置信网络,其中上一个受限玻尔兹曼机的隐层即为下一个受限玻尔兹曼机的显层,上一个受限玻尔兹曼机的输出即为下一个受限玻尔兹曼机的输入。训练过程中,需充分训练上一层的受限玻尔兹曼机后才能训练当前层的受限玻尔兹曼机,直至最后一层。
|
图 2 深度置信网络结构示意图 Fig 2 Schematic diagram of deep confidence network structure |
1.3 深度玻尔兹曼机
全部用受限玻尔兹曼机形成的深层模型即为深度玻尔兹曼机[19]。如果将靠近输入层的部分替换为贝叶斯置信网络,即有向图模型,而远离输入层的部分仍使用受限玻尔兹曼机,则构成深度置信网络。
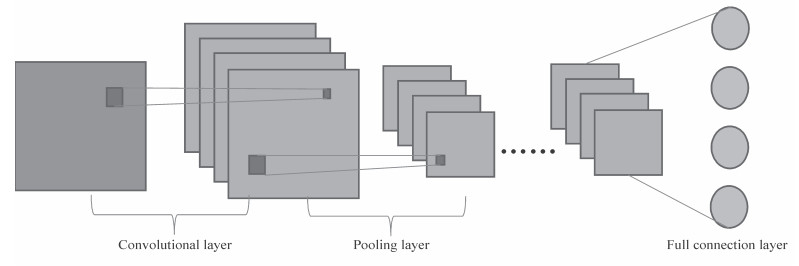
1.4 深度卷积神经网络卷积神经网络是一种前馈神经网络,其以卷积核构成的神经元可以响应一部分覆盖范围内的周围单元,对于图像处理有出色表现[21]。它包括卷积层和池化层。一般卷积神经网络的基本结构包括两层:(1)特征提取层,每个神经元的输入与前一层局部区域相连,并提取该局部的特征,一旦该局部特征被提取,它与其他特征间的位置关系也即确定下来;(2)特征映射层,网络的每个计算层由多个特征映射组成,每个特征映射是一个平面,平面上所有神经元的加权赋值相等。特征映射结构采用影响函数核小的函数(如sigmod)作为卷积神经网络的激活函数,使特征映射具有位移不变性。此外,一个映射面上的所有神经元共享权值减少了网络自由参数的个数。卷积神经网络中的每个卷积层都紧跟着一个用来求局部平均与二次提取的计算层,这种特有的2次特征提取结构减小了特征分辨率。以多层卷积神经网络作为隐藏层即构成了深度卷积神经网络(图 3)。
|
图 3 深度卷积神经网络结构示意图 Fig 3 Schematic diagram of deep convolutional neural network structure |
2 深度学习技术在医学影像中的应用 2.1 分类
图像或检测分类是深度学习的第一个应用领域之一,其对医学图像分析有着重大贡献。在分类检测中,一个或多个图像(检测)通常被用作输入,诊断变量(如是否存在疾病)作为输出。在这种环境中每一个诊断测试都是一个示例,数据集通常小于计算机视觉(如数百个样本)的大小,有力推动了该应用程序迁移学习的普及。
迁移学习本质是使用预先训练好的网络(通常是在自然图像中),试图围绕大网络训练的大数据集(感知)的需求。学习策略的两种迁移,即利用预先培训的网络作为特征提取器和通过医疗数据网络预调或微调已被广泛使用。大量研究使用体系结构来利用医疗数据的独特属性。Hosseini-Asl等[22]使用了三维卷积神经网络代替二维卷积神经网络来对阿尔茨海默病患者进行分类;Kawahara等[23]在磁共振弥散张量成像的脑连接图中应用类似卷积神经网络的结构进行分类,他们开发了几个新的层形成特有的网络结构,进而使用该网络结构预测大脑的发展,结果表明新的网络结构在评估认知和运动成绩方面优于现有的方法。
2.2 检出对影像中感兴趣的物体或病变进行检测是诊断的关键部分,也是临床医师最密集的工作之一。一般任务包括完整图像空间中小病灶的位置确定和识别。在计算机辅助检测系统中有一个长期的传统研究,即自动检测病变,其可以提高检测精度或减少人类专家的阅读时间。
大多数深度学习目标检测系统仍使用神经网络对像素进行分类,并以某种形式的后处理获得候选。如Teramoto等[24]使用多流卷积神经网络检测集成CT和PET数据。Dou等[25]使用三维卷积神经网络在MRI图像中检测微出血。
目标检测与目标分类区别明显,原因是每个像素被分类,类平衡在训练设置中对非对象类有很大的偏向。此外,大多数非物理样本容易受到歧视,阻碍了挑战性样本中深度学习方法的集中。van Grinsven等[26]提出选择性数据采样,其中错误分类的样本常反馈到网络上以聚焦视网膜图像中具有挑战性的区域;然后,采用滑动窗口方式进行结果级冗余计算,对每个像素进行命令分类。Wolterink等[27]的研究也是对象检出方法的一个重要方面。
2.3 分割医学图像中器官与其他子结构的分割是定量分析与体积和形状有关的临床参数的基础,其是计算机辅助检测中重要的第一步。分割任务通常被定义为确定一组像素,这些像素构成感兴趣对象的轮廓或内部。分割是医学影像深入研究的最普遍的主题,也是最广泛使用的方法,包括深度学习方法的应用。
Dan等[28]最早使用深度学习算法进行医学图像分割,他们在电子显微镜图像中采用逐像素分割的方法对滑动窗口中的图像进行分割。Ronneberger等[29]发表的U-net方法是最著名的分割方法之一。U-net两个主要结构的特点是结合了等量的上采样和下采样层,同时网络结合了所谓的跳跃的相对卷积和反褶积,从训练角度看整个图像可以向前方式处理,从而在分割中直接映射。这种方法允许我们利用基于块的细胞神经网络,同时考虑到图像的完整性。Çiçek等[30]的研究显示可以用U-net完成三维分割。此外,还有其他基于U-net的分割研究,如Milletari等[31]在U-net基础上改良得V-net;Drozdzal等[32]研究用短程的残差网络修正长程的U-net结构。这些特定的分割方法有助于获得良好的分割结果和训练神经网络。
2.4 配准医学图像的配准(即空间排列)是一种常见的图像分析任务,其中坐标变换计算从一个图像到另一个图像。这通常是一种优化的迭代框架中的一个特定类型的(非)参数变换假设和RIC(如L2范数)是预定义的。目前使用比较广泛的策略是利用深层学习网络的两种图像驱动迭代优化策略相似性度量和直接回归参数的深度回归网络。
目前已有部分通过深度学习技术来优化配准算法的尝试[33-35]。Cheng等[34]使用2个堆叠的自动编码器评估头部CT和MRI图像之间的局部相似性,该自动编码器使用CT和MRI图像的矢量化,通过4层进行修复和重建;在使用无监督补丁重构网络进行预培训后,使用2个预测层对它们进行微调,并堆叠在SAE的第3级上。这些预测层用于确定这2个补丁是相似的(第1类)还是不相似的(第2类)。Simonovsky等[35]使用类似策略以不同的方式估计2张图像之间的相似性;同时,他们还提出了一种利用这一测度的导数直接优化转换参数的方法,这种转换参数是从网络本身分离出来的。Miao等[36]利用卷积神经网络通过二维X射线配准的三维模型来评估植入物的位置,结果发现其配准成功率高于传统以纯强度为基础的配准方法。Yang等[37]使用脑MRI图像中电流/电流配准的绿洲数据集,他们是基于大变形的微分同胚映射(LDDMM)配准方法。
不同于分类和分割的研究,目前学术界似乎并没有确定在配准方法中整合深度学习技术的最佳途径,相关研究不多,现有研究方法也不尽相同。因此,建议什么是最有前途的方法似乎不太合适,有待将来对医学图像配准进行更深入的研究。
3 小结医学图像分析领域已开始注意深度学习技术在这些关键问题上的发展。然而,从基于人工的系统向从数据中学习特征的系统的过渡是渐进的。Shen等[20]对医学图像分析中深度学习的应用进行了综述。虽然他们进行了大量的工作,但仅从计算机角度来理解似乎是有缺陷的。近年来,深度学习技术的新进展为医学图像分析提供了新思路,它只允许从数据中发现图像中的形态和(或)纹理图案。虽然深度学习技术在许多医学应用研究中似乎已达到最先进的性能,但少有结果表明它能超越传统方法。同时,目前深度学习在医学图像分析领域中的研究还是以技术为主,所采用的评估指标也是计算机领域的评估指标。对于任何一项医学应用,我们更希望看到对于相关技术按照医学规则进行评估,例如通过多中心、随机、对照研究的方法证明该技术比之前的技术有更显著的优势。
此外,医学图像训练中的一个重要挑战是大多数深度模型的训练样本数目与学习参数的数量有关。因此,如何减少过拟合一直是一个问题。当深度学习的训练成果被送到新的中心应用时,模型需要迁移学习以保持性能,这种限制无疑导致了临床应用的低重复性。而稳定性和可重复性是该技术在临床可以被广泛推广使用的基本前提,因此,深度学习领域的相关研究者应当关注算法在前瞻性样本中的可重复性如何得到保障。
此外,用于训练的数据质量也是引起灾难性结果的另一原因。随机噪声的问题比较容易解决,它可以通过某些参数设置提高性能(技术上称为标签的平滑或软标签)。结构性噪声是不同的,它增加了一个真正不同的信号,将实实在在影响模型学习。Rolnick等[38]研究证明标签结构噪声将导致训练结果的性能严重下降。当噪声来自与实际数据相同的源时问题就更严重,因为模型会混淆类,目前深度学习方法的黑箱问题会被无限放大。
除了这些技术上的问题,理论上的障碍似乎更麻烦。目前的深度学习方法并不具有因果逻辑性,仅是相关性的计算,这些方法可能对他们所能执行的认知任务有内在限制[39]。而达成我们所期望的性能目标需要消耗的资源价值也要谨慎评估,以避免掉入非多项式时间问题的陷阱。
综上,深度学习技术为机器学习最重要的特征提取带来了新方法,如果深度学习技术能在所有问题上表现良好,将为医学影像分析处理带来巨大的帮助。然而,深度学习技术并不是终极算法,它只是人工智能几个学派中连接学派的代表,其性能极限需要被合理评估。深度学习过度依赖高质量大数据,对于医学图像来说其经济学效应未必合适。对于深度学习技术适合应用领域的拓展必须非常谨慎,应在恰当的使用场景采用相关技术,避免落入陷阱。
| [1] |
BRODY H. Medical imaging[J]. Nature, 2013, 502: S81. DOI:10.1038/502S81a |
| [2] |
SCHMIDHUBER J. Deep learning in neural networks:an overview[J]. Neural Netw, 2015, 61: 85-117. DOI:10.1016/j.neunet.2014.09.003 |
| [3] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521: 436-444. DOI:10.1038/nature14539 |
| [4] |
HINTON G E, SALAKHUTDINOV R R. Reducing the dimensionality of data with neural networks[J]. Science, 2006, 313: 504-507. DOI:10.1126/science.1127647 |
| [5] |
KARPATHY A, FEI-FEI L. Deep visual-semantic alignments for generating image descriptions[J]. IEEE Trans Pattern Anal Mach Intell, 2017, 39: 664-676. DOI:10.1109/TPAMI.2016.2598339 |
| [6] |
SILVER D, HUANG A, MADDISON C J, GUEZ A, SIFRE L, VAN DEN DRIESSCHE G, et al. Mastering the game of Go with deep neural networks and tree search[J]. Nature, 2016, 529: 484-489. DOI:10.1038/nature16961 |
| [7] |
ZHANG W, LI R, DENG H, WANG L, LIN W, JI S, et al. Deep convolutional neural networks for multi-modality isointense infant brain image segmentation[J]. Neuroimage, 2015, 108: 214-224. DOI:10.1016/j.neuroimage.2014.12.061 |
| [8] |
KLEESIEK J, URBAN G, HUBERT A, SCHWARZ D, MAIER-HEIN K, BENDSZUS M, et al. Deep MRI brain extraction:a 3D convolutional neural network for skull stripping[J]. Neuroimage, 2016, 129: 460-469. DOI:10.1016/j.neuroimage.2016.01.024 |
| [9] |
WU G, KIM M, WANG Q, MUNSELL B C, SHEN D. Scalable high-performance image registration framework by unsupervised deep feature representations learning[J]. IEEE Trans Biomed Eng, 2016, 63: 1505-1516. DOI:10.1109/TBME.2015.2496253 |
| [10] |
SUK H I, LEE S W, SHEN D; Alzheimer's Disease Neuroimaging Initiative. Hierarchical feature representation and multimodal fusion with deep learning for AD/MCI diagnosis[J]. Neuroimage, 2014, 101: 569-582. DOI:10.1016/j.neuroimage.2014.06.077 |
| [11] |
JIU M, SAHBI H. Nonlinear deep kernel learning for image annotation[J/OL]. IEEE Trans Image Process, 2017. doi: 10.1109/TIP.2017.2666038.[Epubaheadofprint].
|
| [12] |
SUK H I, LEE S W, SHEN D; Alzheimer's Disease Neuroimaging Initiative. Latent feature representation with stacked auto-encoder for AD/MCI diagnosis[J]. Brain Struct Funct, 2015, 220: 841-859. DOI:10.1007/s00429-013-0687-3 |
| [13] |
SUK H I, WEE C Y, LEE S W, SHEN D. State-space model with deep learning for functional dynamics estimation in resting-state fMRI[J]. Neuroimage, 2016, 129: 292-307. DOI:10.1016/j.neuroimage.2016.01.005 |
| [14] |
VAN TULDER G, DE BRUIJNE M. Combining generative and discriminative representation learning for lung CT analysis with convolutional restricted Boltzmann machines[J]. IEEE Trans Med Imaging, 2016, 35: 1262-1272. DOI:10.1109/TMI.2016.2526687 |
| [15] |
DOU Q, CHEN H, YU L, ZHAO L, QIN J, WANG D, et al. Automatic detection of cerebral microbleeds from mr images via 3D convolutional neural networks[J]. IEEE Trans Med Imaging, 2016, 35: 1182-1195. DOI:10.1109/TMI.2016.2528129 |
| [16] |
CIREŞAN D C, GIUSTI A, GAMBARDELLA L M, SCHMIDHUBER J. Mitosis detection in breast cancer histology images with deep neural networks[J]. Med Image Comput Comput Assist Interv, 2013, 16(Pt 2): 411-418. |
| [17] |
BEBIS G, GEORGIOPOULOS M. Feed-forward neural networks[J]. IEEE Potentials, 1994, 13: 27-31. DOI:10.1109/45.329294 |
| [18] |
HINTON G. Deep belief nets[M]//SAMMUT C, WEBB G I. Encyclopedia of machine learning. Springer, 2011: 267-269.
|
| [19] |
SALAKHUTDINOV R, HINTON G. Deep Boltzmann machines[J]. J Machine Learning Res, 2009, 5: 1967-2006. |
| [20] |
SHEN D, WU G, SUK H I. Deep learning in medical image analysis[J]. Annu Rev Biomed Eng, 2017, 19: 221-248. DOI:10.1146/annurev-bioeng-071516-044442 |
| [21] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[C]//Proceedings of the 25th International Conference on Neural Information Processing Systems-Volume 1. Curran Associates Inc., USA, 2012: 1097-1105.
|
| [22] |
HOSSEINI-ASL E, GHAZAL M, MAHMOUD A, ASLANTAS A, SHALABY A M, CASANOVA M F, et al. Alzheimer's disease diagnostics by a 3D deeply supervised adaptable convolutional network[J]. Front Biosci (Landmark Ed), 2018, 23: 584-596. DOI:10.2741/4606 |
| [23] |
KAWAHARA J, BROWN C J, MILLER S P, BOOTH B G, CHAU V, GRUNAU R E, et al. BrainNetCNN:convolutional neural networks for brain networks; towards predicting neurodevelopment[J]. Neuroimage, 2017, 146: 1038-1049. DOI:10.1016/j.neuroimage.2016.09.046 |
| [24] |
TERAMOTO A, FUJITA H, YAMAMURO O, TAMAKI T. Automated detection of pulmonary nodules in PET/CT images:ensemble false-positive reduction using a convolutional neural network technique[J]. Med Phys, 2016, 43: 2821-2827. DOI:10.1118/1.4948498 |
| [25] |
DOU Q, CHEN H, YU L, QIN J, HENG A. Multilevel contextual 3-D CNNs for false positive reduction in pulmonary nodule detection[J]. IEEE Trans Biomed Eng, 2017, 64: 1558-1567. DOI:10.1109/TBME.2016.2613502 |
| [26] |
VAN GRINSVEN M J, VAN GINNEKEN B, HOYNG C B, THEELEN T, SANCHEZ C I. Fast convolutional neural network training using selective data sampling:application to hemorrhage detection in color fundus images[J]. IEEE Trans Med Imaging, 2016, 35: 1273-1284. DOI:10.1109/TMI.2016.2526689 |
| [27] |
WOLTERINK J M, LEINER T, DE VOS B D, VAN HAMERSVELT R W, VIERGEVER M A, IŠGUM I. Automatic coronary artery calcium scoring in cardiac CT angiography using paired convolutional neural networks[J]. Med Image Anal, 2016, 34: 123-136. DOI:10.1016/j.media.2016.04.004 |
| [28] |
CIREŞAN D C, GIUSTI A, GAMBARDELLA L M, SCHMIDHUBER J. Deep neural networks segment neuronal membranes in electron microscopy images[J]. Adv Neural Inform Proc Sys, 2012, 25: 2852-2860. |
| [29] |
RONNEBERGER O, FISCHER P, BROX T. U-Net:convolutional networks for biomedical image segmentation[M]. Switzerland: Springer International Publishing, 2015: 234-241.
|
| [30] |
ÇIÇEK Ö, ABDULKADIR A, LIENKAMP S S, BROX T, RONNEBERGER O. 3D U-Net: learning dense volumetric segmentation from sparse annotation[Z/OL]. arXiv: 1606. 06650, 2016. http://cn.arxiv.org/pdf/1606.06650v1.
|
| [31] |
MILLETARI F, NAVAB N, AHMADI S A. V-Net: fully convolutional neural networks for volumetric medical image segmentation[C/OL]//2016 Fourth International Conference on 3D Vision (3DV). Stanford, CA, USA, 2016: 565-571. doi: 10.1109/3DV.2016.79.
|
| [32] |
DROZDZAL M, VORONTSOV E, CHARTRAND G, KADOURY S, PAL C. The importance of skip connections in biomedical image segmentation[Z/OL]. arXiv: 1608. 04117, 2016. http://cn.arxiv.org/pdf/1608.04117v2.
|
| [33] |
WU G, KIM M, WANG Q, GAO Y, LIAO S, SHEN D. Unsupervised deep feature learning for deformable registration of MR brain images[J]. Med Image Comput Comput Assist Interv, 2013, 16(Pt 2): 649-656. |
| [34] |
CHENG X, ZHANG L, ZHENG Y. Deep similarity learning for multimodal medical images[C]//MCCAI 2016: International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer, 2016: 1-6.
|
| [35] |
SIMONOVSKY M, GUTIÉRREZ-BECKER B, MATEUS D, NAVAB N, KOMODAKIS N. A deep metric for multimodal registration[Z/OL]. arXiv: 1609. 05396, 2017. http://cn.arxiv.org/pdf/1609.05396v1.
|
| [36] |
MIAO S, WANG Z J, LIAO R. A CNN regression approach for real-time 2D/3D registration[J]. IEEE Trans Med Imaging, 2016, 35: 1352-1363. DOI:10.1109/TMI.2016.2521800 |
| [37] |
YANG X, KWITT R, NIETHAMMER M. Fast predictive image registration[M]. Switzerland: Springer International Publishing, 2016: 48-57.
|
| [38] |
ROLNICK D, VEIT A, BELONGIE S, SHAVIT N. Deep learning is robust to massive label noise[Z/OL]. arXiv: 1705. 10694, 2017. http://cn.arxiv.org/pdf/1705.10694v3.
|
| [39] |
PEARL J. Theoretical impediments to machine learning with seven sparks from the causal revolution[Z/OL]. arXiv: 1801. 04016, 2017. http://cn.arxiv.org/pdf/1801.04016v1.
|
 2018, Vol. 39
2018, Vol. 39


