2. 上海中医药大学附属龙华医院肿瘤七科, 上海 200032;
3. 解放军81医院全军肿瘤中心肿瘤内科, 南京 210002
2. Department of Oncology(Ⅶ), Longhua Hospital Affiliated to Shanghai University of Traditional Chinese Medicine, Shanghai 200032, China;
3. Department of Medical Oncology, Cancer Center of PLA, No. 81 Hospital of PLA, Nanjing 210002, Jiangsu, China
人工智能是计算机科学发展和超快运算速度的必然产物,其在医疗实践中的应用前景是热点话题,已被应用于医学影像、药物发现、急诊室/医院管理、健康管理、可穿戴设备、风险评估等领域。人工智能技术在医学领域最成功的应用是影像学、病理学和皮肤学的图像分析,其分析速度快,准确度可与临床医学专家媲美[1]。病理图像是疾病诊断的金标准,病理学家通过评估标本的大小、形状、有丝分裂计数、颜色和纹理等属性进行诊断决策[2]。神经网络一直被探索应用于图像分析和自动诊断的复杂过程[2-3]。近年研究发现,基于卷积神经网络的深度学习技术适用于图像分析,其在疾病的早期检测和组织分类方面具有重要价值,如在乳腺癌前哨淋巴结活组织检查中检测转移性癌细胞[4]和在制定乳腺癌与胃癌治疗策略前对人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)染色进行评分[5]。
组织切片数字化是病理人工智能技术发展的前提。数字病理切片是将整张载玻片进行全信息、全方位快速扫描,使物质化的玻璃载玻片变成数字化的病理切片。传统的手术活组织检查和切除标本的病理诊断主要通过光学显微镜观察组织玻片,其成本低、简洁、方便,但难以一次观察整张玻片,只能观察局部视野,而整张玻片数字化技术的进步使显微图像可作为整体呈现在一个可在屏幕上计算和可视化的数字矩阵中。既往研究证明,全玻片成像可用于手术切除或活组织检查样本的诊断和评估,包括H-E染色、免疫组织化学染色及特殊染色[6]。全玻片成像技术产生了大量被称为“大数据”的数字组织玻片,可用于远程病理、会诊、教育、图像计算和科学研究。大数据病理信息结合深度学习为自动诊断、自动免疫组织化学和科学研究带来机遇[3]。病理学包括人类病理学、动物病理学和植物病理学。人类、动物和植物的所有组织切片都可以数字化,然后通过人工智能算法进行评估。本文总结了本研究小组目前在这些领域的初步工作,旨在探讨它们在临床和研究领域的前景。
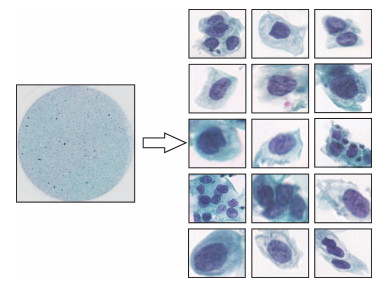
1 数字化全玻片结合人工智能技术为细胞病理学的决策支持工具细胞病理学是一门相对较新的医学学科,通常用于宫颈癌前病变、体腔内液体(腹膜液、胸膜液、心包液和脑脊液),以及癌变或感染病变组织的研究。现阶段细胞病理学的诊断主要基于肉眼观察,即在显微镜下观察玻片中的所有细胞,并根据细胞形态进行诊断。如图 1所示,宫颈癌细胞涂片中有上千个细胞,病理医师对每个细胞进行评估的工作量巨大,且易于疲劳,从而导致漏诊。同时,肿瘤筛查过程中会产生大量玻片,其储存难度大,而且阴性玻片会定时销毁,阳性玻片质量也会随时间推移而下降。数字化全玻片结合人工智能技术可以较好地解决上述难题。首先,细胞涂片数字化解决了玻片的储存和质量问题,而数字化的病理信息可用于深度学习,进而帮助医师解决评估工作量大的问题。细胞病理学诊断在很大程度上依赖于图像,而人工智能技术的应用基于图像特征,包括细胞核形态特征和基于像素的特征。基于上述特点的人工神经网络(artificial neural network,ANN)在胃肠系统、甲状腺、乳腺、泌尿系统、积液的细胞病理学诊断中均已得到广泛应用,为决策支持提供了依据。特别是用于宫颈癌检测的ANN,自动细胞学筛查系统PAPNET是目前宫颈癌检测应用最成功的ANN模型之一。与常规人工筛查相比,PAPNET系统的阳性或疑似病例检出率高出20%,假阴性率降低约2倍;人工筛查为假阴性的玻片中有1/3被重新归类为异常[7]。然而,有关ANN在细胞病理学中应用的报道在2000年达到高峰,随后逐渐减少[8],推测原因可能与过度拟合、难以获得形态测量数据、错误分类和计算能力有关。计算机科学的最新进展将有助于解决这一难题并加快研发速度。Song等[9]采用了基于超像素卷积神经网络的子宫颈癌细胞分割方法,该方法识别核区域的准确度达94.50%,细胞核分割的精度和召回率分别为0.914 3±0.020 2、0.872 6±0.000 8。本研究小组在TensorFlow框架中独立开发了一种人工智能算法,结合锚盒、金字塔提取、Dropout等深度学习技术提高了识别率和定位精度,降低了训练模型所需的样本量,该算法在辅助诊断中使用分布式计算分析15 mm×15 mm切片(像素大小为40 000×40 000)的速度约为100 s(图 1)。
|
图 1 数字化全玻片结合人工智能技术用于宫颈癌早期筛查 Fig 1 Early screening of cervical cancer identified by digital whole slide combined with artificial intelligence technology Left: Digital smear of cervical cancer cells; Right: Representative alien cells identified by artificial intelligence. Original magnification: ×400 (right) |
2 数字化全玻片结合人工智能技术提高了病理诊断的准确性和效率
病理学家的常规工作是基于组织切片的专业评估,并将最终的诊断和证据转化为病理报告。对大多数病理学家而言,这是一项耗时的工作,可以称之为秘书性工作。秘书模拟人工智能可智能、半自主执行许多秘书任务,其输出特点是快速、一致和可持续改进[10]。然而,数字化全玻片成像结合人工智能技术能否提高诊断的准确性和效率呢?癌细胞区域检测竞赛(camelyon16)是通过深度学习算法检测乳腺癌患者的淋巴结转移情况,在模拟常规病理工作流程时,深度学习算法可获得比病理专家更好的诊断性能,同时其解释整张切片的性能也可媲美病理专家[11]。自动组织图像分析系统已在各种肿瘤中得到广泛探索,可用于肿瘤细胞的检测、组织分类、早期检测、肿瘤生物标志物评分等[9, 12-14],这些研究表明在数字化全玻片的基础上,利用基于自动组织图像分析系统的人工智能技术协助病理学家提高常规病理诊断质量具有良好的应用前景。
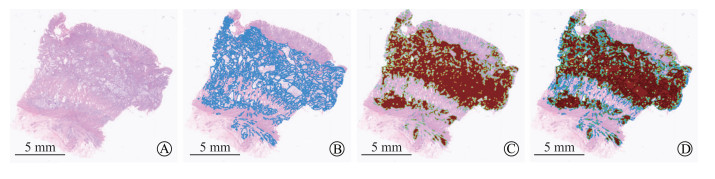
本研究小组训练了基于PyTorch的卷积神经网络,从胃活组织检查和切除标本中检测胃癌细胞。首先建立数据集,纳入了200张胃癌的数字全玻片,每张提取10个典型区域,大小为2 048×2 048像素,对典型区域内的所有肿瘤细胞进行精准标注。然后对数字切片图像进行预处理,提取组织区域;利用训练后的网络完成组织区域的分类,即将组织区域分为多个给定大小的块并对这些块进行分类;最后对各部分结果进行重叠处理,得到位置对应的结果。为每张玻片生成可视化的热力学图,整张玻片图像处理时间为2~10 min。将H-E染色的原组织切片(图 2A)通过人工标记出所有肿瘤细胞(图 2B),同时另外使用人工智能算法处理原组织切片并生成热力学图(图 2C),结果发现人工智能算法的检测精度为97.73%(图 2D)。有研究利用自动图像分析检测获得的胃活组织检查标本的灵敏度、特异度、阳性预测值和阴性预测值分别为89.5%、50.7%、47.7%和90.6%,显示了人工智能技术在筛查胃镜样本中的应用潜力[13]。虽然在病理诊断和肿瘤筛查中应用人工智能技术有一定的局限性,但基于数字化病理切片的人工智能诊断应用前景广阔。
|
图 2 人工智能技术识别胃癌病理切片中的肿瘤细胞 Fig 2 Artificial intelligence technology in identifying tumor cells in whole slide of gastric cancer A: Pathological slide of gastric cancer; B: Manually labeled pathological slide; C: Pathological slide identified by artificial intelligence technology; D: Merged pathological slide |
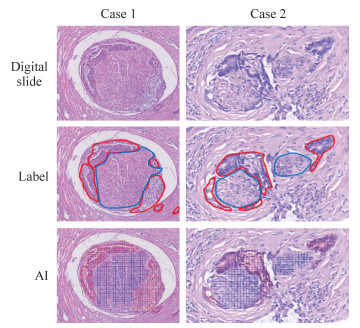
病理报告是疾病诊断的客观描述,其中最重要的描述是确定其是否为肿瘤。此外,还需描述肿瘤的其他重要特征,如肿瘤分化、血管浸润、周围神经浸润、肿瘤栓子、肿瘤细胞百分比等,这些特征在决定治疗策略和预测患者预后方面起着关键作用。既往光学显微镜观察只能提示是否存在病理特征,难以直观展示上述特征的全貌和空间关系,使用数字全玻片扫描系统将病理玻片数字化后,通过人工智能技术能清晰显示病理特征的全貌,还能反映不同特征的空间分布。周围神经浸润是肝门部胆管癌复发和预后差的重要预测因素[15],基于数字化病理切片,训练后的人工智能算法能成功识别病理图像中的神经细胞和浸润的肿瘤细胞,相比人工标注更具优势,对于理解神经侵犯的发生机制和判断预后有积极作用。见图 3。
|
图 3 人工智能技术识别数字病理切片中的肿瘤细胞和神经细胞 Fig 3 Artificial intelligence (AI) technology in identifying tumor cells and nerve cells in digital pathological slide Red indicated tumor cells, and blue indicated nerve cells. Original magnification: ×200 |
3 数字化全玻片结合免疫组织化学自动评分系统
免疫组织化学是检测组织样本中某些蛋白的表达水平和表达模式的技术,用于肿瘤诊断与治疗和科学研究的临床应用。免疫组织化学可检测细胞膜(HER2)、细胞质和细胞核[Ki-67和雌激素受体(estrogen receptor,ER)]中的生物标志物。目前,基于人工智能技术的HER2自动评分方法已得到深入发展[5]。在71例乳腺肿瘤切除样本中,基于人工智能技术的自动评分与病理学家评估结果的一致性为83%;经复查,8例不一致病例的最终诊断得到纠正[12]。对于ER等生物标志物,人工智能算法可以自动评估其大小、形状等特征,从而对细胞核进行正确的分割。模糊c均值算法可识别93.1%的细胞核,其分类灵敏度和特异度分别为95.7%和93.2%[16]。Ki-67指数是深度学习模型的另一主要贡献者,具有高度一致性、重现性和准确性[17]。基于数字组织切片的深度学习技术辅助评分系统可以促进临床决策,并可融入到病理日常工作流程中。
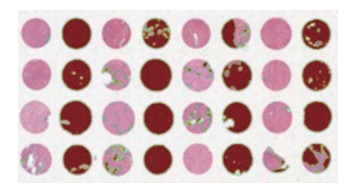
免疫组织化学的科学研究表明,病理图像中有大量预测和预后数据。挖掘“亚视觉”图像特征并对阳性染色细胞进行评分可以量化疾病的外观,并可用于预测疾病侵袭性和评估患者预后。除了用于常规大切片的免疫组织化学染色,数字化全玻片对于组织芯片免疫组织化学染色的判读具有重要作用。传统的组织芯片免疫组织化学染色评估主要依赖医师的肉眼观察和经验,耗时耗力,而且不同医师的判读结果差异较大。本研究小组试图利用人工智能技术解决组织芯片免疫组织化学染色的判读问题,初步研究发现,人工智能技术可快速识别组织芯片中的肿瘤细胞和阳性染色细胞。正常情况下,人工阅读一张组织芯片需要1~2 h,而基于数字组织切片的人工智能技术则能缩短到几分钟。图 4所示为人工智能技术识别组织芯片中的肿瘤细胞,深红色代表人工智能技术识别出的肿瘤细胞。
|
图 4 人工智能技术识别组织芯片中的肿瘤细胞 Fig 4 Tumor cells identified by artificial intelligence technology in tissue microarray Red indicated tumor tissues |
4 数字化全玻片结合人工智能技术客观评价动物模型的病理形态学变化
动物模型是生物医学研究的重要实验对象和材料,其在探索和验证现代生物医学实验方法,了解人类疾病发展、预防和控制策略等方面具有重要意义。成熟的肿瘤动物模型可用于评价抗肿瘤免疫治疗、抗肿瘤药物、抗肿瘤转移和抗肿瘤转移药物的疗效。在用动物模型进行研究时,常规工作流程是获取所需的样本并制成切片,病理学家对这些切片进行评估,研究人员阅读所有玻片并根据相关诊断标准给出最终报告。这种报告通常具有主观性,不同研究者间常发生分歧,因此客观评价十分必要。
数字化动物模型的组织切片和免疫组织化学切片后,利用深度学习算法分析病理图像,可以提高诊断的准确性和效率。本研究小组复制了一个多步骤致胆管癌动物模型[18],该模型在诱导后20周时发展成100%浸润肠型胆管癌和多焦点胆管增生,可获取其数字病理切片。同时,本研究小组开发了一种基于卷积神经网络的自动病变检测框架,用于检测和定位组织区域的纤维化和肿瘤区域。该系统可以热力学图的形式显示肝脏所有病变,并提供病变与全切片的精确比例(图 5)。
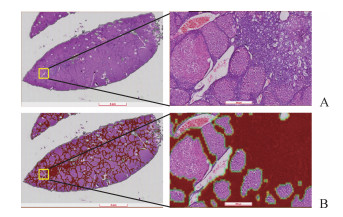
|
图 5 人工智能技术识别数字病理图片中的肝脏病变区域 Fig 5 Artificial intelligence technology in identifying liver lesions in digital pathological imaging A: Digital whole slide of liver; B: Lesion area (red tissue) of liver identified by artificial intelligence technology. Left: Whole slide; right: Local magnification of left imaging. Original magnification: ×200 (right) |
5 人工智能技术提取植物内部特征
中药质量是其临床疗效的基础,基于质量标准的中医智能评价策略为中医药质量管理提供了新视角[19]。传统的质量标记主要关注独特的中药材差异、动态变化及其运输和可追溯性。本研究小组认为草药内部形态结构有助于鉴别中药材质量。每种中药都有其独特的纹理,可以通过自动病理切片扫描仪进行数字化。如图 6所示,数字化太子参和麦冬切片后,显微镜下可见两者的纹理不同。人工智能技术可以识别不同植物的纹理,有助于判断植物的种类和真假,甚至能推测药物的作用部位。

|
图 6 数字化植物切片的展示形式 Fig 6 Representative pictures of digital plant slices A: Radix Pseudostellariae; B: Radix Ophiopogonis |
6 小结
数字化病理切片实现了病理诊断和科研服务划时代的变革。临床工作中观察数字化玻片等同于显微镜下观察,可使病理科医师脱离显微镜,适时通过网络进行病理诊断,有望实现全球在线同步远程会诊或离线远程会诊,体现时间和空间穿插传递的优势。此外,全玻片数字化有利于实现病理切片多层三维重建和病理切片管理数字化,而数字化病理切片图像则大大促进了人工智能技术在医疗领域的发展。
全玻片数字化能提供大量可任意放大和精准标注的数据,最终形成丰富的便于深度学习的数据集,与人工智能技术和计算机强大的运算能力结合后将会产生与疾病诊断和科研服务相关的各类产品,自动检测数字化切片中的病变区域并量化各项指标,在人体病理、动物病理和植物病理等领域将发挥巨大作用。
志谢 感谢上海纳凝微信息科技有限公司和微瞰智能科技(上海)有限公司在肝门部胆管癌和胃癌人工智能识别方面给予的帮助;感谢上海舟朴医疗科技发展有限公司和宁波江丰电子材料有限公司在病理切片数字化方面给予的帮助。
| [1] |
MILLER D D, BROWN E W. Artificial intelligence in medical practice:the question to the answer?[J]. Am J Med, 2018, 131: 129-133. DOI:10.1016/j.amjmed.2017.10.035 |
| [2] |
NAZERAN H, RICE F, MORAN W, SKINNER J. Biomedical image processing in pathology:a review[J]. Australas Phys Eng Sci Med, 1995, 18: 26-38. |
| [3] |
KAYSER K, GORTLER J, BOGOVAC M, BOGOVAC A, GOLDMANN T, VOLLMER E, et al. AI (artificial intelligence) in histopathology-from image analysis to automated diagnosis[J]. Folia Histochem Cytobiol, 2009, 47: 355-361. |
| [4] |
HOLTEN-ROSSING H, TALMAN M M, JYLLING A M B, LAENKHOLM A V, KRISTENSSON M, VAINER B. Application of automated image analysis reduces the workload of manual screening of sentinel lymph node biopsies in breast cancer[J]. Histopathology, 2017, 71: 866-873. DOI:10.1111/his.2017.71.issue-6 |
| [5] |
QAISER T, MUKHERJEE A, REDDY PB C, MUNUGOTI S D, TALLAM V, PITKAAHO T, et al. HER2 challenge contest:a detailed assessment of automated HER2 scoring algorithms in whole slide images of breast cancer tissues[J]. Histopathology, 2017, 72: 227-238. |
| [6] |
MUKHOPADHYAY S, FELDMAN M D, ABELS E, ASHFAQ R, BELTAIFA S, CACCIABEVE N G, et al. Whole slide imaging versus microscopy for primary diagnosis in surgical pathology:a multicenter blinded randomized noninferiority study of 1992 cases (pivotal study)[J]. Am J Surg Pathol, 2018, 42: 39-52. |
| [7] |
ABULAFIA O, SHERER D M. Automated cervical cytology:meta-analyses of the performance of the PAPNET system[J]. Obstet Gynecol Surv, 1999, 54: 253-264. DOI:10.1097/00006254-199904000-00022 |
| [8] |
POULIAKIS A, KARAKITSOU E, MARGARI N, BOUNTRIS P, HARITOU M, PANAYIOTIDES J, et al. Artificial neural networks as decision support tools in cytopathology:past, present, and future[J]. Biomed Eng Comput Biol, 2016, 7: 1-18. |
| [9] |
SONG Y, ZHANG L, CHEN S, NI D, LI B, ZHOU Y, et al. A deep learning based framework for accurate segmentation of cervical cytoplasm and nuclei[J]. Conf Proc IEEE Eng Med Biol Soc, 2014, 2014: 2903-2906. |
| [10] |
YE J J. Artificial intelligence for pathologists is not near-it is here:description of a prototype that can transform how we practice pathology tomorrow[J]. Arch Pathol Lab Med, 2015, 139: 929-935. DOI:10.5858/arpa.2014-0478-OA |
| [11] |
EHTESHAMI BEJNORDI B, VETA M, JOHANNES VAN DIEST P, VAN GINNEKEN B, KARSSEMEIJER N, LITJENS G, et al. Diagnostic assessment of deep learning algorithms for detection of lymph node metastases in women with breast cancer[J]. JAMA, 2017, 318: 2199-2210. DOI:10.1001/jama.2017.14585 |
| [12] |
VANDENBERGHE M E, SCOTT M L, SCORER P W, SÖDERBERG M, BALCERZAK D, BARKER C. Relevance of deep learning to facilitate the diagnosis of HER2 status in breast cancer[J/OL]. Sci Rep, 2017, 7: 45938. doi: 10.1038/srep45938.
|
| [13] |
YOSHIDA H, SHIMAZU T, KIYUNA T, MARUGAME A, YAMASHITA Y, COSATTO E, et al. Automated histological classification of whole-slide images of gastric biopsy specimens[J]. Gastric Cancer, 2018, 21: 249-257. DOI:10.1007/s10120-017-0731-8 |
| [14] |
YOSHIDA H, YAMASHITA Y, SHIMAZU T, COSATTO E, KIYUNA T, TANIGUCHI H, et al. Automated histological classification of whole slide images of colorectal biopsy specimens[J]. Oncotarget, 2017, 8: 90719-90729. |
| [15] |
YU G, YU W, JIN G, XU D, CHEN Y, XIA T, et al. PKM2 regulates neural invasion of and predicts poor prognosis for human hilar cholangiocarcinoma[J]. Mol Cancer, 2015, 14: 193. |
| [16] |
OSCANOA J, DOIMI F, DYER R, ARAUJO J, PINTO J, CASTANEDA B. Automated segmentation and classification of cell nuclei in immunohistochemical breast cancer images with estrogen receptor marker[J]. Conf Proc IEEE Eng Med Biol Soc, 2016, 2016: 2399-2402. |
| [17] |
THAKUR S S, LI H, CHAN A M Y, TUDOR R, BIGRAS G, MORRIS D, et al. The use of automated Ki67 analysis to predict Oncotype DX risk-of-recurrence categories in early-stage breast cancer[J/OL]. PLoS One, 2018, 13: e0188983. doi: 10.1371/journal.pone.0188983.
|
| [18] |
王革芳, 陈颖, 魏培莲, 赵行, 李超富, 高云姝, 等. 阿司匹林中断硫代乙酰胺诱发大鼠胆管上皮癌变[J]. 第二军医大学学报, 2018, 39: 285-290. WANG G F, CHEN Y, WEI P L, ZHAO X, LI C F, GAO Y S, et al. Aspirin interrupts bile duct carcinoma in rats induced by thioacetamide[J]. Acad J Sec Mil Med Univ, 2018, 39: 285-290. |
| [19] |
BAI G, ZHANG T, HOU Y, DING G, JIANG M, LUO G. From quality markers to data mining and intelligence assessment:a smart quality-evaluation strategy for traditional Chinese medicine based on quality markers[J]. Phytomedicine, 2018, 44: 109-116. DOI:10.1016/j.phymed.2018.01.017 |
 2018, Vol. 39
2018, Vol. 39


