2. 上海中医药大学附属龙华医院肿瘤七科, 上海 200032
2. Department of Oncology(Ⅶ), Longhua Hospital Affiliated to Shanghai University of Traditional Chinese Medicine, Shanghai 200032, China
根据世界卫生组织公布的数据,胃癌是最常见的5种癌症之一;在所有病例中胃癌占比为7%,大约有9%的癌症患者由于胃癌致死[1],胃癌位居恶性肿瘤发病率第5位、死亡率第3位[2]。由于人类肉眼不善于观测组织细胞中的变化,这可能导致医学专业人员对观测到的现象有不同理解。因此,在判断是否为癌症时,不同的医学专业人员会得到不一样的结论,而这种误判往往会使患者错过最佳治疗时机。
传统的医学图像分割算法包含区域的分割方法、边缘的分割方法、图论的分割方法和泛函的图像分割方法等。随着人工智能和计算机视觉等相关领域的快速发展,深度学习亦被运用于医学图像分割,并取得了显著效果。Garcia等[1]提出基于深度学习的胃癌免疫组织化学图像的淋巴细胞自动检测方法,该方法主要是基于卷积神经网络(convolutional neural network,CNN)模型的分类任务,可以从一张免疫组织化学染色图像中分辨出患者是否患有癌症,但不能精确给出癌症区域。Sharma等[3]也提出了一种基于CNN的分类方法用于识别胃癌,他们基于典型的AlexNet网络[4]提取图片特征信息,实现对整张数字病理图像胃癌的识别。Ficsor等[5]提出一种细胞计数方法,Zaitoun等[6]提出一种基于句法结构的方法。此外,Cosatto等[7]提出通过半监督学习的方法检测胃癌细胞,Sharma等[8]利用神经网络提取胃癌细胞核内的纹理信息以及支持向量机(support vector machine,SVM)算法实现了对胃癌的识别。然而,这些方法并不能很好地解决胃癌病理切片的识别问题,大多是基于整张病理切片图进行分类识别,虽然能识别出是否为胃癌患者,但不能准确定位到肿瘤区域,对于医师的后续治疗不能起到很大作用。为此,本研究采用基于深度学习的图像分割技术实现对病理切片的胃癌区域识别,帮助医学专业人员精准定位癌症区域,以期对患者的后续治疗产生积极意义。
1 深度学习网络模型 1.1 网络整体框架以U-Net网络[9]为基本架构,设计一种更深层次的CNN模型,将其命名为Deeper U-Net(DU-Net)。该网络包含14个卷积块(convolution block)、7个最大池化层(max-pooling layer)、7个上采样层(upsampling layer)和1个Sigmoid激活函数层。其中每个卷积块含有1个卷积层(convolution layer)、1个批规范层(batch normalization layer)和1个PReLU激活层[10]。网络整体架构如图 1所示。(1)输入:本模型中输入大小为512×512的3通道病理切片图。(2)卷积块:包括卷积层、批规范层、激活层。卷积层均采用3×3、步长(stride)为1的卷积核进行特征提取,每个卷积层之后都带有批规范层和PReLU激活层。批规范层能通过对卷积层的输入数据进行均值与方差上的修正,避免部分饱和非线性激活函数导致模型产生梯度弥散或爆炸。使用PReLU作为激活函数是因为相比ReLU激活函数其能加快模型的收敛速度,提高收敛效果。(3)最大池化层:步长为2×2,输入为卷积块生成的特征图,使用池化层能降低输出维度、避免特征冗余。(4)上采样层:上采样能增大特征图的分辨率,使图像还原到原始大小。每上采样1次,都要与特征提取中对应通道数尺度相融合,但特征提取部分要先整合成相同大小的特征图。(5)输出:最后1个卷积层后接Sigmoid激活函数层,输出与输入大小相同的概率热度图(probability map)。
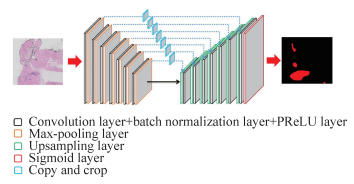
|
图 1 Deeper U-Net的网络结构图 Fig 1 Network structure of Deeper U-Net |
1.2 损失函数
在训练网络时,合适的损失函数能加快模型的收敛速度和效果。为此,本研究定义了一种损失函数Loss,其公式为:
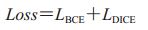
|
式中LBCE表示二值交叉熵代价函数,假设样本数量为m,xi、yi分别表示第i组样本及对应的标签,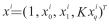

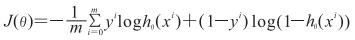
|
LDICE为相似系数损失,可用来综合衡量精度和召回率,通过以下公式计算。
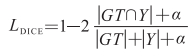
|
式中GT为对应图像的标签,Y为模型进行分割的结果,α=1,避免发生分母为0的情况。
2 训练方法本研究数据均为2 048×2 048像素大小的高分辨医学图像,不能直接作为神经网络的输入图片。因此需要将这些高分辨图片分割成若干低分辨图片,然后进行网络预测。
2.1 数据来源与处理本研究使用数据来源于2017中国大数据人工智能创新创业大赛“病理切片识别AI挑战赛”赛题,选取胃癌数字病理样本,常规H-E染色,放大倍数为20倍,图片大小为2 048×2 048像素,其中训练集1 400张、验证集400张、测试集100张。
由于标注的癌变区域中存在很多白色背景区域,直接对其训练会导致模型将白色背景区域看作癌变区域的一个特征。为此需要过滤大部分背景区域图片,以消除白色背景区域的干扰。对于深度学习来说,大规模数据集有利于提高模型的准确度。本文采用了随机旋转、平移、随机裁剪、随机颜色空间变换方法来进行数据的扩充。
2.2 区域重叠分割法医学图像一般为高分辨图片,通常需直接将高分辨率图片无重叠分割为小方块形图片,然后将这些低分辨率的方块形图片作为网络的输入图片。但本实验发现直接分割法会将一些较大区域的白色腺体分成许多小块,与白色背景相混淆,会直接影响最终实验结果。
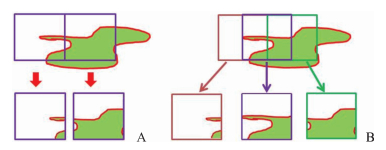
为此,本研究采用区域重叠分割法将大小为2 048×2 048像素的图片分割成大小为512×512像素的小图片,作为深度学习网络的输入图片。图 2表示两种不同的区域分割法,绿色区域为要识别的癌变区域,白色区域为背景区域。分割后的区域不仅包含癌变区域,同时也包含大量白色背景区域。使用无重叠分割法得到的图片进行训练,会使模型将白色背景区域占较大比例的图片误认为正常区域,从而增加预测的不正确性。区域重叠分割增加了图片视野(即观测域)的范围,对不同视野下的相同癌症区域进行重复预测,可有效避免上述情况。
|
图 2 无重叠分割和重叠分割法 Fig 2 No overlapping segmentation and overlapping segmentation methods The green and white areas representing the cancer needs to be recognized and the background, respectively. A: No overlapping segmentation method; B: Overlapping segmentation method |
首先,将宽度为W、高度为H的待预测图片(大小为SW, H)按照一定的步长s分割成w×h的图片(大小为Sw, h)。当s=0时,表示无重叠分割;当s>0时,表示将图片SW, H按照重叠区域为(w-s)h或(h-s)w进行分割。分割得到的图片数量计算公式如下:
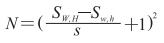
|
式中SW, H=2 048×2 048,Sw, h=512×512,s=256,因此每张2 048×2 048大小的图片可以被分割成49张512×512大小的图片。
2.3 图片分类器本研究设计了一个基于面积阈值的图片分类器,定义了真阳性样本、真阴性样本、假阳性样本、假阴性样本。粗标记中面积区域为癌变区域且被标记正确记为真阳性样本,面积区域为背景区域且被标记正确为真阴性样本,面积区域为背景区域但被标记为癌变区域记为假阳性样本,面积区域为癌变区域但被标记为背景区域记为假阴性样本。
在提取合适的图块时,要尽量避免假阴性和假阳性样本,为此本研究基于面积阈值对图像块进行分类提取。假设阳性区域(癌变区域)面积为UY、阴性区域(背景区域)面积为UN、阈值为β。可得公式如下:
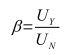
|
β≥0.7即认为该区域为阳性区域,否则为阴性区域。通过面积阈值的分类方法可初步将一张带标签的病理图片分类为癌症或正常细胞。
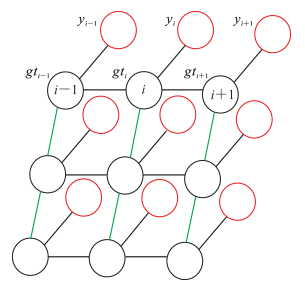
2.4 条件随机场(conditional random field,CRF)近年来,CRF被广泛应用于图像语义分割的后期处理,用来优化预测结果,降低错误率,本研究采用CRF方法对预测结果进行后期处理。假设第i像素所对应的标签为gti,通过模型预测得到预测标签为yi,如果将DU-Net模型所输出的概率热度图中的一个点作为场中的一个节点,并将像素之间的联系看作边,那么就能将这个模型看作CRF。
在传统方法中,CRF用于对带噪声的分割图像进行平滑处理,一般的短程CRF会清除弱分类器产生的错误预测。但本研究目标并不是清除,而是恢复这些细节。为此,本研究采用全连接CRF[11]处理模型的预测结果, 全连接CRF的结构示意图如图 3所示。全连接CRF可以描述像素与像素的联系,在去除噪声时发挥作用,缓解重复学习中的过拟合现象。此外,全连接CRF去除了语义分割后概率热度图的噪声,见图 4。
|
图 3 全连接条件随机场 Fig 3 Fully connected conditional random field |

|
图 4 全连接CRF对胃癌病理图像噪声区域的作用 Fig 4 Effect of full connected CRF on noise region of gastric cancer pathological images A, E: Cancer region is represented by green; B, F: Hot map; C, G: Without using fully connected CRF; D, H: Using fully connected CRF. A-D: Gastric cancer sample 1; E-H: Gastric cancer sample 2. CRF: Conditional random field. Original magnification: ×20 |
2.5 重复学习
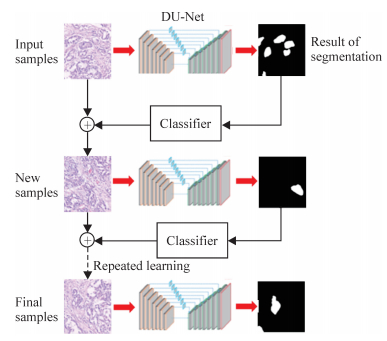
粗标记产生的一些错误样本问题可以通过DU-Net网络模型和区域重叠分割法得到基本解决,但要得到更好的结果以及进一步提升模型的泛化能力,就需要更精准的标注以提升模型的性能。为此,本研究提出了一种新的方法——重复学习法。
重复学习法与fine-tuning网络模型不同。其训练过程可分为3个阶段。第1阶段,使用设计好的DU-Net网络模型基于粗标记样本进行训练,得到初步训练模型,该模型基本可以预测出样本中的正常细胞和癌变细胞。第2阶段,使用区域重叠法分割2 048×2 048大小的图像及其掩码样本标签,然后运用初步训练模型对图像块按像素分类。第3阶段,利用图片分类器进行筛选,重新得到训练集样本。将新的样本放进模型进行微调(fine-tuning)得到新的参数模型,再重复上述步骤,最终得到满意的参数模型。图 5所示为重复学习过程,每次学习后都会产生新的样本,然后使用新样本重新学习得到最终的模型参数。计算模型分类的精确率(precision,Pr)、召回率(recall,Re)、平均交叉联合度量(intersection over union coefficient,IoU)、平均准确率(mean accuracy,MA),其计算公式如下:
|
图 5 重复学习流程 Fig 5 Process of repeated learning |

|
式中TP表示真阳性样本数,FP表示假阳性样本数,TN表示真阴性样本数,FN表示假阴性样本数。根据公式可见提高真阳性样本数才可以提升精确率,而减少假阴性样本数可以提高召回率。新的样本里已大大提高了真阳性样本数,降低了假阴性样本数,因此采用新的样本可以提升精确率和召回率。
3 实验结果和分析本实验基于Intel i7 8700K处理器,搭载了2块Nvidia GTX1080TI 11G的显卡,配备32 G内存,使用TensorFlow框架,依赖OpenCV库和PIL库进行数据处理。
3.1 实现细节训练模型的初始化采用Xavier Uniform[12],以Adam[13]作为网络优化算法。初始学习率为0.000 5,衰减量为0.8,即对验证集测试时的平均IoU连续4个epoch(1个epoch表示模型遍历整个训练集1次)没有发生增长时,取当前学习率乘以0.8作为新的学习率进行训练。微调模型时,学习率被设置为固定值0.000 01。每个批样本数的大小设置为16,即每次加载到模型的图片为16张。每张图片的大小为512×512,其中训练集图片18 000张、验证集6 000张、测试集6 000张。
3.2 实验结果使用图像分类器、区域重叠分割法和重复学习法进行胃癌图像的分割,得到的分割结果采用全连接CRF进行后续处理,并在测试集上进行测试,结果可见对于两个胃癌样本图像,不采用全连接CRF处理的模型分类结果的IoU分别为77.92%、78.17%,而采用全连接CRF处理后的IoU分别上升至84.58%、83.22%,证明全连接CRF能优化语义分割结果。
分别对基础DU-Net网络模型和经3次重复学习的DU-Net模型进行测试,结果如表 1所示,可见不采用重复学习法的DU-Net分类的平均召回率、MA和平均IoU均低于进行重复学习的DU-Net;且随着学习次数的增加,模型的总体性能上升。经过3次重复学习后,DU-Net网络模型的平均IoU达到88.4%,相比基础DU-Net提升了近3%;MA提升至91.5%。实验结果表明,本研究提出的重复学习法可以有效提升胃癌病理切片分割精度。
|
|
表 1 重复学习法在验证集中的表现结果 Tab 1 Performance of repeated learning in the validation set |
4 结论
本研究利用深度学习方法实现了对胃癌病理切片的识别,大幅度提升了模型性能,达到了满意效果。通过区域重叠分割法将数据集分割成若干小块图片,利用预先训练好的全卷积神经网络模型DU-Net对分割的小块图片进行初次分割,然后用图片分类器清除大量假阳性样本,重新合成新的样本。采用重复学习法使用这些新的样本重复训练,后将得到的结果应用全连接CRF进行后续处理,最终得到胃癌分割图片。该方法实现了精准的分割,提高了模型的泛化能力和鲁棒性,能满足临床医学的要求。
| [1] |
GARCIA E, HERMOZA R, CASTANON C B, CANO L, CASTILLO M, CASTANEDA C. Automatic lymphocyte detection on gastric cancer IHC images using deep learning[C/OL]//2017 IEEE 30th International Symposium on Computer-Based Medical Systems. IEEE, 2018:200-204. doi:10.1109/CMBS.2017.94.
|
| [2] |
左婷婷, 郑荣寿, 曾红梅, 张思维, 陈万青. 中国胃癌流行病学现状[J]. 中国肿瘤临床, 2017, 44: 52-58. DOI:10.3969/j.issn.1000-8179.2017.01.881 |
| [3] |
SHARMA H, ZERBE N, KLEMPERT I, HELLWICH O, HUFNAGL P. Deep convolutional neural networks for automatic classification of gastric carcinoma using whole slide images in digital histopathology[J]. Comput Med Imaging Graph, 2017, 61: 2-13. DOI:10.1016/j.compmedimag.2017.06.001 |
| [4] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[C]. International Conference on Neural Information Processing Systems, Curran Associates Inc, 2012:1097-1105.
|
| [5] |
FICSOR L, VARGA V, BERCZI L, MIHELLER P, TAGSCHERER A, WU M L, et al. Automated virtual microscopy of gastric biopsies[J]. Cytometry B Clin Cytom, 2006, 70: 423-431. |
| [6] |
ZAITOUN A M, AL MARDINI H, RECORD C O. Quantitative assessment of gastric atrophy using the syntactic structure analysis[J]. J Clin Pathol, 1998, 51: 895-900. DOI:10.1136/jcp.51.12.895 |
| [7] |
COSATTO E, LAQUERRE P, MALON C, GRAF H, SAITO A, KIYUNA T, et al. Automated gastric cancer diagnosis on H & E-stained sections; training a classifier on a large scale with multiple instance machine learning[J/OL]. SPIE Med Imaging, 2013, 8676:5. doi: 10.1117/12.2007047.
|
| [8] |
SHARMA H, ZERBE N, HEIM D, WIENERT S, BEHRENS H M, HELLWICH O, et al. A multi-resolution approach for combining visual information usingnuclei segmentation and classification in histopathological images[C]//Proceedings of the 10th International Conference on Computer Vision Theoryand Applications (VISAPP 2015), 2015, 3:37-46.
|
| [9] |
RONNEBERGER O, FISCHER P, BROX T. U-Net:convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer, 2015, 9351:234-241.
|
| [10] |
HE K, ZHANG X, REN S, SUN J. Delving deep into rectifiers:surpassing human-level performance on ImageNet classification[C]//IEEE International Conference on Computer Vision (ICCV). IEEE, 2015:1026-1034.
|
| [11] |
CHEN L C, PAPANDREOU G, KOKKINOS I, MURPHY K, YUILLE A L. DeepLab:semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs[J]. IEEE Trans Pattern Anal Mach Intell, 2018, 40: 834-848. DOI:10.1109/TPAMI.2017.2699184 |
| [12] |
GLOROT X, BENGIO Y. Understanding the difficulty of training deep feedforward neural networks[J]. J Mach Learn Res, 2010, 9: 249-256. |
| [13] |
KINGMA D P, BA J. Adam:a method for stochastic optimization[Z/OL]. arXiv:1412.6980, 2014. https://arxiv.org/pdf/1412.6980.pdf.
|
 2018, Vol. 39
2018, Vol. 39


