2. 华东师范大学空间信息与定位导航上海市高校工程技术研究中心, 上海 200241;
3. 上海中医药大学附属龙华医院肿瘤七科, 上海 200032
2. Engineering Center of SHMEC for Space Information and GNSS, East China Normal University, Shanghai 200241, China;
3. Department of Oncology(Ⅶ), Longhua Hospital Affiliated to Shanghai University of Traditional Chinese Medicine, Shanghai 200032, China
近年来乳腺癌的发病率和死亡率逐年上升,严重影响女性身心健康,其早期诊断和早期治疗尤为重要[1]。随着影像诊断技术的飞速发展,X线、计算机断层扫描(computed tomography,CT)、磁共振成像(magnetic resonance imaging,MRI)等的使用不仅可以提高乳腺癌的诊断准确率,也在一定程度上减轻了患者诊断时的痛苦[2-3]。计算机技术和图像处理技术的日趋成熟使计算机影像诊断技术在病理诊断中的应用前景广阔,也使其成为当下病理研究中的一个重要方向[4]。
乳腺癌的临床确诊主要是病理医师根据组织切片中的组织结构和细胞形态做出最终诊断,病理诊断是乳腺癌确诊的金标准。诊断结果的准确性依赖医师的经验,需要临床医师长期培训和学习,人力和物力成本都比较高。随着图像识别、深度学习[5-6]、神经网络[6]等关键技术的突破,“人工智能+医疗”的概念应运而生,其在自诊断、高效率、低成本等方面有着极大的竞争力。由于具有自学习、非线性映射、可塑性高等优点,神经网络在各个领域应用广泛,其在生物医学领域也取得了具有里程碑意义的成果,如皮肤癌诊断[7]、预测阿尔茨海默病[8]等。现有的生物组织病理诊断主要是基于灰度图像或彩色图像,其在计算机中表现出的信息有限,导致对生物组织的分析算法要求较高。
显微高光谱成像技术结合了显微成像技术和高光谱技术,成像图像不仅包含目标的空间信息,还包含了光谱信息[9],其为生物医学临床诊断提供了一种新的有效的辅助手段[10]。本研究结合组织病理学和计算机视觉,使用反向传播(back propagation,BP)神经网络对乳腺癌组织的显微高光谱图像进行识别分析,发现该技术能够区分组织切片中的肿瘤组织和非肿瘤组织,获得了较为准确的分类结果。
1 材料和方法 1.1 研究对象乳腺癌患者30例,均经病理证实,均为女性,年龄为42~82岁,平均(54.48±12.75)岁。切取每例患者的病灶组织,分割为5 mm×15 mm×15 mm大小,使用10%甲醛固定液进行组织固定。使用75%、85%、95%和100%的乙醇进行梯度脱水脱水处理,而后将组织块置于二甲苯中透明,用石蜡包埋机将组织标本包埋成病理石蜡块,用全自动石蜡切片机制成4 μm的病理组织切片,行苏木精-伊红染色(H-E染色)、脱水、透明,以获得乳腺癌组织样本。使用自主研发的显微高光谱成像系统(MHSI)[11]采集组织样本得高光谱图像90组,选取其中72组(占总数的80%)作为训练样本,另外18组作为测试样本,采用5次交叉验证,进行后续分析。
1.2 显微高光谱成像及预处理获取图像和光谱信息的高光谱成像技术将由物质成分决定的光谱与反映物质存在格局的影像完整结合起来,其中每张图像都包含了物质在不同波长下的信息[9, 11]。高光谱立方体数据如图 1所示,其中空间维M×N代表每张单波段图像的大小,光谱维K表示采集的波段总数,在每个波段下都有1幅图像。本研究采用的高光谱成像系统的光谱范围为373~809 nm,显微镜目镜倍数为10倍(固定),物镜倍数为10倍(可调节),图像数据空间分辨率为1 800×1 300,总波段数为60。
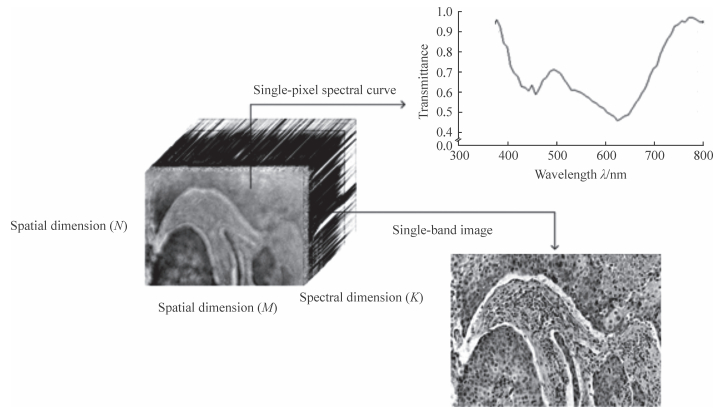
|
图 1 高光谱立方体数据 Fig 1 Hyperspectral data cube |
高光谱图像的质量受系统光源、光学器件、电流噪声等的影响。由于生物样本对光谱的吸收特性相对较小,未处理的图像不能反映生物组织的特异性,图像的预处理不可或缺。根据高光谱图像的性质,预处理的关键是在空间和光谱维度上分别进行校正[11]。平行单色入射光依次经过盖玻片、生物组织和载玻片,定义透光率T(m, n; λ)为:
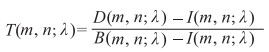
|
其中D(m, n; λ)为生物组织的第λ波段图像的n行m列的像素点值,B(m, n; λ)为空白区域的第λ波段图像的n行m列的像素点值,I(m, n; λ)为该像素点在关闭光源情况下的噪声。
1.3 BP神经网络BP神经网络是一种按照逆向传播算法训练的多层前馈神经网络[6, 12],由于具有很强的非线性映射能力和自适应能力,它在文字识别[13]、语音识别[14]、图像识别[15]等领域都有着广泛的应用。其结构如图 2所示,一般由输入层、隐含层和输出层组成,其训练过程就是不断更新各层神经元的参数。以3层BP神经网络为例,输入向量为X=(X1, …, Xn)T,输出向量为Y=(Y1, …, Yn)T,输入层与隐含层的连接权值为wih,隐含层与输出层的连接权值为who,隐含层各神经元的阈值为bh,输出层各神经元的阈值为bo,D=(D1, …, Dq)T为期望的输出向量,误差函数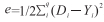

|
图 2 反向传播(BP)神经网络结构图 Fig 2 Structure of back propagation (BP) neural network |
(1)选取范围在(-1,1)内的随机数初始化误差函数e,权值wih、who。
(2)随机选取第k组输入样本Xi和期望输出Di,并重复以下步骤直到误差达到要求。
(3)计算隐含层各神经元的输入和输出:
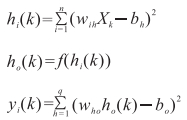
|
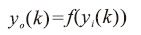
|
其中hi是隐含层的输入,ho是隐含层的输出,yi是输出层的输入,yo是输出层的输出,f(.)是激活函数,常用的激活函数有sigmoid、tanh函数等。
(4)计算误差函数e对输出层各神经元的偏导数Δwih(k),并利用偏导数更新隐含层的连接值:

|
其中η为学习效率,其值越大训练速度越快,但过大可能导致训练结果不收敛。
(5)计算n个样本(输入)、q个类别(输出)的全局误差:
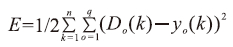
|
(6)从训练样本中继续选取1组输入和期望输出,不断更新各节点的权值和阈值,直至全局误差小于预先设定的一个值,也就是达到收敛状态。
BP神经网络的训练过程主要分为2个阶段,第1阶段是信号的前向传播,第2阶段是误差的反向传播,依次调节隐含层到输出层、输入层到隐含层的权重和偏置,使训练样本的输出和期望值在误差范围内接近,通过自学习完成样本的分类。
应用BP神经网络对显微高光谱乳腺组织图像中的不同区域进行分割,提取感兴趣区域。将算法分割结果与人工分割结果进行比较,包括每个区域正确的分类,被错误分割成其他类以及未被正确分割成对应组织。以T代表肿瘤、S代表基质、B代表空白,定义参数TT(正确标记为肿瘤的像素点)、TS(正确标记为基质的像素点)、TB(正确标记为空白的像素点,若样本不含空白区域,该值标记为0)、FT(错误标记为肿瘤的像素点)、FS(错误标记为基质的像素点)、FB(错误标记为空白的像素点,若样本不含空白区域,该值标记为0)。计算BP神经网络分割每组样本的准确率和精密度,计算公式:准确率(%)=(TT+TS+TB)/(TT+TS+TB+FT+FS+FB)×100%,精密度(%)=TT/(TT+FT)×100%。
2 结果 2.1 实验环境本实验的计算机操作系统为Microsoft Windows 10,Professional(SP2),处理器为Intel®CoreTMi5-4590 CPU 3.30 GHz,内存为4 G。软件平台为ENVI 5.1。首先对数据统一行预处理,然后使用ENVI 5.1中的神经网络工具进行分析。
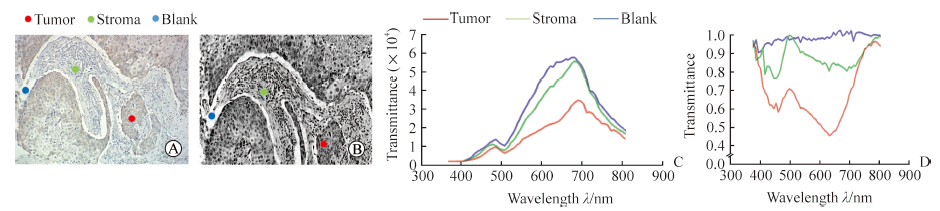
2.2 数据预处理根据显微高光谱图像预处理方法处理采集到的图像数据,实验结果如图 3所示。图 3A所示为系统采集的彩色图像,包含感兴趣的乳腺癌肿瘤组织。由于高光谱系统采集彩色图像和高光谱图像使用的是两个摄像头,所以在观察上会存在位置上的偏移,但彩色图像不是本研究主要对象,其作用是便于观察和对比。图 3B所示为高光谱灰度图像中第40波段对应的图像,该波段下噪声较小,图片质量较高,便于与分类结果进行对比。图中红、绿、蓝3种颜色标记分别表示肿瘤区域、基质区域和空白区域,这3类组织是本研究的主要成分(部分样本不存在空白区域)。通过观察图像预处理前后3类组织对应的光谱曲线可见,预处理前各类组织的光谱曲线趋势类似(图 3C),这是由于系统噪声掩盖了生物的本来特性;预处理削弱或消除了条带噪声等系统噪声,各类组织的光谱曲线开始呈现出各自的特性(图 3D),为下一步神经网络特征学习奠定了基础。
|
图 3 显微高光谱图像预处理 Fig 3 Preprocessing of micro-hyperspectral image A: RGB image (H-E staining); B: 40-band hyperspectral grayscale image (H-E staining); C: Spectral curve of different tissues before preprocessing; D: Spectral curve of different tissues after preprocessing. Original magnification: ×100 (A, B) |
2.3 实验结果分析
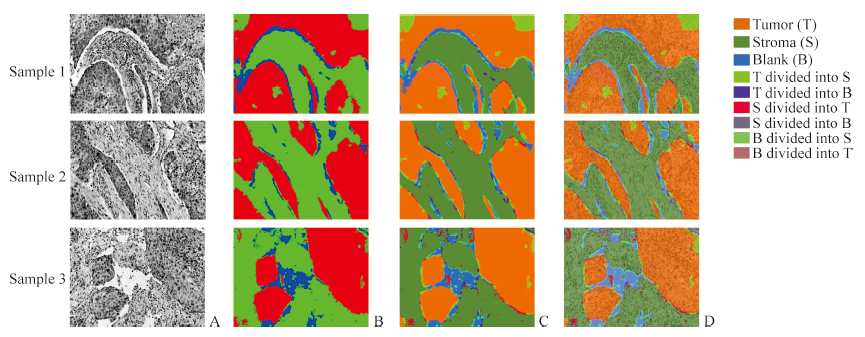
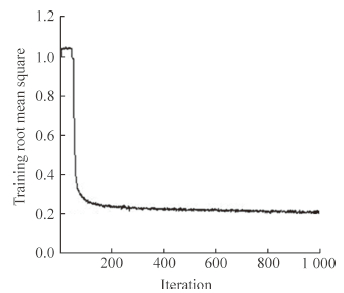
预处理后通过BP神经网络完成组织分割。设计3层BP神经网络结构,即只含有1个隐含层,激活函数采用logistic,设置最大迭代次数为1 000,学习速率为0.2,输出个数为3,即三分类,其结果如图 4所示。第40波段下的高光谱灰度图像和BP神经网络分割结果分别见图 4A和4B,其中红、绿、蓝色分别代表分割出来的肿瘤、基质和空白区域。与人工分割结果(图 4C)相比,BP神经网络算法分割样本的准确率为(85.13±4.73)%,精密度为(79.68±5.94)%。由于部分样本组织具有比较复杂的生物特性,以及不同医师的人工分割结果有所不同,都可能导致实验结果的误差,故将原始图像与分割结果合成到1张图像上,可见BP神经网络的分类效果比较理想(图 4D)。本研究中BP神经网络训练的均方误差(root mean square,RMS)不断趋近0.2,当迭代次数达到200次后,误差更新速率变缓,具有良好的收敛性。见图 5。
|
图 4 显微高光谱图像分割 Fig 4 Segmentation of micro-hyperspectral images A: 40-band hyperspectral grayscale image; B: Segmentation result by back propagation (BP) neural network; C: Manual segmentation result; D: Composition of original image and segmentation result by BP neural network. Original magnification: ×100 |
|
图 5 反向传播(BP)神经网络训练的误差变化 Fig 5 Training root mean square of back propagation (BP) neural network |
3 讨论
乳腺癌是女性发病率最高的肿瘤,随着医学技术的发展,乳腺癌患者的治疗效果和总体生存率均得到改善。近年来,随着计算机医学的发展,人工智能技术辅助诊断和治疗在乳腺癌中也取得了长足进步。目前人工智能技术在乳腺癌领域的研究包括乳腺癌风险评估[16]、计算机辅助诊断[17]、疾病复发和预后模型[18]、免疫分型[19]、治疗指导[20]和病理诊断[21]等。医学影像和病理图像是乳腺癌人工智能技术进展最快的领域,目前多项研究表明基于特定的算法和病理图片数字化处理,通过训练和算法优化,可开发出高精度、高效率的病理识别算法模型[21-22]。
本研究旨在探求一种由神经网络与生物医学结合的图像分割方法。首先,使用显微高光谱成像仪器采集乳腺癌样本图像;然后对显微高光谱图像进行预处理,消除系统中产生的一些干扰和噪声,突显生物组织的特异性;最后将预处理后的图像作为神经网络的输入,经过自学习后得到组织分类的图像。神经网络的参数设置对分类结果有直接影响,如学习速率过大虽然可以加速训练过程,但也可能导致不收敛,失去训练的意义,这要求从大量样本中找到适合训练的框架。本实验结果证明了利用BP神经网络进行乳腺癌样本中各区域的分割,避免了人工操作的耗时、耗力,且具有良好的准确率,可辅助病理医师进行精确的临床诊断。除临床病理诊断外,该技术结合图像提取还有助于对肿瘤内部的不同特征进行分类,以及对免疫组织化学染色进行提取和分类,同时对上述特征予以量化分析。
本研究初步证明了神经网络结合生物医学图像在临床中的应用价值,但本研究也存在不足,如纳入病例数较少、肿瘤类型单一、缺乏免疫学指标等。因此,需进一步展开后续研究:(1)扩大样本量,进一步证实和优化现有的算法;(2)实现样本多样化,纳入更多的病理类型,包括导管内癌、小叶原位癌、导管内乳头状癌、乳头湿疹样乳腺癌、乳头状癌、髓样癌(伴大量淋巴细胞浸润)、鳞状细胞癌、浸润性导管癌、浸润性小叶癌等;(3)加入乳腺癌分子分型,纳入雌激素受体、孕激素受体、人表皮生长因子受体2和Ki-67等指标。生物医学图像与人工智能技术的有效结合,必将促进医学发展,造福患者和医师。
| [1] |
VETA M, PLUIM J P, VAN DIEST P J, VIERGEVER M A. Breast cancer histopathology image analysis:a review[J]. IEEE Trans Biomed Eng, 2014, 61: 1400-1411. DOI:10.1109/TBME.2014.2303852 |
| [2] |
ATIYA A, 高燕, 王培军.乳腺癌MRI诊断进展[J]. 2018, 34:306-309. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=ZYXX201802049&dbname=CJFD&dbcode=CJFQ
|
| [3] |
孙艳霞, 丁敏. MRI、CT及X线在乳腺癌术前诊断及治疗指导中的应用[J]. 中国CT和MRI杂志, 2017, 15: 43-46. |
| [4] |
CHABI M L, BORGET I, ARDILES R, ABOUD G, BOUSSOUAR S, VILAR V, et al. Evaluation of the accuracy of a computer-aided diagnosis (CAD) system in breast ultrasound according to the radiologist's experience[J]. Acad Radiol, 2012, 3: 311-319. |
| [5] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521: 436-444. DOI:10.1038/nature14539 |
| [6] |
SCHMIDHUBER J. Deep learning in neural networks:an overview[J]. Neural Net, 2015, 61: 85-117. DOI:10.1016/j.neunet.2014.09.003 |
| [7] |
ESTEVA A, KUPREL B, NOVOA R A, KO J, SWETTER S M, BLAU H M, et al. Dermatologist-level classification of skin cancer with deep neural networks[J]. Nature, 2017, 542: 115-118. DOI:10.1038/nature21056 |
| [8] |
SUK H I, SHEN D. Deep learning-based feature representation for AD/MCI classification[J]. Med Image Comput Comput Assist Interv, 2013, 16(Pt 2): 583-590. |
| [9] |
李庆利, 肖功海, 薛永祺, 张敬法. 基于显微高光谱成像的人血细胞研究[J]. 光电工程, 2008, 35: 98-101. |
| [10] |
FREEMAN J, DOWNS F, MARCUCCI L, LEWIS E N, BLUME B, RISH J. Multispectral and hyperspectral imaging:applications for medical and surgical diagnostics[C]//19th Annual International Conference of the IEEE Engineering in Medicine & Biology Society. IEEE, 2002:700-701.
|
| [11] |
刘洪英, 李庆利, 顾彬, 王依婷, 薛永祺. 新型分子高光谱成像系统性能分析及数据预处理[J]. 光谱学与光谱分析, 2012, 32: 3161-3166. DOI:10.3964/j.issn.1000-0593(2012)11-3161-06 |
| [12] |
ZHENG Q, MILTHORPE B K, JONES A S. Direct neural network application for automated cell recognition[J]. Cytometry A, 2004, 57: 1-9. |
| [13] |
苗琦龙, 栾新. 基于遗传算法和BP网络的文字识别方法[J]. 计算机应用, 2005, 25: 330-332. DOI:10.3969/j.issn.2095-347X.2005.03.020 |
| [14] |
戚龙, 赵丹. 基于BP神经网络的非特定人语音识别算法[J]. 科学技术与工程, 2017, 17: 277-281. |
| [15] |
杨治明, 王晓蓉, 彭军, 陈应祖. BP人工神经网络在图像分割中的应用[J]. 计算机科学, 2007, 34: 234-236. |
| [16] |
SEPANDI M, TAGHDIR M, REZAIANZADEH A, RAHIMIKAZEROONI S. Assessing breast cancer risk with an artificial neural network[J]. Asian Pac J Cancer Prev, 2018, 19: 1017-1019. |
| [17] |
BURT J R, TOROSDAGLI N, KHOSRAVAN N, RAVIPRAKASH H, MORTAZI A, TISSAVIRASINGHAM F, et al. Deep learning beyond cats and dogs:recent advances in diagnosing breast cancer with deep neural networks[J/OL]. Br J Radiol, 2018:20170545. doi: 10.1259/bjr.20170545.
|
| [18] |
SAHA A, HAROWICZ M R, WANG W, MAZUROWSKI M A. A study of association of oncotype DX recurrence score with DCE-MRI characteristics using multivariate machine learning models[J]. J Cancer Res Clin Oncol, 2018, 144: 799-807. DOI:10.1007/s00432-018-2595-7 |
| [19] |
THAKUR S S, LI H, CHAN A M Y, TUDOR R, BIGRAS G, MORRIS D, et al. The use of automated Ki67 analysis to predict oncotype DX risk-of-recurrence categories in early-stage breast cancer[J/OL]. PLoS One, 2018, 13:e0188983. doi: 10.1371/journal.pone.0188983.
|
| [20] |
SOMASHEKHAR S P, SEPÚLVEDA M J, PUGLIELLI S, NORDEN A D, SHORTLIFFE E H, ROHIT KUMAR C, et al. Watson for Oncology and breast cancer treatment recommendations:agreement with an expert multidisciplinary tumor board[J]. Ann Oncol, 2018, 29: 418-423. DOI:10.1093/annonc/mdx781 |
| [21] |
BEJNORDI B E, VETA M, VAN DIEST P J, VAN GINNEKEN B, KARSSEMEIJER N, LITJENS G, et al. Diagnostic assessment of deep learning algorithms for detection of lymph node metastases in women with breast cancer[J]. JAMA, 2017, 318: 2199-2210. DOI:10.1001/jama.2017.14585 |
| [22] |
ROBERTSON S, AZIZPOUR H, SMITH K, HARTMAN J. Digital image analysis in breast pathology-from image processing techniques to artificial intelligence[J]. Transl Res, 2018, 194: 19-35. DOI:10.1016/j.trsl.2017.10.010 |
 2018, Vol. 39
2018, Vol. 39


