丙型肝炎病毒(hepatitis C virus, HCV)是一种全球流行的嗜肝RNA病毒, 全世界约有1.3亿人感染HCV。HCV的慢性感染率高达75%~80%, 而慢性HCV感染是导致肝硬化、肝癌的重要原因之一[1-2]。针对HCV蛋白酶和RNA聚合酶的新一代直接抗病毒药物(direct acting antivirals, DAAs)可彻底清除病毒, 达到治愈丙型肝炎的目的, 但其价格昂贵, 且存在病毒耐药突变[3-4]、在乙型肝炎病毒(hepatitis B virus, HBV)/HCV共感染者中增强HBV复制[5-7]等潜在问题。
从1989年HCV被发现以后, HCV疫苗的研发在美、欧发达国家备受关注, 有多种类型的疫苗进入了临床试验, 但迟迟没有HCV疫苗上市[8-10]。HCV感染与致病的任何阶段都涉及病毒与宿主因子的相互作用, 正是大量宿主因子的参与才使得HCV能够完成其完整的复制周期和引起肝组织的疾病。因此, 寻找、鉴定与HCV互相作用的宿主因子, 对于认识HCV感染与致病机制以及寻找可能用于HCV防治的干预靶点仍然具有重要意义。
前期我们通过转录组测序筛选出在HCV感染的人肝癌细胞系Huh7细胞中表达水平上调的乙酰胆碱酯酶(acetylcholinesterase, AchE; 未发表资料)。本研究拟进一步验证HCV感染对Huh7细胞AchE表达的影响, 并探讨AchE表达水平对HCV感染的影响, 为认识HCV建立持续感染的机制以及发现新的针对于宿主因子的抗HCV靶点提供依据, 同时为认识其他病毒, 尤其是黄病毒科其他成员的感染、免疫与致病机制提供新线索。
1 材料和方法 1.1 细胞、质粒与试剂人肝癌细胞系Huh7细胞的衍生细胞系Huh7.5.1细胞由中国科学院上海巴斯德研究所钟劲研究员馈赠, 用于制备细胞培养产生的HCV(HCVcc); 人肝癌细胞系Huh7细胞由本实验室保存, 用于研究HCVcc感染与AchE表达二者之间的相关性。J6/JFH-1嵌合全长HCV基因组转录质粒由洛克菲勒大学Charles Rice教授馈赠。
AchE抑制剂盐酸多奈哌齐(donepezil hydrochloride, Done)和盐酸伊托必利(itopride hydrochloride, Ito)购自美国Selleck公司; AchE活性检测试剂盒(Cat# MAK119)购自美国Sigma公司; 转染试剂Lipofectamine 3000、辣根过氧化物酶(horse radish peroxidase, HRP)标记的羊抗兔或羊抗小鼠IgG、Alexa Fluor®488标记的羊抗人IgG购自美国Invitrogen公司; CCK-8细胞增殖检测试剂盒购自日本Dojindo公司; 十二烷基硫酸钠-聚丙烯酰胺凝胶电泳配胶试剂盒购自上海碧云天生物技术有限公司; 小干扰RNA(small interfering RNA, siRNA)由广州锐博生物技术公司合成; SYBR Premix Ex Taq试剂购自日本TaKaRa公司; AchE抗体(ab31276)和HCV core抗体(ab2740)购自美国Abcam公司, 内参GAPDH兔单克隆抗体购自美国CST公司。
1.2 HCVcc制备将Huh7.5.1细胞消化并用无血清培养液Opti-MEM重悬, 调细胞密度为2×106/mL; 将10 μg J6/JFH-1 HCV RNA与10 mL Huh7.5.1细胞悬液混合, 置于电穿杯进行电穿转染, 条件为270 V、950 μF、100 Ω。随后加入2 mL新鲜DMEM培养液, 转移至6 cm培养皿中继续培养细胞。在48 h、72 h收集细胞培养上清(HCVcc), 以5 000×g离心5 min除去杂质和细胞碎片, 再将上清分装, 于-80 ℃冻存备用。
1.3 AchE活性检测将Huh7细胞接种到6孔板, HCVcc感染Huh7细胞12、36、60 h后分别用5 mL离心管收集细胞进行超声破碎(2×106 Hz, 超声3 s, 间隔10 s, 重复30次), 然后以12 000×g离心5 min, 取上清利用BCA法进行蛋白定量, 最终蛋白浓度为100 μg/mL, 然后按照AchE活性检测试剂盒实验方法对其进行检测。根据公式计算AchE活性:AchE活性(U/L)=(D412B-D412A)/(D412校-D412M)×200N, 其中D412B是上清孵育10 min后412 nm波长处光密度值, D412A是上清孵育2 min后412 nm波长处光密度值, D412校是校准液10 min后412 nm波长处光密度值, D412M是10 min后对照组上清412 nm波长处光密度值, N是上清稀释倍数。
1.4 AchE抑制剂处理细胞用二甲基亚砜(dimethyl sulfoxide, DMSO)溶解2种AchE抑制剂Done和Ito, 制备浓度分别为10 mmol/L和50 mmol/L的储存液。首先检测AchE抑制剂Ito和Done的细胞毒性。将Huh7细胞接种到96孔板中, 当细胞融合度达到70%时将抑制剂分别稀释成60、125、250、500 μmol/L 4个浓度梯度处理细胞, 60 h后用CCK-8检测细胞增殖水平。
接下来将Huh7细胞接种到24孔板中, 当细胞融合度达到70%时, 用DMEM完全培养液将储存液稀释, 加入细胞培养上清中, 终浓度分别为60、125、250 μmol/L, 对照组加入等体积的DMEM培养液。60 h后用AchE活性检测试剂盒检测AchE活性。
1.5 siRNA转染将siRNA干粉溶于RNase-free水中, 浓度为20 μmol/L, 分装, -80 ℃保存。将Huh7细胞以较低密度接种于24孔板, 细胞生长融合度达到40%~50%时, 用Lipofectamine 3000转染siRNA(包括针对AchE基因的siRNA和对照siRNA), 每孔siRNA用量为40 pmol, 具体步骤参照试剂使用说明。针对AchE基因的siRNA共3条, siAchE1序列为5′-GCT CGT ATT TTC TGG TTT A-3′, siAchE2序列为5′-CGA CAT CAG TGA CGC TGT T-3′, siAchE3序列为5′-GCT ACG AGA TCG AGT TCA T-3′。
1.6 实时荧光定量PCR(real-time quantitative PCR, qPCR)检测AchE和HCV mRNA用RNAiso Plus试剂提取细胞总RNA, RNA沉淀溶解于DEPC处理水, 多功能酶标仪测定RNA浓度和纯度。然后用PrimeScriptTM RT Master Mix试剂反转录获得cDNA, 随后用SYBR Primer Ex Taq试剂进行qPCR以检测AchE和HCV mRNA水平。qPCR操作参考试剂盒说明书。引物序列如下:AchE上游5′-CTT CGT GCC TGT GGT AGA T-3′, 下游5′-GCT GAT GAG AGA CTC GTT GTC-3′; JFH-1型HCV上游5′-TCT GCG GAA CCG GTG AGT A-3′, 下游5′-TCA GGC AGT ACC ACA AGG C-3′; 内参照基因GAPDH上游5′-GAA GGT GAA GGT CGG AGT C-3′, 下游5′-GAA GAT GGT GAT GGG ATT TC-3′。
1.7 蛋白质印迹法与免疫荧光法检测AchE及HCV蛋白的表达用HCVcc感染Huh7细胞, 以加入与HCVcc等体积的Huh7.5.1细胞培养上清的Huh7细胞作为对照组。分别在培养12 h、36 h、60 h后用RIPA蛋白裂解液(强)裂解细胞, 制备总蛋白样品。利用BCA法进行蛋白定量, 最终每孔用20 μg蛋白量进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳, 随后将凝胶中的蛋白样品电转至硝酸纤维素膜, 以GAPDH作为内参, 用蛋白质印迹法检测细胞裂解液中的AchE和HCV core蛋白表达量。
将Huh7细胞接种到96孔板, 过夜培养, 以HCVcc感染, 同时加入浓度为60、125、250 μmol/L的AchE抑制剂, 混合均匀继续培养60 h后, 用免疫荧光法检测细胞中的HCV蛋白, 一抗为HCV抗体阳性的丙型肝炎患者血清[由海军军医大学(第二军医大学)长海医院实验诊断科提供], 二抗为Alexa Fluor®488标记的羊抗人IgG, 用DAPI复染细胞核。用Bioteck细胞荧光成像系统计数荧光阳性细胞。
1.8 统计学处理应用GraphPad Prism 5.0软件进行数据处理和统计学分析。呈正态分布的计量资料以x±s表示, 两组间比较采用两独立样本t检验, 多组间比较采用单因素方差分析(one-way ANOVA)。检验水准(α)为0.05。
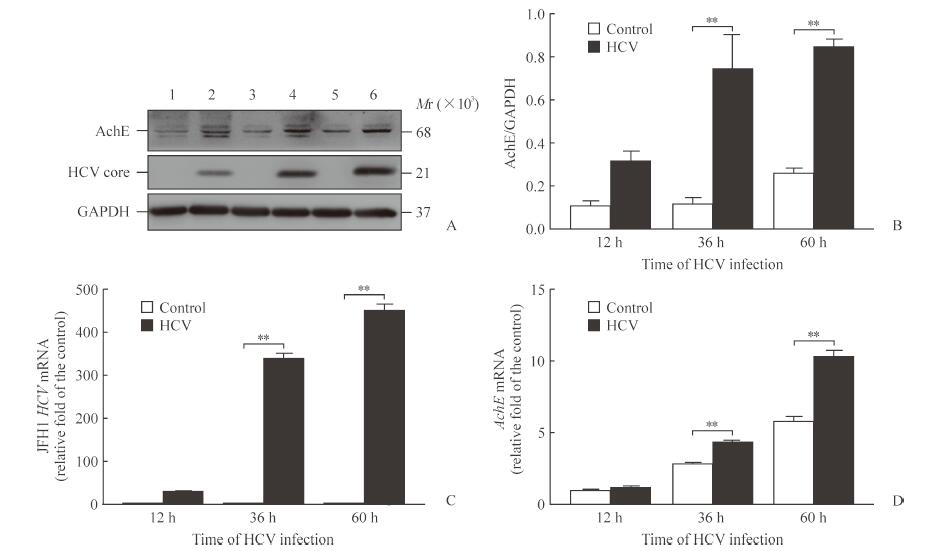
2 结果 2.1 HCV感染上调Huh7细胞AchE的表达图 1A、1B结果显示, 在HCVcc感染Huh7细胞36 h和60 h后, 与对照组相比AchE蛋白表达均上调(P均<0.01)。用qPCR检测细胞内AchE mRNA的变化, 图 1C、1D结果显示HCVcc感染Huh7细胞36 h和60 h后, 与对照组相比细胞内AchE mRNA含量均增加(P均<0.01), 与前期转录组深度测序结果相符合。表明HCV感染可以明显上调Huh7细胞内AchE的表达。
|
图 1 HCV感染Huh7细胞上调AchE表达 Fig 1 HCV infection up-regulating AchE expression in Huh7 cells A:Western blotting of AchE expression levels after HCV infection.1, 2:12 h after HCV infection; 3, 4:36 h after HCV infection; 5, 6:60 h after HCV infection.1, 3, 5:Control group; 2, 4, 6:HCV group.B:Grayscale analysis of AchE expression level relative to GAPDH.C, D:qPCR of JFH1 HCV and AchE mRNA relative fold change after HCV infection, respectively.HCV:Hepatitis C virus; AchE:Acetylcholinesterase; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase; qPCR:Real-time quantitative PCR; JFH1:Japanese fulminant hepatitis 1.**P < 0.01.n=3, x±s |
2.2 HCV感染上调Huh7细胞内AchE活性
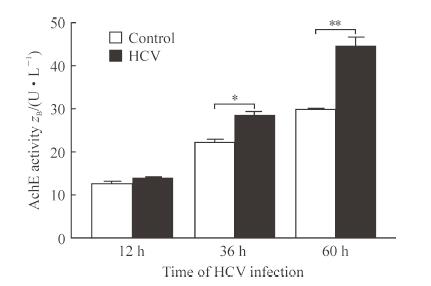
图 2结果显示, AchE活性随着HCVcc感染Huh7细胞的时间延长而增强, 在HCVcc感染细胞36 h和60 h时AchE活性与对照组相比差异均有统计学意义(P均<0.01)。
|
图 2 HCV感染上调Huh7细胞内AchE活性 Fig 2 HCV infection up-regulating AchE activity in Huh7 cells HCV:Hepatitis C virus; AchE:Acetylcholinesterase.*P < 0.05, **P < 0.01.n=3, x±s |
2.3 AchE抑制剂可抑制HCV感染
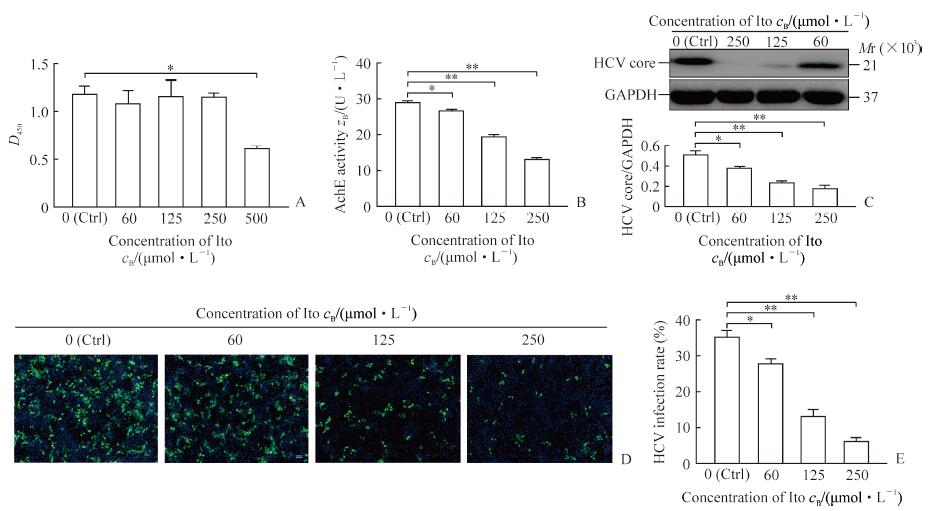
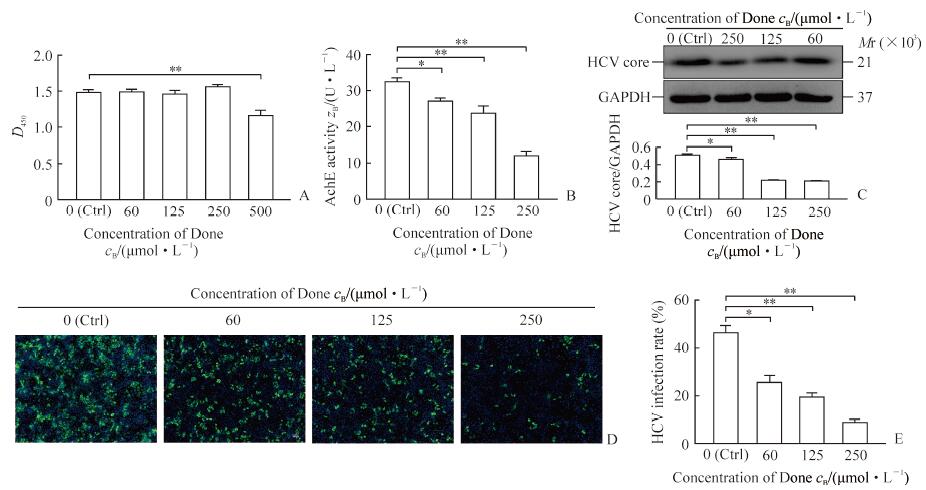
图 3A结果显示, 与DMSO处理的细胞比较, 当抑制剂Ito浓度达到500 μmol/L时对细胞产生毒性作用(P<0.05)。图 4A结果显示, 当抑制剂Done浓度达到500 μmol/L时对细胞产生毒性作用(P<0.01)。2种抑制剂以3个较低浓度处理细胞均未见明显细胞毒性。图 3B、4B结果都显示, 2种抑制剂的浓度越高对AchE活性的抑制作用越明显, 其中当抑制剂浓度达到250 μmol/L时对AchE活性抑制效果最明显(P<0.01)。图 3C、4C结果显示, 2种抑制剂均剂量依赖性降低HCV core蛋白的表达, 与对照组相比, 当抑制剂浓度为60 μmol/L时HCV core蛋白的表达下调比较明显(P<0.05), 而当抑制剂浓度达到125 μmol/L和250 μmol/L时, HCV core蛋白的表达下调非常明显(P<0.01)。图 3D、3E和图 4D、4E中的免疫荧光检测结果类似, 当抑制剂浓度为60 μmol/L时HCV对Huh7细胞阳性感染率的降低比较明显(P<0.05或P<0.01), 而当抑制剂浓度达到125 μmol/L和250 μmol/L时HCV对Huh7细胞阳性感染率的降低非常明显(P<0.01)。
|
图 3 AchE抑制剂Ito抑制AchE活性并降低HCV感染 Fig 3 AchE inhibitor Ito inhibiting AchE activity and reducing HCV infection A:CCK-8 detection of Ito cytotoxicity on Huh7 cells; B:The effect of Ito on AchE activity; C:Western blotting of Ito inhibiting HCV core protein expression and relative grayscale analysis; D:Immunofluorescence of Ito inhibition of HCV infection; E:HCV positive infection rate according to the immunofluorescence.Ctrl:Dimethyl sulfoxide as control; AchE:Acetylcholinesterase; Ito:Itopride hydrochloride; HCV:Hepatitis C virus; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase.Original magnification:×100 (D).*P < 0.05, **P < 0.01.n=3, x±s |
|
图 4 AchE抑制剂Done抑制AchE活性并降低HCV感染 Fig 4 AchE inhibitor Done inhibiting AchE activity and reducing HCV infection A:CCK-8 detection of Done cytotoxicity on Huh7 cell; B:The effect of Done on AchE activity; C:Western blotting of Done inhibiting HCV core protein expression and relative grayscale analysis; D:Immunofluorescence of Done inhibition on HCV infection; E:HCV positive infection rate according to the immunofluorescence.Ctrl:Dimethyl sulfoxide as control; AchE:Acetylcholinesterase; Done:Donepezil hydrochloride; HCV:Hepatitis C virus; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase.Original magnification:×100 (D).*P < 0.05, **P < 0.01.n=3, x±s |
2.4 下调AchE表达能降低HCV对Huh7细胞的感染
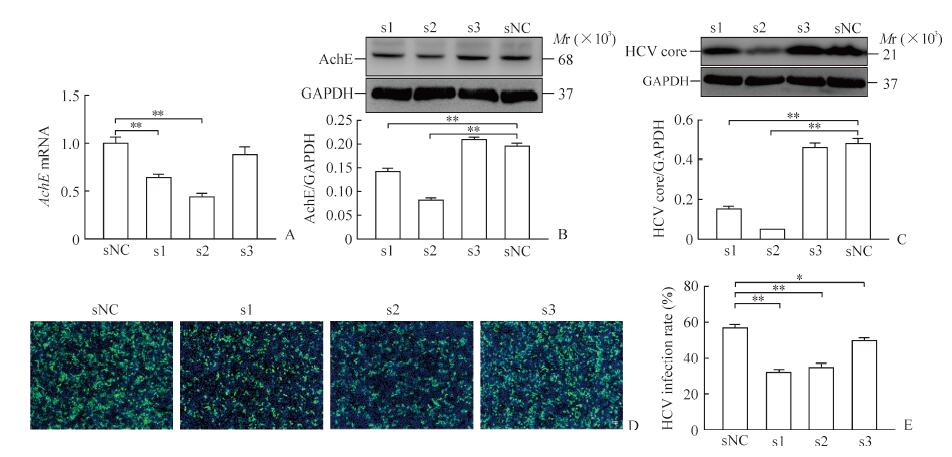
图 5A及5B分别显示, AchE siRNA1和AchE siRNA2均可下调Huh7细胞中AchE的mRNA以及蛋白表达水平(P均<0.01), 而AchE siRNA3不能下调Huh7细胞中AchE的表达水平。图 5C的蛋白质印迹结果显示, 下调AchE的表达后Huh7细胞中HCV core蛋白的表达水平降低, 与对照组比较差异有统计学意义(P<0.01)。图 5D、5E免疫荧光检测结果也显示, 当Huh7细胞中AchE表达被下调后HCV的阳性感染率降低, 设计的3条AchE siRNA所致结果与对照组相比差异均有统计学意义(P<0.05或P<0.01)。
|
图 5 下调AchE的表达降低HCV对Huh7细胞的感染 Fig 5 Down-regulation of AchE expression reducing HCV infection of Huh7 cells A:qPCR of AchE knockdown effect; B:Western blotting of AchE knockdown effect; C:Western blotting of AchE knockdown affecting HCV core protein expression; D:Immunofluorescence of AchE knockdown affecting HCV infection; E:The HCV positive infection rate according to the immunofluorescence.sNC:Negative control of the siRNA; s1:The first siRNA of AchE; s2:The second siRNA of AchE; s3:The third siRNA of AchE; AchE:Acetylcholinesterase; HCV:Hepatitis C virus; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase; qPCR:Real-time quantitative PCR; siRNA:Small interfering RNA.Original magnification:×100 (D).*P < 0.05, **P < 0.01.n=3, x±s |
3 讨论
前期我们通过转录组测序发现HCVcc感染能明显上调Huh7细胞内AchE的表达, HCV感染12 h、36 h、60 h后AchE mRNA相比未感染HCVcc的细胞分别升高1.44、4.93、13.15倍(未发表资料)。越来越多的研究发现, AchE不仅存在于神经细胞中, 在成纤维细胞、红细胞、血管内皮细胞、肝细胞等细胞中也广泛存在[11-13], 而且参与细胞增殖、凋亡以及分化, 并与细胞氧化应激和炎症反应有关[14-17]。有报道在240例原发性肝癌患者中, 69.2%患者的肝癌细胞中AchE表达下降, AchE低表达可以促进肝癌细胞体外增殖以及在裸鼠体内的致瘤性, 同时还可降低肝癌细胞的化学治疗敏感性[18]。然而目前尚未见AchE影响HCV感染的相关报道, 本研究发现当AchE活性被抑制及其蛋白表达被下调后, HCV对肝癌细胞的感染也随之下降, 表明AchE也可以有效地影响肝癌细胞HCV的感染。
AchE能够将神经递质乙酰胆碱降解为胆碱, 胆碱对脂肪有亲和力, 可促进脂肪以磷脂形式由肝脏通过血液输送出去或改善脂肪酸本身在肝脏中的利用, 并防止脂肪在肝脏内的异常积聚。胆碱本身对肝细胞也具有保护作用, 临床上常用于辅助治疗肝硬化、肝炎, 效果良好。肝细胞通过胞苷二磷酸胆碱代谢途径(CDP-choline pathway)利用胆碱参与各种磷脂的合成以及促进脂肪酸的合理分配, 从而调控肝脏脂肪代谢。研究发现, 肝癌细胞对放射性标记胆碱的摄入量高于其周围的细胞, 故推荐将甲基化的胆碱作为一种代谢标记结合正电子发射型计算机断层显像(positron emission computed tomography, PET)用于肝癌细胞中脂质合成的成像研究[19]。
HCV的细胞侵入、复制以及病毒体的组装与释放的每个过程均与肝细胞的脂质代谢密切相关[20-22]。在细胞侵入阶段, HCV需要与定位于细胞膜脂筏中的受体分子结合; 在复制阶段, 由HCV非结构蛋白完成的基因组复制发生于细胞内的膜网结构; HCV核衣壳的组装起始于细胞内的脂滴结构; HCV的释放则依赖于细胞的低密度脂蛋白分泌途径。因此, 我们推测AchE可能通过参与肝细胞的脂质代谢影响HCV感染。另外HCV感染肝细胞长时间不引起细胞病变, 从而造成无症状感染, 这是否与HCV上调AchE促进胆碱的产生有关?下一步我们将针对胆碱在脂代谢中的作用以及其对肝细胞的保护作用两个方面探讨AchE影响HCV感染的机制, 明确该机制可能为研发新的丙型肝炎治疗药物提供思路。
| [1] |
MESSINA J P, HUMPHREYS I, FLAXMAN A, BROWN A, COOKE G S, PYBUS O G, et al. Global distribution and prevalence of hepatitis C virus genotypes[J]. Hepatology, 2015, 61: 77-87. DOI:10.1002/hep.27259 |
| [2] |
GOLDBERG D, DITAH I C, SAEIAN K, LALEHZARI M, ARONSOHN A, GOROSPE E C, et al. Changes in the prevalence of hepatitis C virus infection, nonalcoholic steatohepatitis, and alcoholic liver disease among patients with cirrhosis or liver failure on the waitlist for liver transplantation[J]. Gastroenterology, 2017, 152: 1090-1099. DOI:10.1053/j.gastro.2017.01.003 |
| [3] |
SARRAZIN C. The importance of resistance to direct antiviral drugs in HCV infection in clinical practice[J]. J Hepatol, 2016, 64: 486-504. DOI:10.1016/j.jhep.2015.09.011 |
| [4] |
PAWLOTSKY J M. Hepatitis C virus resistance to direct-acting antiviral drugs in interferon-free regimens[J]. Gastroenterology, 2016, 151: 70-86. DOI:10.1053/j.gastro.2016.04.003 |
| [5] |
KAWAGISHI N, SUDA G, ONOZAWA M, KIMURA M, MAEHARA O, OHARA M, et al. Comparing the risk of hepatitis B virus reactivation between direct-acting antiviral therapies and interferon-based therapies for hepatitis C[J]. J Viral Hepat, 2017, 24: 1098-1106. DOI:10.1111/jvh.2017.24.issue-12 |
| [6] |
CHEN G, WANG C, CHEN J, JI D, WANG Y, WU V, et al. Hepatitis B reactivation in hepatitis B and C coinfected patients treated with antiviral agents:a systematic review and meta-analysis[J]. Hepatology, 2017, 66: 13-26. DOI:10.1002/hep.v66.1 |
| [7] |
WANG C, JI D, CHEN J, SHAO Q, LI B, LIU J, et al. Hepatitis due to reactivation of hepatitis B virus in endemic areas among patients with hepatitis C treated with direct-acting antiviral agents[J]. Clin Gastroenterol Hepatol, 2017, 15: 132-136. DOI:10.1016/j.cgh.2016.06.023 |
| [8] |
PIERCE B G, BOUCHER E N, PIEPENBRINK K H, EJEMEL M, RAPP C A, THOMAS W D Jr, et al.Structure-based design of hepatitis C virus vaccines that elicit neutralizing antibody responses to a conserved epitope[J/OL].J Virol, 2017, 91: e01032-17.doi: 10.1128/JVI.01032-17.
|
| [9] |
OYARZUN P, KOBE B. Recombinant and epitope-based vaccines on the road to the market and implications for vaccine design and production[J]. Hum Vaccin Immunother, 2016, 12: 763-767. DOI:10.1080/21645515.2015.1094595 |
| [10] |
KAZI A, CHUAH C, LEOW C Y, LIM B H, MAJEED A B A, LEOW C H. Current progress of immunoinformatics approach harnessed for cellular-and antibody-dependent vaccine design[J]. Pathog Glob Health, 2018, 112: 123-131. DOI:10.1080/20477724.2018.1446773 |
| [11] |
MONTENEGRO M F, RUIZ-ESPEJO F, CAMPOY F J, MUÑOZ-DELGADO E, DE LA CADENA M P, RODRÍGUEZ-BERROCAL F J, et al. Cholinesterases are down-expressed in human colorectal carcinoma[J]. Cell Mol Life Sci, 2006, 63: 2175-2182. DOI:10.1007/s00018-006-6231-3 |
| [12] |
GUPTA S, BELLE V S, KUMBARAKERI RAJASHEKHAR R, JOGI S, PRABHU R K. Correlation of red blood cell acetylcholinesterase enzyme activity with various rbc indices[J]. Indian J Clin Biochem, 2018, 33: 445-449. DOI:10.1007/s12291-017-0691-0 |
| [13] |
WEIST R, FLÖRKEMEIER T, ROGER Y, FRANKE A, SCHWANKE K, ZWEIGERDT R, et al. Differential expression of cholinergic system components in human induced pluripotent stem cells, bone marrow-derived multipotent stromal cells, and induced pluripotent stem cell-derived multipotent stromal cells[J]. Stem Cells Dev, 2018, 27: 166-183. DOI:10.1089/scd.2017.0162 |
| [14] |
YE W, GONG X, XIE J, WU J, ZHANG X, OUYANG Q, et al. AChE deficiency or inhibition decreases apoptosis and p53 expression and protects renal function after ischemia/reperfusion[J]. Apoptosis, 2010, 15: 474-487. DOI:10.1007/s10495-009-0438-3 |
| [15] |
ZHANG X J, YANG L, ZHAO Q, CAEN J P, HE H Y, JIN Q H, et al. Induction of acetylcholinesterase expression during apoptosis in various cell types[J]. Cell Death Differ, 2002, 9: 790-800. DOI:10.1038/sj.cdd.4401034 |
| [16] |
LAYER P G. Cholinesterases during development of the avian nervous system[J]. Cell Mol Neurobiol, 1991, 11: 7-33. DOI:10.1007/BF00712798 |
| [17] |
BEN AZIZ-ALOYA R, STERNFELD M, SOREQ H. Promoter elements and alternative splicing in the human ACHE gene[J]. Prog Brain Res, 1993, 98: 147-153. DOI:10.1016/S0079-6123(08)62392-4 |
| [18] |
ZHAO Y, WANG X, WANG T, HU X, HUI X, YAN M, et al. Acetylcholinesterase, a key prognostic predictor for hepatocellular carcinoma, suppresses cell growth and induces chemosensitization[J]. Hepatology, 2011, 53: 493-503. DOI:10.1002/hep.24079 |
| [19] |
KUANG Y, SALEM N, TIAN H, KOLTHAMMER J A, CORN D J, WU C, et al. Imaging lipid synthesis in hepatocellular carcinoma with [methyl-11C]choline:correlation with in vivo metabolic studies[J]. J Nucl Med, 2011, 52: 98-106. DOI:10.2967/jnumed.110.080366 |
| [20] |
STOECK I K, LEE J, TABATA K, ROMERO-BREY I, PAUL D, SCHULT P, et al.Hepatitis C virus replication depends on endosomal cholesterol homeostasis[J/OL].J Virol, 2018, 92: e01196-17.doi: 10.1128/JVI.01196-17.
|
| [21] |
KIM J Y, WANG L, LEE J, OU J J.Hepatitis C virus induces the localization of lipid rafts to autophagosomes for its RNA replication[J/OL].J Virol, 2017, 91: e00541-17.doi: 10.1128/JVI.00541-17.
|
| [22] |
KOLETZKO L, MAHLI A, HELLERBRAND C. Development of an in vitro model to study hepatitis C virus effects on hepatocellular lipotoxicity and lipid metabolism[J]. Pathol Res Pract, 2018, 214: 1700-1706. DOI:10.1016/j.prp.2018.08.013 |
 2018, Vol. 39
2018, Vol. 39


