2. 海军军医大学(第二军医大学)长海医院临床实验中心, 上海 200433
2. Clinical Research Center, Changhai Hospital, Navy Medical University(Second Military Medical University), Shanghai 200433, China
肝细胞癌(hepatocellular carcinoma, HCC)是肝恶性肿瘤中最常见的病理类型, 其进展快, 恶性程度较高, 预后较差, 死亡率较高[1]。肿瘤转移是人类恶性肿瘤疾病中致死性因素之一[2], 探讨肿瘤转移的机制对于开发相关治疗策略、降低转移相关死亡率具有重要意义。长链非编码RNA(long non-coding RNA, lncRNA)是一类转录本长度超过200核苷酸单位(nucleotide)的功能性RNA分子, 参与包括HCC在内的多种癌症的发生和发展过程, 且与许多肿瘤的发展与转移密切相关[3-5]。
多能因子是保持细胞多潜能性和自我更新的核心基因, 具有抑制细胞分化的重要作用, 参与调控胚胎干细胞自我更新和多能性维持。核蛋白oct4、nanog和sox2是多能因子的3个主要成员[6], 近年研究发现, 这3个多能因子在生殖细胞肿瘤和多种实体肿瘤中异常高表达, 参与正常细胞恶性转化, 调控肿瘤细胞的干性, 促进其增殖、迁移和侵袭等[7-9]。它们的多重作用可能是推动肿瘤发展和转移的重要因素。本研究旨在寻找与多能因子相关的lncRNA, 探讨其在HCC发展和转移中的作用, 为进一步探究HCC转移的机制提供新的思路。
1 材料和方法 1.1 主要材料临床标本为2012年1月至8月海军军医大学(第二军医大学)东方肝胆外科医院肝外六科行手术切除并经病理确诊的HCC原发灶组织及转移灶组织标本各3例, 标本获取后立即浸入RNA保存液并保存于-80 ℃冰箱中。人肝癌细胞系Huh7、Hep3B的原始细胞株均购自中国科学院上海生命科学研究院细胞库。培养细胞的培养液、血清、胰酶、青霉素、链霉素、胰岛素样生长因子(insulin-like growth factor, IGF)、成纤维细胞生长因子(fibroblast growth factor, FGF)、表皮生长因子(epidermal growth factor, EGF)均购自美国Gibco公司; 引物为生工生物工程(上海)股份有限公司合成; 转染试剂脂质体3000及TRIzol试剂均购自美国Invitrogen公司; cDNA反转录试剂盒及实时荧光定量PCR试剂盒购自美国Thermo Fisher Scientific公司; SYBR Taq酶购自日本TaKaRa公司; lncRNA-8439原位杂交探针、缓冲液等购自广州市锐博生物科技有限公司; 蛋白质印迹检测中使用的nanog抗体购自美国Cell Signaling公司, 二抗购自美国Santa Cruz Biotechnology公司。
1.2 差异表达lncRNA的筛选 1.2.1 LncRNA测序采集HCC患者原发灶与转移灶组织标本各3例, 根据TRIzol试剂操作说明抽提总RNA, 检测RNA的纯度及完整性, 样品合格后分装保存于-80 ℃冰箱。采用AMPure XP beads核酸纯化试剂盒(美国Beekman Coulte Beverly公司)筛选cDNA。之后进行PCR扩增并再次使用AMPure XP beads核酸纯化试剂盒纯化PCR产物。获得cDNA文库并使用Agilent2100生物分析仪(美国Agilent Technologies公司)对cDNA文库进行质检。LncRNA测序由上海康成生物有限公司完成。采用Illumina HiSeq测序平台, 对HCC患者原发灶和转移灶组织进行双端测序。采用STAR软件、HTSeq软件和edgeR软件进行表达差异分析与筛选, 筛选的标准为变化倍数>2或<0.5且P<0.05, 并利用Tree Viewer软件进行集簇分析。
1.2.2 生物信息学分析使用starBase V2.0工具筛选与多能因子sox2、oct4、nanog相关的lncRNA, 使用lncRNAdb工具预测lncRNA-8439与nanog的相互作用位点。
1.3 细胞培养 1.3.1 细胞系培养人肝癌细胞系Huh7和Hep3B采用含10%胎牛血清、100 U/mL青霉素和0.1 g/L链霉素的DMEM培养液, 置于37 ℃、5% CO2的培养箱中培养, 每天观察细胞生长情况, 隔天换液。
1.3.2 悬浮球培养将1 000个单个Huh7和Hep3B细胞分别接种于包被聚2-羟乙基甲基丙烯酸酯[poly(2-hydroxyethyl methacrylate), polyHEMA]涂层的6孔板。悬浮球采用20 ng/mL IGF、1.0 ng/mL FGF以及20 ng/mL EGF的DMEM/F12培养体系, 置于37 ℃、5% CO2的培养箱中培养1周。
1.4 实时荧光定量PCR用TRIzol试剂提取细胞及组织总RNA, 根据cDNA反转录试剂盒操作说明进行反转录合成cDNA的第一链。以合成的cDNA为模板, 扩增lncRNA-8439、lncRNA-1711、lncRNA-3440、lncRNA-4374、lncRNA-5958、lncRNA-6183、lncRNA-6779、lncRNA-7921、lncRNA-5834、nanog以及GAPDH基因(引物序列见表 1)。PCR反应体系为20 μL; 反应条件:94 ℃预变性2 min, 94 ℃变性15 s、60 ℃退火60 s、72 ℃延伸15 s, 共45个循环。以lncRNA-8439及nanog扩增产物荧光强度分别与GAPDH扩增产物荧光强度的比值代表其相对表达情况。以水为模板扩增的反应结果作为PCR扩增的阴性对照。
|
|
表 1 LncRNA引物及小干扰RNA序列 Tab 1 LncRNA primer and small interfering RNA sequences |
1.5 荧光原位杂交
于24孔培养板中置入10 mm×10 mm玻片, 取生长状态良好并处于对数生长期的Huh7和Hep3B细胞, 分别按照每孔6×103个细胞的密度接种于24孔板中。培养24 h后去除上清, 用4 ℃多聚甲醛溶液固定, 用0.1%的Triton X-100通透, 加入预杂交液在37 ℃下预杂交, 再分别加入18S、U6、lncRNA-8439 anti-sense和lncRNA-8439 sense探针于42 ℃下杂交16~20 h。然后用2×柠檬酸钠缓冲液冲洗, 滴加DAPI荧光染液于切片杂交区域染色10 min, 用磷酸盐缓冲液(phosphate buffer saline, PBS)清洗后在荧光显微镜下观察。
1.6 LncRNA-8439干扰实验取生长状态良好并处于对数生长期的Huh7和Hep3B细胞, 按照每孔5×105个细胞的密度接种于6孔板中, 培养24 h。构建lncRNA-8439的小干扰RNA(small interfering RNA, siRNA)序列siRNA-1、siRNA-2、siRNA-3和空载体。分别将每种细胞分为4组, 即空载体对照组、siRNA-1干扰组、siRNA-2干扰组、siRNA-3干扰组, 待细胞贴壁后, 将脂质体3000和siRNA (或空载体)的混合物滴入接种有细胞的培养板中, 混匀培养, 培养4~6 h后换为含血清的培养液, 再培养48 h后用实时荧光定量PCR方法检测siRNA的干扰效率。siRNA-1并无明显干扰效果, siRNA-2和siRNA-3能有效抑制lncRNA-8439在2种细胞系中的表达, 且siRNA-2干扰效果最佳, 故后续用siRNA-2 (序列见表 1)进行干扰实验。通过siRNA干扰敲低lncRNA-8439表达后, 采用实时荧光定量PCR和蛋白质印迹法检测2种HCC细胞中nanog的表达水平, 并观察悬浮球生长情况。
1.7 LncRNA-8439过表达实验通过NCBI数据库获取lncRNA-8439的部分序列, 根据其序列的反向互补序列(reverse complementary sequence, RCS)构建过表达载体(序列见表 1)。取生长状态良好并处于对数生长期的Huh7和Hep3B细胞, 按照每孔5×105个细胞的密度接种于6孔板中, 转染时细胞融合度达60%~70%。按照脂质体3000转染试剂说明书分别将过表达质粒与对照质粒转染至细胞, 转染48 h后提取RNA检测转染效率。Huh7过表达组lncRNA-8439的表达量约为对照组的10倍, Hep3B过表达组lncRNA-8439的表达量约为对照组的8倍。随后采用实时荧光定量PCR和蛋白质印迹法检测2种HCC细胞中nanog的表达。
1.8 蛋白质印迹分析收集细胞, 用含蛋白酶抑制剂的RIPA细胞裂解液裂解, 提取蛋白并定量。取40 μL总蛋白进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳, 转膜后以5%脱脂牛奶溶液封闭1 h, 加入一抗于4 ℃下孵育过夜, 再加入二抗常温孵育1 h。用TBST洗膜3次, 每次10 min。曝光, 以内参β-actin为标准分析目的蛋白的相对表达量。
1.9 统计学处理采用SPSS 20.0软件进行数据处理。呈正态分布的计量资料以x±s表示, 两组间比较采用两样本均数比较的t检验(若方差不齐则采用Welch校正的t检验)。检验水准(α)为0.05。
2 结果 2.1 多能因子相关lncRNA筛选应用lncRNA测序分析3对HCC原发灶与转移灶组织lncRNA表达谱, 对筛选出的差异表达lncRNA集簇分析, 直观反映出HCC原发灶与转移灶组织标本间lncRNA有较大差异(图 1)。实时定量验证结果与测序数据趋势基本一致, 证实结果是准确、可靠的。通过生物信息学手段预测后筛选出9个与多能因子nanog、oct4、sox2相关的lncRNA, 分别为lncRNA-1711、lncRNA-3440、lncRNA-4374、lncRNA-5958、lncRNA-6183、lncRNA-6779、lncRNA-7921、lncRNA-8439和lncRNA-5834。
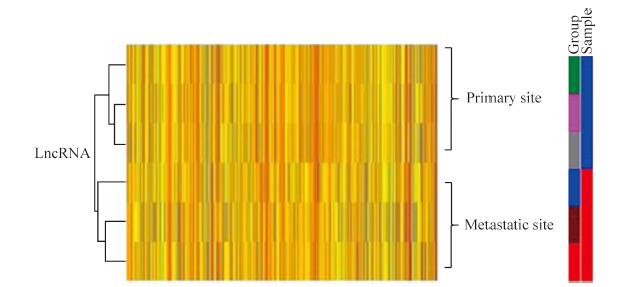
|
图 1 集簇分析3对肝癌原发灶与转移灶中的差异表达lncRNA Fig 1 Cluster analysis of differentially expressed lncRNAs in 3 pairs of primary and metastatic tissues of hepatocellular carcinoma LncRNA:Long non-coding RNA |
2.2 LncRNA-8439在2种肿瘤悬浮球中呈一致的上调表达
采用实时荧光定量PCR检测上述9个lncRNA在人肝癌细胞系Huh7和Hep3B及其相应的肿瘤悬浮球中的表达量。结果(图 2)显示9个与肿瘤干细胞多能因子相关的lncRNA中, 仅有lncRNA-8439在2种肿瘤细胞悬浮球中呈一致上调表达, 与对应的贴壁细胞系表达量相比差异均有统计学意义(P均<0.01), 故选择lncRNA-8439作进一步研究。
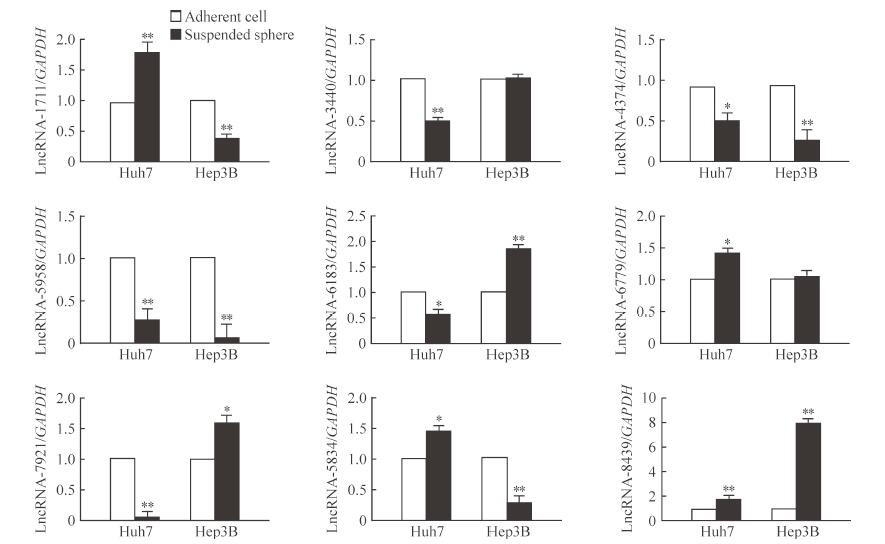
|
图 2 qPCR检测Huh7和Hep3B贴壁细胞与悬浮球中9个多能因子相关lncRNA的表达量 Fig 2 Expression levels of 9 lncRNAs related to pluripotency factor in Huh7 and Hep3B adherent cells and their corresponding suspended spheres by qPCR LncRNA:Long non-coding RNA; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase; qPCR:Real-time quantitative PCR.*P < 0.05, **P < 0.01 vs adherent cell.n=3, x±s |
2.3 LncRNA-8439与多能因子nanog相关
使用lncRNAdb工具对lncRNA-8439进行生物信息学分析, 得出lncRNA-8439与nanog结合具有可能性, LncRNA-8439的3′端存在nanog结合位点(图 3)。
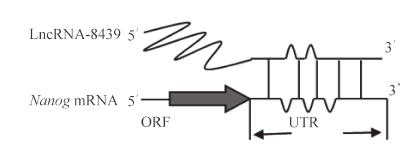
|
图 3 LncRNA-8439与多能因子nanog的结合位点 Fig 3 Binding site of lncRNA-8439 and pluripotency factor nanog LncRNA:Long non-coding RNA; ORF:Open reading frame; UTR:Untranslated region |
2.4 LncRNA-8439在HCC细胞中的分布
使用lncRNA-8439探针对Huh7和Hep3B细胞进行原位杂交, 结果(图 4)显示lncRNA-8439基本分布在细胞核中, 细胞质中并无表达。
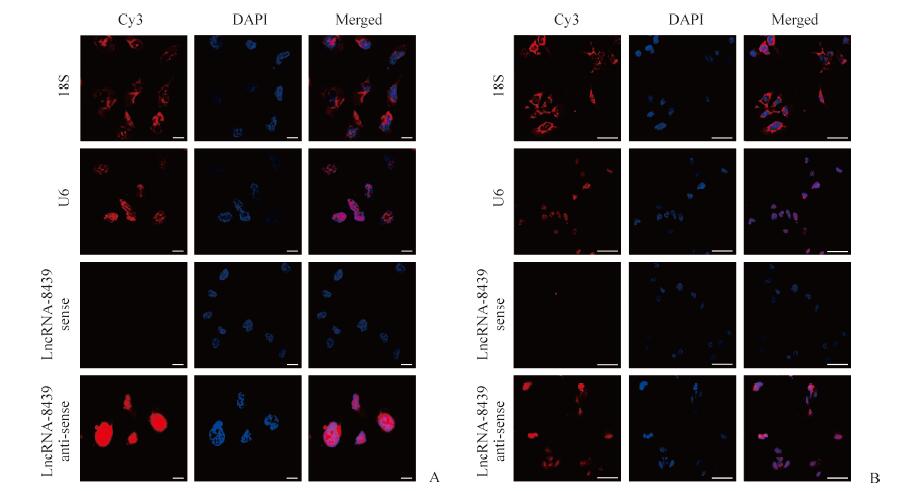
|
图 4 荧光原位杂交显示lncRNA-8439在Hep3B细胞(A)和Huh7细胞(B)中的分布 Fig 4 Fluorescence in situ hybridization showing distributions of lncRNA-8439 in Huh7 (A) and Hep3B (B) cells LncRNA:Long non-coding RNA.Scale bar:50 μm |
2.5 敲低lncRNA-8439能降低nanog表达水平和肿瘤细胞悬浮球数量
使用siRNA-2在Huh7和Hep3B细胞中敲低lncRNA-8439的表达后, 分别收集细胞利用实时荧光定量PCR和蛋白质印迹法检测nanog的表达量, 结果显示干扰组中的nanog表达量在RNA(图 5A)和蛋白水平(Huh7细胞:40.57±1.19 vs 435.81±6.75, Hep3B:18.50±0.75 vs 470.13±2.18;图 5B)均降低, 与对照组相比差异均有统计学意义(P均<0.01)。同时, 干扰组的肿瘤细胞悬浮球数量明显少于对照组(图 5C)。

|
图 5 干扰lncRNA-8439降低nanog表达水平及Hep3B和Huh7细胞悬浮球数量 Fig 5 Nanog expression level and number of suspended spheres were significantly reduced after knocking down lncRNA-8439 A, B:RNA and protein expression levels of nanog were reduced after lncRNA-8439 siRNA transfection respectively; C:Number of Hep3B and Huh7 suspended spheres was reduced after lncRNA-8439 siRNA transfection.Blank:The cells were transfected with blank vector.LncRNA:Long non-coding RNA; siRNA:Small interfering RNA; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase.**P < 0.01 vs blank.n=3, x±s.Scale bar:100 μm |
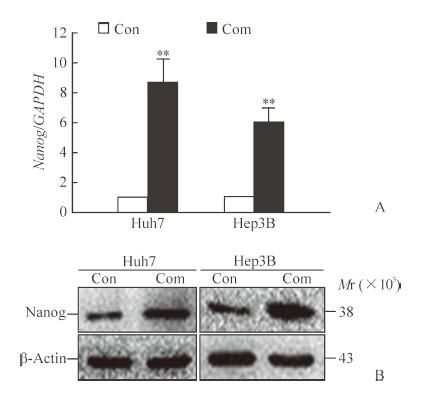
2.6 过表达lncRNA-8439能提高HCC细胞中nanog的表达水平
在Huh7和Hep3B细胞中过表达lncRNA-8439, 收集细胞利用实时荧光定量PCR和蛋白质印迹法检测nanog的表达量, 结果显示过表达组中的nanog表达量在RNA(图 6A)和蛋白水平(Huh7细胞:497.12±7.28 vs 59.30±0.86, Hep3B细胞:530.80±2.95 vs 80.31±1.27;图 6B)均增高, 差异均有统计学意义(P均<0.01)。
|
图 6 过表达lncRNA-8439使nanog表达水平提高 Fig 6 Expression level of nanog was increased afterup-regulation of lncRNA-8439 A:RNA expression; B:Protein expression.LncRNA:Long non-coding RNA; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase; Con:Control; Com:Complementary.**P < 0.01 vs control.n=3, x±s |
3 讨论
HCC在我国的发病率和病死率均居前列, 手术切除仍是目前主要的治疗方式。然而, HCC切除术后的复发和转移成为影响手术效果和患者生存期的瓶颈。HCC一旦发生转移, 即使积极治疗, 这些患者的3年生存率也不足20%[10]。包括HCC在内, 肿瘤转移导致的相关死亡率为90%[11], 因此对HCC转移机制的深入探讨有助于临床设计相关防治策略。
LncRNA在表观遗传学、转录调控及转录后调控等多个层面上调控基因的表达水平, 近年来不断被证明其空间结构、表达水平与结合分子相互作用的异常与肿瘤的发生和转移有着密不可分的关系。如lncRNA AK058003在乳腺癌组织中的表达水平显著升高, 且可能通过影响γ突触核蛋白基因(γ-synuclein gene, SNCG)的表达水平促进乳腺癌的增殖、侵袭和转移[12]; Huang等[13]报道, 在乙型肝炎相关HCC中, lncRNA Dreh可以通过抑制波形蛋白(vimentin)的表达改变细胞骨架结构, 从而抑制转移。以上研究表明lncRNA可能通过诱导某些关键基因的表达促进包括HCC在内的多种肿瘤的侵袭与转移。
恶性肿瘤细胞具有无限的自我更新和不断分化形成新的亚克隆的能力, 从而赋予肿瘤组织生长、侵袭、转移和对治疗的抗性, 这两个能力与多能因子的表达密切相关。人胚胎nanog基因定位于12号染色体, nanog能促进胚胎干细胞的自我更新, 使其保持未分化特性, 还能使终末分化细胞发生重编程而显示多能性[13]。已有一些研究表明, nanog能促进肿瘤细胞的侵袭与转移[14-15]。在HCC中, 过表达的nanog可直接结合Nodal启动子并上调其表达, 激活Smad3信号通路, 诱导上皮间质转化(epithelial mesenchymal transition, EMT), 导致细胞侵袭能力增强, 肝内转移也明显增加[16]。Nanog可促进MMTV-Wntl转基因小鼠乳腺癌细胞增殖和远端转移, 同时能促进血小板源性生长因子(platelet derived growth factor, PDGF)等EMT相关基因的表达[17]。另一方面, 近年来“肿瘤干细胞(cancer stem cell, CSC)”假说逐渐被广泛接受, CSC无限增殖、自我更新、侵袭性以及多向分化潜能被认为在肿瘤的发生、发展和侵袭、转移中发挥着重要作用[18]。Nanog通过调控IGF信号途径参与HCC CSC的维持, 并且使其体内致瘤能力更强[19]。这些研究均表明nanog参与恶性肿瘤的发展与转移, 并参与某些外界因子对肿瘤转移的调控。
本研究通过3对HCC原发灶和转移灶的lncRNA进行测序和集簇分析, 发现原发灶和转移灶之间lncRNA表达谱有较大差异。为了探索多能因子相关的lncRNA在HCC细胞中的调控作用及对HCC发展与转移的影响, 我们从差异表达的lncRNA中利用starBase V2.0工具确定了9个与多能因子oct4、nanog、sox2相关的lncRNA。通过实时荧光定量PCR检测这些lncRNA在Hep3B和Huh7细胞及其对应的肿瘤细胞悬浮球中的表达量, 挑选出lncRNA-8439作进一步研究。进一步利用生物信息学工具lncRNAdb深入分析得出lncRNA-8439与nanog结合的可能性, 明确了lncRNA-8439和nanog的结合位点, 为两者存在作用关系找到了理论依据。荧光原位杂交实验进一步验证了lncRNA-8439在HCC细胞中表达, 其主要分布于细胞核中。通过干扰及过表达实验证实lncRNA-8439能调控nanog的表达, 且影响HCC细胞的成球能力。
综上所述, nanog是lncRNA-8439调控HCC的重要靶基因, 影响HCC的转移和侵袭。但lncRNA-8439与nanog之间是否存在中间环节, 是否存在下游的调控分子以及相关的具体机制还有待进一步阐明。
| [1] |
MESSINA J P, HUMPHREYS I, FLAXMAN A, BROWN A, COOKE G S, PYBUS O G, et al. Global distribution and prevalence of hepatitis C virus genotypes[J]. Hepatology, 2015, 61: 77-87. DOI:10.1002/hep.27259 |
| [2] |
GOLDBERG D, DITAH I C, SAEIAN K, LALEHZARI M, ARONSOHN A, GOROSPE E C, et al. Changes in the prevalence of hepatitis C virus infection, nonalcoholic steatohepatitis, and alcoholic liver disease among patients with cirrhosis or liver failure on the waitlist for liver transplantation[J]. Gastroenterology, 2017, 152: 1090-1099. DOI:10.1053/j.gastro.2017.01.003 |
| [3] |
SARRAZIN C. The importance of resistance to direct antiviral drugs in HCV infection in clinical practice[J]. J Hepatol, 2016, 64: 486-504. DOI:10.1016/j.jhep.2015.09.011 |
| [4] |
PAWLOTSKY J M. Hepatitis C virus resistance to direct-acting antiviral drugs in interferon-free regimens[J]. Gastroenterology, 2016, 151: 70-86. DOI:10.1053/j.gastro.2016.04.003 |
| [5] |
KAWAGISHI N, SUDA G, ONOZAWA M, KIMURA M, MAEHARA O, OHARA M, et al. Comparing the risk of hepatitis B virus reactivation between direct-acting antiviral therapies and interferon-based therapies for hepatitis C[J]. J Viral Hepat, 2017, 24: 1098-1106. DOI:10.1111/jvh.2017.24.issue-12 |
| [6] |
CHEN G, WANG C, CHEN J, JI D, WANG Y, WU V, et al. Hepatitis B reactivation in hepatitis B and C coinfected patients treated with antiviral agents:a systematic review and meta-analysis[J]. Hepatology, 2017, 66: 13-26. DOI:10.1002/hep.v66.1 |
| [7] |
WANG C, JI D, CHEN J, SHAO Q, LI B, LIU J, et al. Hepatitis due to reactivation of hepatitis B virus in endemic areas among patients with hepatitis C treated with direct-acting antiviral agents[J]. Clin Gastroenterol Hepatol, 2017, 15: 132-136. DOI:10.1016/j.cgh.2016.06.023 |
| [8] |
PIERCE B G, BOUCHER E N, PIEPENBRINK K H, EJEMEL M, RAPP C A, THOMAS W D Jr, et al.Structure-based design of hepatitis C virus vaccines that elicit neutralizing antibody responses to a conserved epitope[J/OL].J Virol, 2017, 91: e01032-17.doi: 10.1128/JVI.01032-17.
|
| [9] |
OYARZUN P, KOBE B. Recombinant and epitope-based vaccines on the road to the market and implications for vaccine design and production[J]. Hum Vaccin Immunother, 2016, 12: 763-767. DOI:10.1080/21645515.2015.1094595 |
| [10] |
KAZI A, CHUAH C, LEOW C Y, LIM B H, MAJEED A B A, LEOW C H. Current progress of immunoinformatics approach harnessed for cellular-and antibody-dependent vaccine design[J]. Pathog Glob Health, 2018, 112: 123-131. DOI:10.1080/20477724.2018.1446773 |
| [11] |
MONTENEGRO M F, RUIZ-ESPEJO F, CAMPOY F J, MUÑOZ-DELGADO E, DE LA CADENA M P, RODRÍGUEZ-BERROCAL F J, et al. Cholinesterases are down-expressed in human colorectal carcinoma[J]. Cell Mol Life Sci, 2006, 63: 2175-2182. DOI:10.1007/s00018-006-6231-3 |
| [12] |
GUPTA S, BELLE V S, KUMBARAKERI RAJASHEKHAR R, JOGI S, PRABHU R K. Correlation of red blood cell acetylcholinesterase enzyme activity with various rbc indices[J]. Indian J Clin Biochem, 2018, 33: 445-449. DOI:10.1007/s12291-017-0691-0 |
| [13] |
WEIST R, FLÖRKEMEIER T, ROGER Y, FRANKE A, SCHWANKE K, ZWEIGERDT R, et al. Differential expression of cholinergic system components in human induced pluripotent stem cells, bone marrow-derived multipotent stromal cells, and induced pluripotent stem cell-derived multipotent stromal cells[J]. Stem Cells Dev, 2018, 27: 166-183. DOI:10.1089/scd.2017.0162 |
| [14] |
YE W, GONG X, XIE J, WU J, ZHANG X, OUYANG Q, et al. AChE deficiency or inhibition decreases apoptosis and p53 expression and protects renal function after ischemia/reperfusion[J]. Apoptosis, 2010, 15: 474-487. DOI:10.1007/s10495-009-0438-3 |
| [15] |
ZHANG X J, YANG L, ZHAO Q, CAEN J P, HE H Y, JIN Q H, et al. Induction of acetylcholinesterase expression during apoptosis in various cell types[J]. Cell Death Differ, 2002, 9: 790-800. DOI:10.1038/sj.cdd.4401034 |
| [16] |
LAYER P G. Cholinesterases during development of the avian nervous system[J]. Cell Mol Neurobiol, 1991, 11: 7-33. DOI:10.1007/BF00712798 |
| [17] |
BEN AZIZ-ALOYA R, STERNFELD M, SOREQ H. Promoter elements and alternative splicing in the human ACHE gene[J]. Prog Brain Res, 1993, 98: 147-153. DOI:10.1016/S0079-6123(08)62392-4 |
| [18] |
ZHAO Y, WANG X, WANG T, HU X, HUI X, YAN M, et al. Acetylcholinesterase, a key prognostic predictor for hepatocellular carcinoma, suppresses cell growth and induces chemosensitization[J]. Hepatology, 2011, 53: 493-503. DOI:10.1002/hep.24079 |
| [19] |
KUANG Y, SALEM N, TIAN H, KOLTHAMMER J A, CORN D J, WU C, et al. Imaging lipid synthesis in hepatocellular carcinoma with [methyl-11C]choline:correlation with in vivo metabolic studies[J]. J Nucl Med, 2011, 52: 98-106. DOI:10.2967/jnumed.110.080366 |
| [20] |
STOECK I K, LEE J, TABATA K, ROMERO-BREY I, PAUL D, SCHULT P, et al.Hepatitis C virus replication depends on endosomal cholesterol homeostasis[J/OL].J Virol, 2018, 92: e01196-17.doi: 10.1128/JVI.01196-17.
|
| [21] |
KIM J Y, WANG L, LEE J, OU J J.Hepatitis C virus induces the localization of lipid rafts to autophagosomes for its RNA replication[J/OL].J Virol, 2017, 91: e00541-17.doi: 10.1128/JVI.00541-17.
|
| [22] |
KOLETZKO L, MAHLI A, HELLERBRAND C. Development of an in vitro model to study hepatitis C virus effects on hepatocellular lipotoxicity and lipid metabolism[J]. Pathol Res Pract, 2018, 214: 1700-1706. DOI:10.1016/j.prp.2018.08.013 |
 2018, Vol. 39
2018, Vol. 39


