2. 首都医科大学北京天坛医院神经外科, 北京 100050
2. Department of Neurosurgery, Beijing Tian Tan Hospital, Capital Medical University, Beijing 100050, China
脑胶质瘤是发病率最高的原发性颅内肿瘤, 在我国20~59岁的人群中脑胶质瘤发病率约占颅内肿瘤的31.1%[1]。世界卫生组织(World Health Organization, WHO)在2007年发布的中枢神经系统肿瘤分类指南中, 根据胶质瘤的组织病理类型和恶性进展阶段, 将弥漫性胶质瘤分为WHO Ⅱ、Ⅲ、Ⅳ级[2]。其中WHO Ⅱ级胶质瘤根据组织病理形态分为星形细胞瘤、少突胶质细胞瘤和少突星形细胞瘤; WHO Ⅲ级胶质瘤分为间变性星形细胞瘤、间变性少突胶质细胞瘤及间变性少突星形细胞瘤; WHO Ⅳ级的多形性胶质母细胞瘤(glioblastoma, GBM)是侵袭性最强、恶性程度最高的胶质瘤[2]。5%~10%的GBM是由低级别的胶质瘤发展而来[3-4]。WHO Ⅱ、Ⅲ级胶质瘤进展到GBM的平均时间分别是5年和2年[5]。低级别胶质瘤进展为GBM后其恶性进展更为迅速, 患者的生活质量显著降低。基于中国脑胶质瘤基因组图谱(Chinese Glioma Genome Atlas, CGGA)的统计, WHO Ⅱ级胶质瘤患者的中位生存期为78.1个月, WHO Ⅲ级的中位生存期为37.6个月, GBM的中位生存期则仅为14.4个月[1]。
低级别胶质瘤通常会发展为恶性程度更大的GBM, 但是驱动低级别胶质瘤发生级别进展和恶性进展的生物学机制尚未被充分揭示。本团队基于CGGA全转录组表达谱芯片数据和甲基化谱数据筛选出7个可能与胶质瘤恶性进展显著相关的基因:含表皮生长因子结构域的fibulin样细胞外基质蛋白2(epidermal growth factor-containing fibulin-like extracellular matrix protein 2, EFEMP2)、F-盒结构蛋白17(F-box protein 17, FBXO17)、平足蛋白(podoplanin, PDPN)、BICD货物接头蛋白1(BICD cargo adaptor 1, BICD1)、脱氧胞苷酸脱氨酶(deoxycytidylate deaminase, DCTD)、聚合酶Ⅰ和转录释放因子(polymeraseⅠand transcript release factor, PTRF)和间充质同源盒蛋白2(mesenchyme homeobox 2, MEOX2)[6], 然而这7个重要基因在胶质瘤发生、发展中的具体作用机制尚待研究。BICD1基因编码的细胞骨架样双尾蛋白(bicaudal)D同源蛋白是一种动力蛋白货物接头蛋白, 该蛋白作为细胞内重要的动力蛋白激活动力结合蛋白(dynactin-binding protein), 可将Rab6连接至动力蛋白/动力蛋白激活蛋白复合体, 从而调节高尔基体囊泡向内质网转运[7]。已有研究报道BICD1可能与胶质母细胞瘤对替莫唑胺化学治疗和放射治疗的反应及患者的预后有关[8]。本实验中我们集中揭示BICD1的表达水平与胶质瘤病理级别的相关性, 探讨BICD1与胶质瘤恶性程度及患者预后的关系, 初步探索BICD1作为低级别胶质瘤级别进展和恶性进展的有效分子标志物的潜在作用。
1 材料和方法 1.1 胶质瘤组织标本的来源301例WHO Ⅱ~Ⅳ级脑胶质瘤标本来自CGGA中登记的2005年1月至2012年12月接受手术切除的患者, 其中男180例、女121例。所有组织标本和患者随访信息的采集均经过北京天坛医院伦理委员会审核批准。
1.2 胶质瘤病理学级别确定术中切除的新鲜肿瘤组织立即置于液氮中保存。取少量液氮保存的组织经甲醛固定及梯度脱水后进行石蜡包埋, 4 μm切片经H-E染色制片。所有胶质瘤样本的H-E染色图片均由2位具备5年以上工作经验的神经病理学专家, 根据WHO中枢神经系统肿瘤分级标准重复确定病理级别, 肿瘤细胞占组织中细胞总数的比例>80%的样本方可纳入研究。
1.3 RNA提取及全转录组表达谱芯片分析与全转录组测序将冻存肿瘤组织从液氮中取出后立即加入组织裂解液, 用手持微量匀浆机(北京天根生化科技有限公司)充分研磨破碎后加入蛋白酶K, 置于56 ℃水浴中消化15 min。用RNA提取试剂盒(德国QIAGEN公司)抽提总RNA。用NanoDrop 2000(美国Thermo公司)检测RNA浓度和纯度(纯度合格RNA的260 nm和280 nm处光密度比值为1.8~2.0), 将总RNA稀释10倍后进行琼脂糖凝胶电泳以检测RNA完整性(28S条带和18S条带清晰锐利, 且亮度之比约为2:1的RNA完整性较好)。取1 μg质检合格的总RNA用SuperScript Ⅲ反转录试剂盒(美国Invitrogen公司)合成cDNA。用人类全基因组微阵列芯片(美国Agilent公司)对组织的全基因组mRNA表达水平进行检测分析。
CGGA全转录组测序的mRNA抽提和质检步骤同上, 测序平台为Illumina HiSeq platform(美国Illumina公司)。
1.4 胶质瘤经典分子遗传学事件的检测本研究中涉及的异柠檬酸脱氢酶1(isocitrate dehydrogenase 1, IDH1)、人第10号染色体缺失的磷酸酶和张力蛋白同源基因(phosphatase and tensin homolog deleted on chromosome ten, PTEN)及肿瘤抑制蛋白P53(tumor suppressor protein P53, TP53)突变的状态, Ki67表达水平、1号染色体短臂和19号染色体长臂(1p19q)联合缺失的状态及表皮生长因子受体(epidermal growth factor receptor, EGFR)扩增等胶质瘤经典分子遗传学事件的数据均来自CGGA中对样本的标注。以上遗传学事件的检测方法在中国脑胶质瘤分子诊疗指南附录[9]中进行了详细描述。
1.5 胶质瘤分子亚型的确定本研究中胶质瘤分子亚型的分型信息来源于CGGA中对样本进行的分型标注。CGGA对样本的分型标注依据Brennan等[10]报道的方法:将样本分为4个转录特征的分子亚型(前神经元型、神经元型、经典型和间质型)。这4个分子亚型的转录特征采取Verhaak等[11]报道中采用的转录特征。
1.6 实时荧光定量PCR(real-time quantitative PCR, qPCR)随机选取WHO Ⅱ~Ⅳ级胶质瘤组织各10例, 采用动物组织总RNA提取试剂盒(北京天根生化科技有限公司)提取组织总RNA。进行RNA浓度、纯度及完整性检测后, 使用SuperScript Ⅲ反转录试剂盒(美国Invitrogen公司)合成cDNA, 然后进行qPCR操作。BICD1引物序列:上游5′-TCC ATC CAC CGG AAG GTT G-3′, 下游5′-GGC TCT GTT TCA GCT CGT TC-3′; 内参照基因GAPDH引物序列:上游5′-GGA GCG AGA TCC CTC CAA AAT-3′, 下游5′-GGC TGT TGT CAT ACT TCT CAT GG-3′。使用SYBR® Select预混液(美国Applied Biosystems公司)进行PCR扩增, 扩增条件依照该产品说明书进行。采用ΔΔCT法计算BICD1的相对表达量。
1.7 统计学处理采用GraphPad Prism 5软件进行统计学分析与绘图。呈正态分布的计量资料以x±s表示, 两组间比较采用两独立样本t检验, 多组间比较采用单因素方差分析; 采用Kaplan-Meier法进行生存分析。检验水准(α)为0.05。
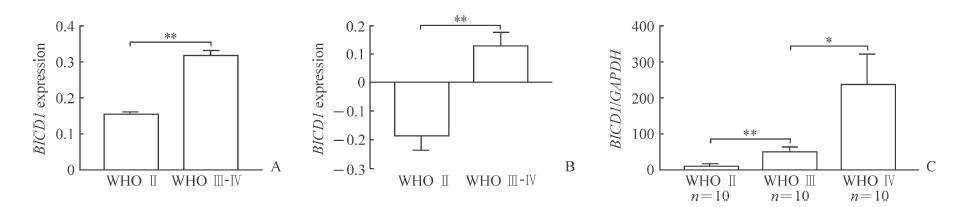
2 结果 2.1 各级别胶质瘤BICD1表达水平不同301例胶质瘤样本中WHO Ⅱ级122例(男71例、女51例, 平均年龄为39岁)、Ⅲ级51例(男29例、女22例, 平均年龄为43岁)、Ⅳ级128例(男80例、女48例, 平均年龄为47岁)。CGGA全转录组测序结果示, 高级别(WHO Ⅲ及Ⅳ级)胶质瘤样本中BICD1的表达水平高于低级别(WHO Ⅱ级)胶质瘤样本, 差异有统计学意义(t=7.901, P<0.01;图 1A)。CGGA全基因组微阵列芯片分析结果也印证了低级别和高级别胶质瘤样本中BICD1的表达水平相似的差异(t=4.026, P<0.01;图 1B)。qPCR结果显示, WHO Ⅱ级胶质瘤样本中BICD1的表达水平低于WHO Ⅲ级, 而WHO Ⅲ级胶质瘤样本中BICD1的表达水平低于WHO Ⅳ级, 差异均有统计学意义(t=3.514, P<0.01;t=2.128, P=0.037 6;图 1C)。
|
图 1 各级别胶质瘤样本中BICD1的表达水平 Fig 1 Expression levels of BICD1 in each grade of glioma samples A:BICD1 expressing data were derived from whole transcriptome sequencing database of CGGA; B:BICD1 expressing data were derived from whole transcriptome expression profile array database of CGGA; C:BICD1 expression levels were detected by qPCR.BICD1:BICD cargo adaptor 1; GAPDH:Glyceraldehyde-3-phosphate dehydrogenase; WHO:World Health Organization; CGGA:Chinese Glioma Genome Atlas; qPCR:Real-time quantitative PCR.*P < 0.05, **P < 0.01.x±s |
2.2 BICD1表达水平影响胶质瘤患者的无进展生存期和总生存期
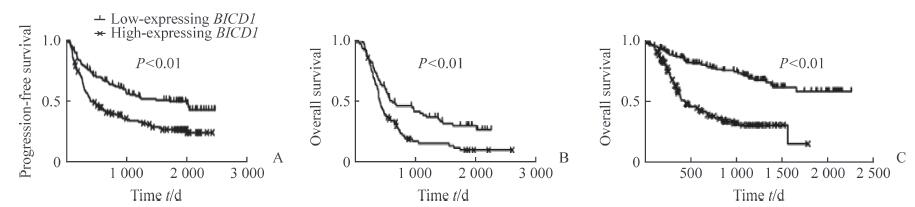
根据CGGA全转录组表达谱芯片数据库中提取出的BICD1表达值将患者分为BICD1高表达(≥-0.000 925 064)及BICD1低表达(<-0.000 925 064)两组。Kaplan-Meier分析结果示, BICD1低表达患者的无进展生存期长于BICD1高表达患者, 差异有统计学意义(P<0.01, 图 2A); 在高级别(WHO Ⅲ及Ⅳ级)胶质瘤患者中, BICD1高表达者的总生存期短于BICD1低表达者, 差异有统计学意义(P<0.01, 图 2B); CGGA全转录组测序数据库中BICD1高表达的胶质瘤(WHO Ⅱ~Ⅳ级)患者的总生存期短于BICD1低表达者, 差异有统计学意义(P<0.01, 图 2C)。
|
图 2 BICD1表达水平影响胶质瘤患者的无进展生存期及总生存期 Fig 2 BICD1 expression levels influencing progression-free survival and overall survival of glioma patients A:The WHO Ⅱ-Ⅳ grade glioma patients with high-expression level of BICD1 had shorter progression-free survival (BICD1 expression data were derived from transcriptome expression profile array database of CGGA); B:The WHO Ⅲ-Ⅳ grade glioma patients with high-expression level of BICD1 had shorter overall survival (BICD1 expression data were derived from transcriptome expression profile array database of CGGA); C:The WHO Ⅱ-Ⅳ grade glioma patients with high-expression level of BICD1 had shorter overall survival (BICD1 expression data were derived from transcriptome sequencing database of CGGA).BICD1:BICD cargo adaptor 1; WHO:World Health Organization; CGGA:Chinese Glioma Genome Atlas |
2.3 BICD1表达水平与胶质瘤经典分子病理事件的相关性
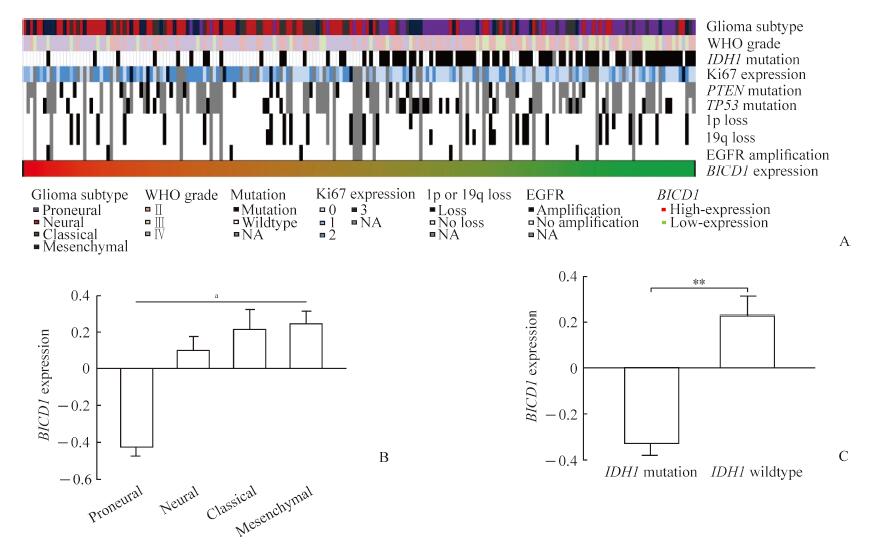
基于CGGA中对胶质瘤样本经典分子病理事件的标注(是否发生IDH1、PTEN和TP53突变、Ki67的表达水平、是否有染色体1p19q联合缺失以及是否具有EGFR扩增), 对BICD1表达水平(提取自CGGA全转录组表达谱芯片数据库)与胶质瘤经典分子病理事件的相关性进行研究。如图 3A所示, IDH1突变、染色体1p19q联合缺失在BICD1低表达的胶质瘤患者中发生率更高, PTEN突变在BICD1高表达的胶质瘤患者中发生率更高, Ki67高表达的样本在BICD1表达水平高的患者队列中更集中, 而TP53突变和EGFR扩增没有表现出与BICD1表达水平明显的相关性。
|
图 3 BICD1表达水平与胶质瘤经典分子病理事件的相关性 Fig 3 Correlation of BICD1 expression level with typical genomic alterations of glioma A:Correlation of BICD1 expression level with IDH1 mutation, Ki67 expression level, PTEN mutation, TP53 mutation, chromosome 1p19q loss and EGFR amplification (0-3 indicate the scores of Ki67 expression intension, and NA indicates missing value); B:Expression levels of BICD1 in different molecular subtypes of glioma samples; C:Expression levels of BICD1 in IDH1 mutation and wildtype glioma samples.BICD1:BICD cargo adaptor 1; IDH1:Isocitrate dehydrogenase 1; PTEN:Phosphatase and tensin homolog deleted on chromosome ten; TP53:Tumor suppressor protein 53; EGFR:Epidermal growth factor receptor; WHO:World Health Organization; CGGA:Chinese Glioma Genome Atlas.a:The difference was significant among the 4 groups.**P < 0.01.x±s |
对CGGA标注的不同分子亚型胶质瘤样本中CGGA全转录组表达谱芯片BICD1表达水平的分析结果(图 3B)示, BICD1表达水平在前神经元型、神经元型、经典型和间质型胶质瘤样本中的差异有统计学意义(F=21.8, P<0.01), 其中间质型胶质瘤样本中BICD1表达水平最高, 前神经元型胶质瘤样本中表达水平最低。
比较IDH1突变型和野生型胶质瘤样本中BICD1的表达水平, 结果(图 3C)示IDH1突变型胶质瘤样本中BICD1的表达水平低于野生型样本, 差异有统计学意义(t=7.769, P<0.01)。
3 讨论本研究探讨了BICD1与胶质瘤级别进展及恶性程度的相关性, 为进一步深入研究BICD1作为指示低级别胶质瘤发生级别进展和恶性进展的有效分子标志物奠定了基础。本研究是基于中国人群最大的脑胶质瘤生物信息及患者信息数据库开展的, 充足的样本信息保证了研究结果的可靠性和在中国人群的普适性。充分揭示BICD1表达水平与低级别胶质瘤发生进展相关性的生物学机制, 不仅有利于更好地理解胶质瘤的发生、发展的机制, 而且有助于开发阻断胶质瘤进展的有效手段。
BICD1可将Rab6连接至动力蛋白/动力蛋白激活蛋白复合体上, 参与高尔基体到内质网的胞内运输[7]。BICD1参与了神经营养因子受体的胞内运输, 对于通过囊泡内吞作用进入细胞内的神经营养因子受体, BICD1还能调节其循环再利用的途径[12]。本研究通过从CGGA中进行数据取样并分析, 发现BICD1的表达水平随着胶质瘤级别的进展逐渐升高, 提示其可能是一种与胶质瘤恶性进展相关的重要基因。依据胶质瘤组织中BICD1的表达水平, 将患者分为高表达及低表达组进行的生存分析, 发现BICD1高表达组患者的无进展生存期和总生存期均短于低表达组。这一结果进一步提示BICD1高表达可能与胶质瘤的恶性进展及患者预后有关。本研究除对全级别(WHO Ⅱ~Ⅳ级)胶质瘤的无进展生存期和总生存期进行分析外, 还分析了高级别(WHO Ⅲ和Ⅳ级)胶质瘤中BICD1的表达水平与患者总生存期的关系, 结果提示BICD1不仅可能参与了驱动低级别胶质瘤向高级别胶质瘤发生级别进展, 在高级别胶质瘤的内部, 高水平的BICD1也与胶质瘤的恶性程度有相关性。
IDH1 R132H突变(第132位的精氨酸突变为组氨酸)和染色体1p19q联合缺失是被广泛认可的指示胶质瘤患者有较好预后的分子标志物[13-14], Ki67是指示肿瘤细胞增殖的分子标志物[15], PTEN突变往往预示着恶性程度高的肿瘤表型[16]。本研究结果提示这些肿瘤恶性表型特征与BICD1表达水平显著相关。除了WHO病理级别分型, 基于基因表达特征的分子分型方法体现出胶质瘤分子亚型与神经细胞谱系的显著相关性, 有利于揭示不同胶质瘤亚型的细胞起源[11], 因而成为一种被广泛认同的胶质瘤分型方法。本研究对不同分子亚型胶质瘤中BICD1表达水平的研究结果提示, 细胞起源不同的胶质瘤可能有不同的BICD1表达水平。
鉴于胶质瘤级别进展过程中伴随着BICD1表达水平的增高, BICD1可能作为胶质瘤级别进展的分子标志物。虽然本研究提出的假说有待进一步研究和验证, 但是确认BICD1作为胶质瘤级别进展标志物的潜在作用对预测低级别胶质瘤患者的恶性进展时间和总生存期, 以及及时进行治疗方案的决策具有重要的临床意义。
| [1] |
JIANG T, MAO Y, MA W, MAO Q, YOU Y, YANG X, et al. CGCG clinical practice guidelines for the management of adult diffuse gliomas[J]. Cancer Lett, 2016, 375: 263-273. DOI:10.1016/j.canlet.2016.01.024 |
| [2] |
LOUIS D N, OHGAKI H, WIESTLER O D, CAVENEE W K, BURGER P C, JOUVET A, et al. The 2007 WHO classifcation of tumours of the central nervous system[J]. Acta Neuropathol, 2007, 114: 97-109. DOI:10.1007/s00401-007-0243-4 |
| [3] |
OHGAKI H, KLEIHUES P. Genetic pathways to primary and secondary glioblastoma[J]. Am. J Pathol, 2007, 170: 1445-1453. DOI:10.2353/ajpath.2007.070011 |
| [4] |
OHGAKI H, DESSEN P, JOURDE B, HORSTMANN S, NISHIKAWA T, DI PATRE P L, et al. Genetic pathways to glioblastoma: a population-based study[J]. Cancer Res, 2004, 64: 6892-6899. DOI:10.1158/0008-5472.CAN-04-1337 |
| [5] |
OHGAKI H, KLEIHUES P. Population-based studies on incidence, survival rates, and genetic alterations in astrocytic and oligodendroglial gliomas[J]. J Neuropathol Exp Neurol, 2005, 64: 479-489. DOI:10.1093/jnen/64.6.479 |
| [6] |
HU H, WANG Z, LI M, ZENG F, WANG K, HUANG R, et al. Gene expression and methylation analyses suggest DCTD as a prognostic factor in malignant glioma[J/OL]. Sci Rep, 2017, 7: 11568. doi: 10.1038/s41598-017-11962-y.
|
| [7] |
FUCHS E, SHORT B, BARR F A. Assay and properties of rab6 interaction with dynein-dynactin complexes[J]. Methods Enzymol, 2005, 403: 607-618. DOI:10.1016/S0076-6879(05)03053-3 |
| [8] |
HUANG S P, CHANG Y C, LOW Q H, WU A T H, CHEN C L, LIN Y F, et al. BICD1 expression, as a potential biomarker for prognosis and predicting response to therapy in patients with glioblastomas[J]. Oncotarget, 2017, 8: 113766-113791. |
| [9] |
中国脑胶质瘤协作组, 中国脑胶质瘤基因组图谱计划. 中国脑胶质瘤分子诊疗指南附录[J]. 中华神经外科杂志, 2014, 30: 523-527. DOI:10.3760/cma.j.issn.1001-2346.2014.05.025 |
| [10] |
BRENNAN C W, VERHAAK R G, MCKENNA A, CAMPOS B, NOUSHMEHR H, SALAMA S R, et al. The somatic genomic landscape of glioblastoma[J]. Cell, 2013, 155: 462-477. DOI:10.1016/j.cell.2013.09.034 |
| [11] |
VERHAAK R G, HOADLEY K A, PURDOM E, WANG V, QI Y, WILKERSON M D, et al. Integrated genomic analysis identifes clinically relevant subtypes of glioblastoma characterized by abnormalities in PDGFRA, IDH1, EGFR, and NF1[J]. Cancer Cell, 2010, 17: 98-110. DOI:10.1016/j.ccr.2009.12.020 |
| [12] |
JOHNSTON A L, LUN X, RAHN J J, LIACINI A, WANG L, HAMILTON M G, et al. The p75 neurotrophin receptor is a central regulator of glioma invasion[J/OL]. PLoS Biol, 2007, 5: e212. doi: 10.1371/journal.pbio.0050212.
|
| [13] |
YAN H, PARSONS D W, JIN G, MCLENDON R, RASHEED B A, YUAN W, et al. IDH1 and IDH2 mutations in gliomas[J]. N Engl J Med, 2009, 360: 765-773. DOI:10.1056/NEJMoa0808710 |
| [14] |
BOURNE T D, SCHIFF D. Update on molecular fndings, management and outcome in low-grade gliomas[J]. Nat Rev Neurol, 2010, 6: 695-701. DOI:10.1038/nrneurol.2010.159 |
| [15] |
PAULUS W. GFAP, Ki67 and IDH1: perhaps the golden triad of glioma immunohistochemistry[J]. Acta Neuropathol, 2009, 118: 603-604. DOI:10.1007/s00401-009-0600-6 |
| [16] |
LIU Y, HU H, ZHANG C, WANG Z, LI M, JIANG T. Integrated analysis identified genes associated with a favorable prognosis in oligodendrogliomas[J]. Genes Chromosomes Cancer, 2016, 55: 169-176. DOI:10.1002/gcc.22323 |
 2018, Vol. 39
2018, Vol. 39


