2. 海军军医大学(第二军医大学)长海医院生殖医学中心, 上海 200433
2. Department of Reproductive Medicine, Changhai Hospital, Navy Medical University(Second Military Medical University), Shanghai 200433, China
随着对肿瘤信号转导研究的不断深入,针对细胞受体、细胞周期和血管生成等性状的靶向治疗已成为继手术、放射治疗和化学治疗后的重要治疗方式[1]。迄今为止,很多靶向药物已经在临床中起到了极其重要的作用,有些已经按照循证医学原则被纳入国际肿瘤学界的标准治疗方案和规范[2]。但是,正是由于靶向治疗仅仅针对一个或几个信号转导通路,只有少数患者适合接受靶向治疗并从中获益;而且即便是靶向治疗初期效果明显的患者,随着治疗时间的延长也可能会出现获得性耐药而最终治疗失败[3]。
表皮生长因子受体酪氨酸激酶抑制剂(epidermal growth factor receptor-tyrosine kinase inhibitor,EGFR-TKI)被广泛应用于非小细胞肺癌等肿瘤的治疗,但多数患者在用药半年到2年内会发生耐药[4]。目前已发现表皮生长因子受体(epidermal growth factor receptor,EGFR)T790M突变、MET原癌基因扩增等耐药机制,但这些机制仅能解释60%~70%的耐药突变,仍有30%~40%的患者耐药机制不明[4]。粒状头样蛋白2(grainyhead-like protein 2,GRHL2)是粒状头样家族成员之一,近年研究表明,GRHL2下调可促进肿瘤细胞的上皮间质转化(epithelial-mesenchymal transition,EMT),而EMT与肿瘤耐药密切相关[5]。本研究利用浓度逐步递增的体外诱导方法[6-7]获得了EGFR-TKI耐药的肿瘤细胞株,分析GRHL2在耐药细胞株中的表达及其与EMT的关系,为探讨EGFR-TKI耐药的关键基因和相关耐药机制提供依据。
1 材料和方法 1.1 细胞株与主要试剂人结直肠癌细胞DiFi和人肺腺癌细胞HCC4006购自南京科佰生物科技有限公司;DMEM、RPMI 1640培养液和胎牛血清(fetal bovine serum,FBS)购自美国Hyclone公司;CCK-8试剂盒购自日本同仁化学公司;E-cadherin、Vimentin、GRHL2抗体购自Sigma-Aldrich公司;β-actin抗体购于北京中杉金桥生物技术有限公司;免疫印迹化学发光试剂购自美国Millipore公司;吉非替尼购自英国阿斯利康公司。脂质体转染试剂LipofectamineTM 2000购自美国Thermo Fisher Scientific公司。pcDNA3.1-GRHL2由本实验室构建。
1.2 细胞培养DiFi细胞培养于含10% FBS的DMEM培养液中,HCC4006细胞培养于含10% FBS的RPMI 1640培养液中,培养条件均为37 ℃、5% CO2。在倒置显微镜下观察细胞形态及生长状态,每隔1~2 d传代1次,取对数生长期细胞进行实验。
1.3 耐药细胞株的建立采用剂量逐步递增的方法[6-7]体外诱导耐药细胞株。取对数生长期的DiFi细胞和HCC4006细胞,加入含吉非替尼的培养液,吉非替尼从低浓度20 nmol/L开始,敏感细胞逐渐死亡,弃去培养液,用磷酸盐缓冲液(phosphate buffer saline,PBS)漂洗3次后,换不含药物的新鲜培养液继续培养至对数期。逐渐提高吉非替尼的浓度,通过反复诱导、换液传代,经过8个月筛选,直至细胞能够在1.0 μmol/L吉非替尼中稳定生长,最终得到耐药细胞株DiFi-GR、HCC4006-GR1和HCC4006-GR2。
1.4 CCK-8法检测细胞的耐药性取对数生长期的细胞,加入胰酶消化成单细胞悬液,调整细胞密度至4×104/mL,将细胞接种于96孔板,每孔100 μL,培养24 h后吸弃培养液,将含不同浓度吉非替尼的新鲜培养液加入培养孔。培养48 h后吸弃培养液并用PBS清洗,每孔加入100 pL含10% CCK-8的培养液并于37 ℃孵箱中避光孵育1.5 h。取出培养板,用酶标仪测定450 nm波长处各孔光密度(D)值,绘制细胞生长曲线,计算药物作用后细胞的半数抑制浓度(half maximal inhibitory concentration,IC50)。
1.5 RNA测序分析细胞转录组差异取对数生长期的DiFi细胞与耐药细胞株DiFi-GR、HCC4006细胞与耐药细胞株HCC4006-GR1,委托欧易生物技术有限公司进行RNA测序,提取出差异表达基因(标准为差异倍数≥1.3且P<0.05)并进行聚类分析。利用实时荧光定量PCR对差异表达基因进行验证。
1.6 耐药细胞株中GRHL2拯救实验培养耐药细胞株DiFi-GR、HCC4006-GR1至对数生长期,经胰酶消化后传代至6孔板,24 h后利用LipofectamineTM 2000转染pcDNA3.1-GRHL2质粒,以pcDNA3.1空载体转染作为阴性对照。转染后24 h传代到96孔板,加入含不同浓度吉非替尼的培养液。用蛋白质印迹法检测细胞中GRHL2的表达,CCK-8法检测DiFi-GR和HCC4006-GR1细胞过表达GRHL2后的耐药情况。
1.7 蛋白质印迹法检测EMT标志物E-cadherin、Vimentin的表达取对数生长的HCC4006细胞、DiFi细胞及相应的耐药细胞株,消化后计数并调整细胞密度,以每孔2×105个细胞的密度接种于6孔板中,将培养板置于37 ℃、5% CO2培养箱中培养24 h至其生长良好后,收集细胞。先用预冷的PBS洗2次,裂解细胞1 h,提取细胞总蛋白,收集于离心管中,沸水煮10 min,-20 ℃保存备用。以每个样品30 μg的蛋白量上样,在10%分离胶、5%浓缩胶的十二烷基硫酸钠-聚丙烯酰胺凝胶中进行电泳,转膜后室温封闭2 h。TBST洗3次,加入一抗缓慢摇动并孵育过夜。洗膜后加入用TBST稀释的二抗,化学发光显色。
1.8 CellMinerTM数据库分析GRHL2在60种细胞系中的表达利用CellMinerTM数据库[8](https://discover.nci.nih.gov/cellminer)分析GRHL2在美国国立研究所60种人类肿瘤细胞系(NCI-60)中的表达,并与Park等[9]通过蛋白质印迹法检测的NCI-60中EMT标志蛋白E-cadherin与Vimentin的比值进行比较。Park等[9]根据E-cadherin与Vimentin的比值将NCI-60细胞系分为3类:上皮细胞表型、间质细胞表型和不确定型。
1.9 统计学处理应用GraphPad Prism 19.0软件进行统计学分析,数据以x±s表示,两组间的比较采用独立样本t检验,多组间的比较采用单因素方差分析。检验水准(α)为0.05。
2 结果 2.1 吉非替尼耐药细胞株的筛选和建立选择2株对吉非替尼敏感的细胞系DiFi和HCC4006,利用浓度逐步递增的体外诱导法,获得了吉非替尼耐药DiFi克隆和耐药HCC4006克隆各2个,其中在DiFi的一个耐药克隆中发现K-Ras的扩增,未做进一步研究;其余3个克隆均未发现EGFR(T790M)、K-Ras、BRAF(V600E)、PIK3A中常见的获得性耐药突变,也没有发现MET原癌基因的扩增,提示可能存在其他的耐药机制,用于后续研究。将获得的3株耐药细胞株分别命名为DiFi-GR、HCC4006-GR1和HCC4006-GR2。采用CCK-8法检测耐药细胞对吉非替尼的敏感性,发现耐药细胞在吉非替尼作用下的活力与对照细胞相比明显增强。经计算,DiFi细胞的IC50为52 nmol/L,耐药细胞株DiFi-GR的IC50为1 600 nmol/L;HCC4006细胞的IC50为112 nmol/L,耐药细胞株HCC4006-GR1和HCC4006-GR2的IC50分别为19 000和18 000 nmol/L。
2.2 亲本细胞和耐药细胞差异表达基因的筛选利用RNA测序的方法比较DiFi与DiFi-GR、HCC4006与HCC4006-GR1的基因表达谱差异,共获得51个共同的差异表达基因。对差异表达基因进行聚类分析和实时荧光定量PCR验证,发现癌基因Ras相关蛋白Rab25、转录因子GRHL2以及上皮相关分子Claudin4和E-cadherin在吉非替尼耐药细胞株DiFi-GR、HCC4006-GR1、HCC4006-GR2中表达下调,而间质标志蛋白Vimentin和调控因子ZEB1表达上调(图 1)。
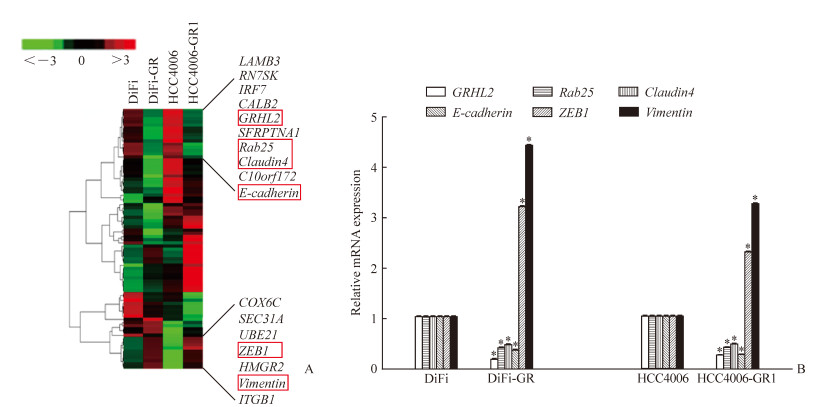
|
图 1 DiFi和HCC4006细胞与相应的吉非替尼耐药细胞株差异表达基因的筛选 Fig 1 Differentially expressed gene screening in DiFi and HCC4006 cells and corresponding gefitinib-resistant cell lines A: Clustering analysis; B: Quantitative real-time PCR. DiFi-GR and HCC4006-GR1 cells are the gefitinib-resistant cell lines. GRHL2: Grainyhead-like protein 2. *P < 0.05 vs DiFi cell or HCC4006 cell, respectively. n=3, x±s |
2.3 过表达GRHL2增强耐药细胞对吉非替尼的敏感性
在DiFi-GR和HCC4006-GR1细胞中转染pcDNA3.1-GRHL2质粒后,蛋白质印迹分析结果表明细胞中GRHL2表达量明显升高,CCK-8实验结果表明细胞对吉非替尼的敏感性明显增强(图 2)。说明GRHL2是DiFi-GR和HCC4006-GR1耐药的关键因子之一。
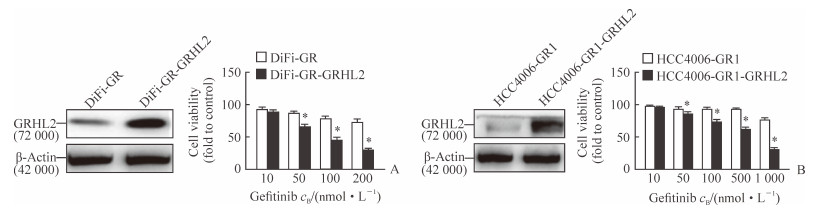
|
图 2 过表达GRHL2增强DiFi-GR和HCC4006-GR1细胞对吉非替尼的敏感性 Fig 2 Overexpression of GRHL2 enhances sensitivity of DiFi-GR and HCC4006-GR1 cells to gefitinib A: Gefitinib-resistant cell line DiFi-GR; B: Gefitinib-resistant cell line HCC4006-GR1. GRHL2: Grainyhead-like protein 2. *P < 0.05 vs DiFi-GR cell or HCC4006-GR1 cell, respectively. n=3, x±s |
2.4 耐药细胞发生EMT
利用蛋白质印迹法检测EMT关键分子的表达,结果显示耐药细胞DiFi-GR、HCC4006-GR1、HCC4006-GR2中上皮标志蛋白E-cadherin表达下调,而间质标志蛋白Vimentin表达上调(图 3)。证实耐药细胞发生了EMT。
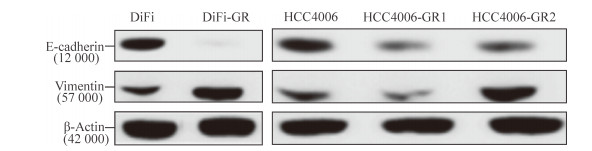
|
图 3 蛋白质印迹法检测E-cadherin和Vimentin在DiFi-GR、HCC4006-GR1和HCC4006-GR2细胞中的表达 Fig 3 Expression of E-cadherin and Vimentin in DiFi-GR, HCC4006-GR1 and HCC4006-GR2 cells by Western blotting DiFi-GR, HCC4006-GR1 and HCC4006-GR2 cells are the gefitinib-resistant cell lines |
2.5 GRHL2表达下调与EMT表型密切相关
利用CellMinerTM数据库分析GRHL2在NCI-60细胞系中的表达,结果显示GRHL2的表达与Park等[9]检测的NCI-60中E-cadherin/Vimentin的比值高度一致(图 4)。表明GRHL2表达下调可能是耐药细胞发生EMT进而耐药的原因。
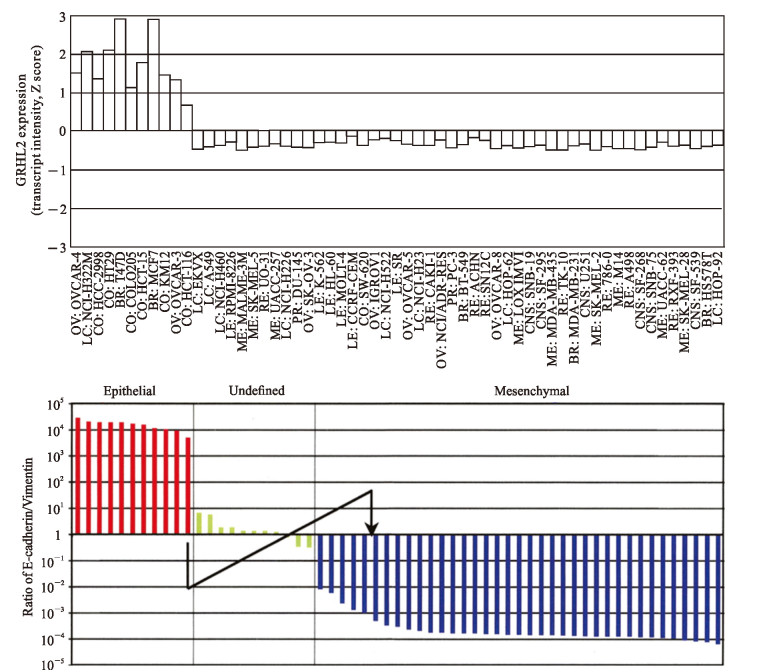
|
图 4 GRHL2表达与NCI-60细胞系的EMT表型相一致 Fig 4 GRHL2 expression is consistent with EMT phenotype of NCI-60 cell lines The ratio of E-cadherin to Vimentin was examined by Park, et al[9], and the GRHL2 expression was available at CellMinerTM database (https://discover.nci.nih.gov/cellminer/CellMiner). GRHL2: Grainyhead-like protein 2; EMT: Epithelial-mesenchymal transition |
3 讨论
目前比较明确的EGFR-TKI耐药机制主要有2种:50%的获得性耐药与EGFR的T790M突变有关[10],15%~20%与MET的扩增有关[11]。另外有研究发现,EGFR相关信号通路中K-Ras突变、PTEN表达缺失、AKT和MAPK的异常活化等也与吉非替尼的反应性有关[7]。EGFR家族的其他成员(如HER-2和HER-3)和其他生长因子受体(如IGFR-1和PDGFR)的突变和(或)活性增强在耐受机制中也发挥了重要作用[12],但这些突变所占的比例较小,目前仍有30%~40%的患者耐药机制不明[4]。
为了探讨EGFR-TKI耐药的关键基因和耐药机制,本研究选择了2种对吉非替尼敏感的细胞系:结直肠癌细胞系DiFi(高表达EGFR,野生型,K-Ras、BRAF和PIK3CA均为野生型)和HCC4006肺癌细胞系[高表达EGFR,敏感型突变(exon19del L747~E749),K-Ras为野生型][13],利用低剂量递增的体外诱导法[6-7]获得吉非替尼耐药的DiFi克隆和HCC4006克隆各2个,其中DiFi的一个耐药克隆中发现K-Ras扩增,其余3个克隆均未发现EGFR(T790M)、K-Ras、BRAF(V600E)、PIK3A中常见的获得性耐药突变,也没有发现MET原癌基因的扩增,提示可能存在其他的耐药机制。
利用RNA测序的方法对DiFi与DiFi-GR、HCC4006与HCC4006-GR1的基因表达谱进行研究,共获得了51个共同的差异表达基因。进一步对差异表达基因的功能进行聚类分析,发现了一个重要的转录因子GRHL2,经实时荧光定量PCR验证,该分子在耐药细胞株DiFi-GR、HCC4006-GR1和HCC4006-GR2中均表达下调;而GRHL2拯救实验发现在耐药细胞中过表达GRHL2后细胞恢复了对吉非替尼的敏感性。说明GRHL2是DiFi-GR、HCC4006-GR1和HCC4006-GR2耐药的关键因子之一。
近年来研究发现,EMT是西妥昔单抗耐药的机制之一[14]。Fuchs等[15]将12种人肝癌细胞系根据上皮细胞标志分子E-cadherin和间质细胞标志分子Vimentin的表达分为上皮和间质样细胞系,发现上皮细胞系对EGFR抑制剂西妥昔单抗、吉非替尼敏感,而间质细胞系对上述药物不敏感,进一步研究发现间质细胞系能够不依赖于EGFR而激活下游AKT和STAT3信号转导途径。类似的,Basu等[16]发现,间质样头颈癌细胞系中EGFR的表达降低,激活AKT和ERK信号转导途径产生耐药。Frederick等[17]的研究也表明,检测头颈癌和非小细胞肺癌细胞系EMT标志分子E-cadherin、claudin-4和claudin-7的表达能够预测细胞对吉非替尼的敏感性。
GRHL2是粒状头样蛋白3个家族成员之一,参与调控胚胎发育、上皮细胞分化、表皮屏障形成以及表皮损伤修复等一系列重要生命过程[18],并可通过靶向上皮表型基因E-cadherin和Cludin-4调控上皮顶端连接复合体的形成[19]。由于细胞损伤修复与EMT具有很多类似的特性[20],而Cieply等[21]也报道了GRHL2在乳腺癌间质样上皮细胞中表达降低。本研究利用CellMinerTM数据库[8](http://discover.nci.nih.gov/cellminer/)分析了GRHL2的表达,结果发现GRHL2的表达与NCI-60细胞系的上皮和间质表型高度[9]一致。因此,GRHL2表达下调促进细胞的EMT可能是其耐药机制之一。
总之,本研究结果提示,GRHL2可能是DiFi-GR、HCC4006-GR1和HCC4006-GR2对吉非替尼耐药的关键因子。GRHL2表达下调可能通过诱导EMT促进肿瘤细胞对吉非替尼耐药,但GRHL2调控EMT的分子机制及相关信号通路尚需进一步深入研究。
| [1] |
PÉREZ-HERRERO E, FERNÁNDEZ-MEDARDE A. Advanced targeted therapies in cancer:drug nanocarriers, the future of chemotherapy[J]. Eur J Pharm Biopharm, 2015, 93: 52-79. DOI:10.1016/j.ejpb.2015.03.018 |
| [2] |
HABER D A, GRAY N S, BASELGA J. The evolving war on cancer[J]. Cell, 2011, 145: 19-24. DOI:10.1016/j.cell.2011.03.026 |
| [3] |
REDMOND K L, PAPAFILI A, LAWLER M, VAN SCHAEYBROECK S. Overcoming resistance to targeted therapies in cancer[J]. Semin Oncol, 2015, 42: 896-908. DOI:10.1053/j.seminoncol.2015.09.028 |
| [4] |
OHASHI K, SEQUIST L V, ARCILA M E, MORAN T, CHMIELECKI J, LIN Y L, et al. Lung cancers with acquired resistance to EGFR inhibitors occasionally harbor BRAF gene mutations but lack mutations in KRAS, NRAS, or MEK1[J]. Proc Natl Acad Sci USA, 2012, 109: E2127-E2133. DOI:10.1073/pnas.1203530109 |
| [5] |
FADDAOUI A, SHETA R, BACHVAROVA M, PLANTE M, GREGOIRE J, RENAUD M C, et al. Suppression of the grainyhead transcription factor 2 gene (GRHL2) inhibits the proliferation, migration, invasion and mediates cell cycle arrest of ovarian cancer cells[J]. Cell Cycle, 2017, 16: 693-706. DOI:10.1080/15384101.2017.1295181 |
| [6] |
MISALE S, YAEGER R, HOBOR S, SCALA E, JANAKIRAMAN M, LISKA D, et al. Emergence of KRAS mutations and acquired resistance to anti-EGFR therapy in colorectal cancer[J]. Nature, 2012, 486: 532-536. DOI:10.1038/nature11156 |
| [7] |
CHMIELECKI J, FOO J, OXNARD G R, HUTCHINSON K, OHASHI K, SOMWAR R, et al. Optimization of dosing for EGFR-mutant non-small cell lung cancer with evolutionary cancer modeling[J/OL]. Sci Transl Med, 2011, 3: 90ra59. doi: 10.1126/scitranslmed.3002356.
|
| [8] |
REINHOLD W C, SUNSHINE M, LIU H, VARMA S, KOHN K W, MORRIS J, et al. CellMiner:a web-based suite of genomic and pharmacologic tools to explore transcript and drug patterns in the NCI-60 cell line set[J]. Cancer Res, 2012, 72: 3499-3511. DOI:10.1158/0008-5472.CAN-12-1370 |
| [9] |
PARK S M, GAUR A B, LENGYEL E, PETER M E. The miR-200 family determines the epithelial phenotype of cancer cells by targeting the E-cadherin repressors ZEB1 and ZEB2[J]. Genes Dev, 2008, 22: 894-907. DOI:10.1101/gad.1640608 |
| [10] |
HATA A, KATAKAMI N, YOSHIOKA H, KAJI R, MASAGO K, FUJITA S, et al. Spatiotemporal T790M heterogeneity in individual patients with EGFR-mutant non-small-cell lung cancer after acquired resistance to EGFR-TKI[J]. J Thorac Oncol, 2015, 10: 1553-1559. DOI:10.1097/JTO.0000000000000647 |
| [11] |
PRESUTTI D, SANTINI S, CARDINALI B, PAPOFF G, LALLI C, SAMPERNA S, et al. MET gene amplification and MET receptor activation are not sufficient to predict efficacy of combined MET and EGFR inhibitors in EGFR TKI-resistant NSCLC cells[J/OL]. PLoS One, 2015, 10: e0143333. doi: 10.1371/journal.pone.0143333.
|
| [12] |
LI X, XU Y, DING Y, LI C, ZHAO H, WANG J, et al. Posttranscriptional upregulation of HER3 by HER2 mRNA induces trastuzumab resistance in breast cancer[J/OL]. Mol Cancer, 2018, 17: 113. doi: 10.1186/s12943-018-0862-5.
|
| [13] |
WARE K E, HINZ T K, KLECZKO E, SINGLETON K R, MAREK L A, HELFRICH B A, et al. A mechanism of resistance to gefitinib mediated by cellular reprogramming and the acquisition of an FGF2-FGFR1 autocrine growth loop[J/OL]. Oncogenesis, 2013, 2: e39. doi: 10.1038/oncsis.2013.4.
|
| [14] |
MITRA A, MISHRA L, LI S. EMT, CTCs and CSCs in tumor relapse and drug-resistance[J]. Oncotarget, 2015, 6: 10697-10711. |
| [15] |
FUCHS B C, FUJⅡ T, DORFMAN J D, GOODWIN J M, ZHU A X, LANUTI M, et al. Epithelial-to-mesenchymal transition and integrin-linked kinase mediate sensitivity to epidermal growth factor receptor inhibition in human hepatoma cells[J]. Cancer Res, 2008, 68: 2391-2399. DOI:10.1158/0008-5472.CAN-07-2460 |
| [16] |
BASU D, NGUYEN T T, MONTONE K T, ZHANG G, WANG L P, DIEHL J A, et al. Evidence for mesenchymal-like sub-populations within squamous cell carcinomas possessing chemoresistance and phenotypic plasticity[J]. Oncogene, 2010, 29: 4170-4182. DOI:10.1038/onc.2010.170 |
| [17] |
FREDERICK B A, HELFRICH B A, COLDREN C D, ZHENG D, CHAN D, BUNN P A Jr, et al. Epithelial to mesenchymal transition predicts gefitinib resistance in cell lines of head and neck squamous cell carcinoma and non-small cell lung carcinoma[J]. Mol Cancer Ther, 2007, 6: 1683-1691. DOI:10.1158/1535-7163.MCT-07-0138 |
| [18] |
唐冠楠, 颜宏利, 王玉招, 孙树汉. 粒状头样2(GRHL2)与肿瘤[J]. 第二军医大学学报, 2013, 34: 1243-1247. TANG G N, YAN H L, WANG Y Z, SUN S H. Grainyhead-like-2(GRHL2) and carcinogenesis:an advance[J]. Acad J Sec Mil Med Univ, 2013, 34: 1243-1247. |
| [19] |
WERTH M, WALENTIN K, AUE A, SCHÖNHEIT J, WUEBKEN A, PODE-SHAKKED N, et al. The transcription factor grainyhead-like 2 regulates the molecular composition of the epithelial apical junctional complex[J]. Development, 2010, 137: 3835-3845. DOI:10.1242/dev.055483 |
| [20] |
KALLURI R. EMT:when epithelial cells decide to become mesenchymal-like cells[J]. J Clin Invest, 2009, 119: 1417-1419. DOI:10.1172/JCI39675 |
| [21] |
CIEPLY B, RILEY P 4th, PIFER P M, WIDMEYER J, ADDISON J B, IVANOV A V, et al. Suppression of the epithelial-mesenchymal transition by grainyhead-like-2[J]. Cancer Res, 2012, 72: 2440-2453. DOI:10.1158/0008-5472.CAN-11-4038 |
 2018, Vol. 39
2018, Vol. 39


