肝纤维化是多种慢性肝病的共同病理过程, 可由各型病毒性肝炎和其他损伤因素引起,最初可无症状表现, 部分经过20~40年左右发展成为预后极差的肝硬化甚至肝癌[1]。肝纤维化的早期诊治是该领域的重中之重[2]。目前肝穿刺活组织检查仍为诊断肝纤维化的金标准,但因其有创性而得不到广泛、及时的实施。微RNA(microRNA, miRNA)是大小约20~25个核苷酸序列的小分子片段,研究表明miRNA与肝纤维化发生密切相关,可通过影响肝星状细胞(hepatic stellate cell,HSC)状态而在肝纤维化进程中起重要作用[3]。近年来研究发现,miRNA可随脂质小体释放入血并受其保护而不被降解,使其稳定游走于血液与细胞之间,并通过改变相应靶基因的表达水平发挥作用[4]。而在循环血液中,亦有包括miR-122、miR-125a-5p、miR-181b等在内的多种miRNA呈显著性差异表达,在一定程度上反映了肝纤维化是否已发生以及其进展状况,为肝纤维化的无创性防治研究提供了新的视角[5-7]。
本课题组前期工作中通过芯片技术筛选了肝纤维化动物模型血液中差异表达的miRNA,结果显示miR-484在肝纤维化动物循环血中显著下调,提示miR-484或许在肝纤维化发生、发展中具有重要作用[8]。研究发现,miR-484能够通过与Fis1(mitochondrial fission 1 protein)的氨基酸编码序列结合而抑制其翻译,并在心肌细胞和肾上腺皮质癌细胞中抑制Fis1调控的线粒体分裂和凋亡[9]。本研究探讨了miR-484靶向Fis1对HSC生物学功能的影响,为肝纤维化诊疗提供新的分子靶标。
1 材料和方法 1.1 HSC-T6细胞培养HSC-T6细胞株购自美国模式培养物保藏所(American Type Culture Collection,ATCC),用含10%胎牛血清(FBS,Gibco)的DMEM培养液置于37 ℃细胞培养箱(Thermo)中培养,隔天换液,待细胞生长到铺满培养皿(Corning)80%~90%时可进行传代。
1.2 细胞转染使用广州锐博生物技术有限公司的riboFectTM CP转染试剂进行核酸分子miRNA NC(阴性对照组)或miR-484 inhibitor的转染, 转染步骤按照说明书操作进行,空白对照组不转染核酸分子。miRNA NC是与其他任何miRNA不具有同源性的miR-22(广州锐博生物技术有限公司),miR-484 inhibitor(广州锐博生物技术有限公司)是一种即用型的、经过化学修饰的成熟miRNA互补单链,可有效抑制miR-484的表达,序列为5′-AUC GGG AGG GGA CUG AGC CUG A-3′。转染24 h后,用riboMONITOR转染指示剂(广州锐博生物技术有限公司)避光染色,检测转染效率。
1.3 RNA抽提、反转录及qPCRHSC-T6转染24 h后换液,用含10% FBS的DMEM培养液继续培养24 h,TRIzol法(Invitrogen)进行总RNA提取。分别参照广州锐博生物技术有限公司反转录试剂盒及TaKaRa反转录试剂盒说明书,将miRNA及普通RNA以20 μL体系反转录为cDNA。利用SYBR Green试剂盒(TaKaRa) 20 μL反应体系和Applied Biosystems 7900型PCR仪进行qPCR。反应条件:95 ℃ 10 min;95 ℃ 15 s、60 ℃ 60 s,40个循环,在75~95 ℃条件下进行熔解曲线分析。引物序列见表 1。
|
|
表 1 qPCR引物序列 Tab 1 Gene-specific primers for the qPCR |
1.4 细胞凋亡检测
用Annexin Ⅴ-FITC/PI双染法区分活细胞、早期凋亡细胞和死亡细胞。收集处理后的细胞悬液,PBS洗2次, 用1×结合缓冲液调整细胞密度为1×106/mL,加5 μL FITC和5 μL PI,轻轻涡旋, 避光室温孵育15 min,最后加入400 μL 1×结合缓冲液, 在1 h内利用流式细胞仪(贝克曼库尔特有限公司)检测凋亡率。
1.5 蛋白质印迹分析用PMSF裂解细胞,提取总蛋白,BCA法测蛋白浓度。行SDS-PAGE,浓缩胶电压75 V,分离胶电压120 V,适时终止。使用PVDF膜(Millipore)进行转膜,5%脱脂奶粉(0.5%TBST配制)封闭1 h,然后分别加入一抗和二抗(Abcam)进行孵育。滴加ECL发光液,置于凝胶成像仪(Bio-Rad Muiltilmager)中曝光、显影。
1.6 统计学处理采用SPSS 21.0软件进行统计学分析。计量资料以x±s表示,组间比较采用独立样本t检验。检验水准(α)为0.05。
2 结果 2.1 HSC-T6细胞中miR-484 inhibitor的转染效率以125 nmol/L的浓度将miR-484 inhibitor转染HSC-T6细胞,转染效率达90%以上(图 1)。qPCR检测结果显示,转染了miR-484 inhibitor的细胞中miR-484的表达相较阴性对照组下降了92.42%,差异有统计学意义(P<0.05);而空白组与阴性对照组比较差异无统计学意义(图 2)。
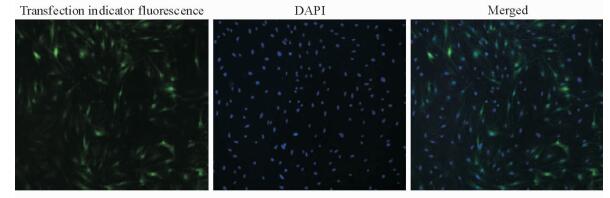
|
图 1 转染指示剂检测转染效率 Fig 1 Transfection efficiency dectected by transfection indicator The green fluorescence indicated that the cells were successfully transfected, the rate of transfection was more than 90%. DAPI: 4’, 6-Diamidino-2-phenylindole. Original magnification: ×200 |
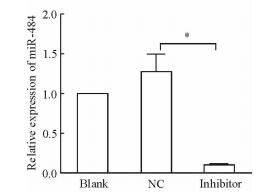
|
图 2 转染miR-484 inhibitor后miR-484表达下降(qPCR检测结果) Fig 2 MiR-484 down-regulated after transfected with inhibitor by qPCR NC: Negative control. *P < 0.05. n=3, x±s |
2.2 Fis1为miR-484的靶基因
qPCR提示,相较阴性对照组,miR-484 inhibitor组Fis1和caspase-3 mRNA的表达量分别升高至1.56倍和1.88倍,而Bcl-2 mRNA的表达量下降了72.67%,差异均有统计学意义(P<0.05);蛋白质印迹结果与qPCR结果一致(图 3)。提示Fis1为miR-484的靶基因。

|
图 3 下调miR-484后Fis1、caspase-3和Bcl-2的表达变化 Fig 3 Changes of Fis1, caspase-3 and Bcl-2 expression after transfected with miR-484 inhibitors A, B, and C: Fis1, caspase-3, and Bcl-2 mRNA expression in different groups detected by qPCR assay; D: The protein expression of Fis1, caspase-3, and Bcl-2 in different groups detected by Western blotting. NC: Negative control; Fis1: Mitochondrial fission 1 protein. *P < 0.05. n=3, x±s |
2.3 下调miR-484后纤维化指标降低
qPCR结果显示,相对于阴性对照组,miR-484 inhibitor组HSC-T6中α-平滑肌肌动蛋白(α-SMA)和Ⅰ型胶原(Col)Ⅰ的mRNA表达量分别降低了43.73%和65.43%, 差异有统计学意义(P<0.05);蛋白质印迹结果与qPCR结果一致(图 4)。提示HSC-T6活化减少,纤维化指标降低。
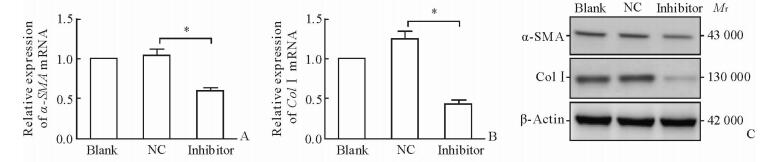
|
图 4 下调miR-484后α-SMA和Col Ⅰ表达下降 Fig 4 α-SMA and ColⅠ expression were decreased when miR-484 were down-regulated A, B: α-SMA and ColⅠ mRNA expressions in different groups detected by qPCR; C: The protein expresions of α-SMA and ColⅠ in different groups detected by Western blotting. α-SMA: α-Smooth muscle actin; Col Ⅰ: Collagen type Ⅰ; NC: Negative control. *P < 0.05. n=3, x±s |
2.4 下调miR-484后HSC-T6凋亡率升高
利用Annexin Ⅴ-FITC/PI双染法检测转染miR-484 inhibitor 48 h后细胞凋亡的改变,结果发现空白对照组细胞凋亡率为(37.27±2.94)%,阴性对照组为(32.81±3.21)%,miR-484 inhibitor组为(57.54±6.76)%(图 5)。空白对照组和阴性对照组差异无统计学意义(P=0.15),而miR-484 inhibitor组细胞凋亡率高于阴性对照组,差异有统计学意义(P<0.05)。
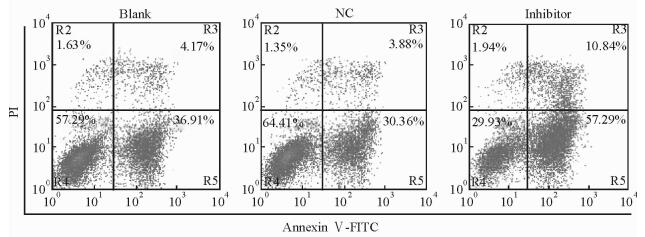
|
图 5 miR-484下调时HSC-T6细胞凋亡率升高 Fig 5 Apoptosis rate was increased when miR-484 were down-regulated PI: Propidium iodide; FITC: Fluorescein isothiocyanate; NC: Negative control |
3 讨论
因肝穿刺的有创性,肝纤维化的早期诊断成为临床上面临的一大难题。在基础研究中,研究者们更倾向于寻找有效的无创性诊疗方法[10]。目前已有多种研究表明miRNA与肝纤维化发生密切相关,miRNA可通过影响HSC状态而在肝纤维化进程中起重要作用[11]。HSC活化、增殖是肝纤维化发生、发展的关键环节。各种细胞因子包括转化生长因子(TGF)、血小板源性生长因子(PDGF)等促进HSC的活化,miRNA在HSC活化过程中同样起着重要作用[3, 12]。近年来循环血液中miRNA的表达变化与疾病发生、发展的关系在多个研究领域受到重视。研究发现循环血液中miRNA可作为无创性血液学指标,在疾病诊断、治疗及预后评价中起重要作用[13-14]。而在肝病患者循环血液中,亦有多种miRNA呈显著性差异表达,以此提示肝纤维化是否发生以及进展状况,为肝纤维化的无创性防治研究提供了新的视角[15-16]。本课题组长期从事miRNA与肝纤维化发生、发展的研究, 各领域中关于循环miRNA的研究[16-20]为我们的研究工作提供了新的视野。本课题组前期通过构建肝纤维化动物模型,并用芯片筛选了肝纤维化血液中差异表达的miRNA,芯片结果显示miR-484显著下调[8]。MiR-484的显著性差异表达,提示其可能在肝纤维化发生、发展中起重要作用,或许可作为肝纤维化发生及进展的分子标志,为肝纤维化的诊疗提供新的血液学指标。由此我们针对相关分子机制开展了本次研究。
线粒体不断发生融合和分裂,是维持细胞器保真度的两个必要过程。异常线粒体分裂参与很多疾病的发生,但人们对其调控仍然很不了解。Wang等[9]研究发现,miR-484在心肌细胞缺氧期间下调,而其靶基因线粒体分裂蛋白Fis1表达增高;Fis1是细胞线粒体分裂和凋亡过程的关键蛋白,其高表达可促进心肌细胞凋亡和心肌梗死的形成。为探索此现象是否具有细胞特异性,该研究还以肾上腺皮质癌细胞为例,验证了miR-484靶向调控Fis1对癌细胞同样具有抑制凋亡作用。
HSC是肝纤维化发生的关键细胞,本研究重点利用HSC-T6细胞株体外实验探讨了miR-484影响其功能的作用。qPCR和蛋白质印迹实验结果都表明,当miR-484表达下调时,Fis1在mRNA和蛋白水平均升高,说明在HSC-T6中miR-484同样可直接靶向调控Fis1,对HSC-T6凋亡造成影响,且在此过程中伴有凋亡相关分子caspase-3和Bcl-2的表达变化。Caspase-3是多种凋亡途径的共同下游效应分子,而Bcl-2在线粒体凋亡发生过程中起抵抗细胞凋亡的作用,二者常被作为研究细胞凋亡的关键分子[21]。本研究中,当人为下调miR-484的表达后,Bcl-2表达下降而caspase-3表达升高,Annexin Ⅴ-FITC/PI双染法检测显示HSC-T6凋亡率升高,这些研究结果提示miR-484靶向Fis1可促进HSC凋亡的产生,且此作用部分是通过线粒体途径实现的。同时,当miR-484的表达下调时,HSC-T6细胞中α-SMA和ColⅠ在mRNA和蛋白水平都下调,说明miR-484本身促进HSC活化并增加细胞外基质合成,加速肝纤维化的发生和进展。
综上所述,本研究验证了miR-484可靶向Fis1促进HSC的活化并抑制其凋亡的产生,并通过此两种途径促进肝纤维化的形成及进展。若在肝纤维化形成早期干预miR-484的表达,或许可成功阻遏肝纤维化进展。本研究为疾病的早期诊断和治疗提供了新的分子靶标。
| [1] | FRIEDMAN S L. Evolving challenges in hepatic fibrosis[J]. Nat Rev Gastroenterol Hepatol, 2010, 7: 425–436. DOI: 10.1038/nrgastro.2010.97 |
| [2] | HERNANDEZ-GEA V, FRIEDMAN S L. Pathogenesis of liver fibrosis[J]. Annu Rev Pathol, 2011, 6: 425–456. DOI: 10.1146/annurev-pathol-011110-130246 |
| [3] | GUO C J, PAN Q, CHENG T, JIANG B, CHEN G Y, LI D G. Changes in microRNAs associated with hepatic stellate cell activation status identify signaling pathways[J]. FEBS J, 2009, 276: 5163–5176. DOI: 10.1111/j.1742-4658.2009.07213.x |
| [4] | STREMERSCH S, VANDENBROUCKE R E, van WONTERGHEM E, HENDRIX A, DE SMEDT S C, RAEMDONCK K. Comparing exosome-like vesicles with liposomes for the functional cellular delivery of small RNAs[J]. J Control Release, 2016, 232: 51–61. DOI: 10.1016/j.jconrel.2016.04.005 |
| [5] | HALÁSZ T, HORVÁTH G, PÁR G, WERLING K, KISS A, SCHAFF Z, et al. miR-122 negatively correlates with liver fibrosis as detected by histology and FibroScan[J]. World J Gastroenterol, 2015, 21: 7814–7823. DOI: 10.3748/wjg.v21.i25.7814 |
| [6] | ZHENG J, ZHOU Z, XU Z, LI G, DONG P, CHEN Z, et al. Serum microRNA-125a-5p, a useful biomarker in liver diseases, correlates with disease progression[J]. Mol Med Rep, 2015, 12: 1584–1590. DOI: 10.3892/mmr.2015.3546 |
| [7] | YU F, ZHOU G, LI G, CHEN B, DONG P, ZHENG J. Serum miR-181b is correlated with hepatitis B virus replication and disease progression in chronic hepatitis B patients[J]. Dig Dis Sci, 2015, 60: 2346–2352. DOI: 10.1007/s10620-015-3649-1 |
| [8] | LI B B, LI D L, CHEN C, LIU B H, XIA C Y, WU H J, et al. Potentials of the elevated circulating miR-185 level as a biomarker for early diagnosis of HBV-related liver fibrosis[J]. Sci Rep, 2016, 6: 34157. DOI: 10.1038/srep34157 |
| [9] | WANG K, LONG B, JIAO J Q, WANG J X, LIU J P, LI Q, et al. miR-484 regulates mitochondrial network through targeting Fis1[J]. Nat Commun, 2012, 3: 781. DOI: 10.1038/ncomms1770 |
| [10] | LURIE Y, WEBB M, CYTTER-KUINT R, SHTEINGART S, LEDERKREMER G Z. Non-invasive diagnosis of liver fibrosis and cirrhosis[J]. World J Gastroenterol, 2015, 21: 11567–11583. DOI: 10.3748/wjg.v21.i41.11567 |
| [11] | TENG K Y, GHOSHAL K. Role of noncoding RNAs as biomarker and therapeutic targets for liver fibrosis[J]. Gene Expr, 2015, 16: 155–162. DOI: 10.3727/105221615X14399878166078 |
| [12] | CHEN C, WU C Q, ZHANG Z Q, YAO D K, ZHU L. Loss of expression of miR-335 is implicated in hepatic stellate cell migration and activation[J]. Exp Cell Res, 2011, 317: 1714–1725. DOI: 10.1016/j.yexcr.2011.05.001 |
| [13] | TIAN X, SHIVAPURKAR N, WU Z, HWANG J J, PISHVAIAN M J, WEINER L M, et al. Circulating microRNA profile predicts disease progression in patients receiving second-line treatment of lapatinib and capecitabine for metastatic pancreatic cancer[J]. Oncol Lett, 2016, 11: 1645–1650. |
| [14] | ARROYO J D, CHEVILLET J R, KROH E M, RUF I K, PRITCHARD C C, GIBSON D F, et al. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma[J]. Proc Natl Acad Sci USA, 2011, 108: 5003–5008. DOI: 10.1073/pnas.1019055108 |
| [15] | CERMELLI S, RUGGIERI A, MARRERO J A, IOANNOU G N, BERETTA L. Circulating microRNAs in patients with chronic hepatitis C and non-alcoholic fatty liver disease[J]. PLoS One, 2011, 6: e23937. DOI: 10.1371/journal.pone.0023937 |
| [16] | BUTT A M, RAJA A J, SIDDIQUE S, KHAN J S, SHAHID M, TAYYAB G U, et al. Parallel expression profiling of hepatic and serum microRNA-122 associated with clinical features and treatment responses in chronic hepatitis C patients[J]. Sci Rep, 2016, 6: 21510. DOI: 10.1038/srep21510 |
| [17] | YAN G, LI B, XIN X, XU M, JI G, YU H. MicroRNA-34a promotes hepatic stellate cell activation via targeting ACSL1[J]. Med Sci Monit, 2015, 21: 3008–3015. DOI: 10.12659/MSM.894000 |
| [18] | LI A, YU J, KIM H, WOLFGANG C L, CANTO M I, HRUBAN R H, et al. MicroRNA array analysis finds elevated serum miR-1290 accurately distinguishes patients with low-stage pancreatic cancer from healthy and disease controls[J]. Clin Cancer Res, 2013, 19: 3600–3610. DOI: 10.1158/1078-0432.CCR-12-3092 |
| [19] | ZEARO S, KIM E, ZHU Y, ZHAO J T, SIDHU S B, ROBINSON B G, et al. MicroRNA-484 is more highly expressed in serum of early breast cancer patients compared to healthy volunteers[J]. BMC Cancer, 2014, 14: 200. DOI: 10.1186/1471-2407-14-200 |
| [20] | KJERSEM J B, IKDAHL T, LINGJAERDE O C, GUREN T, TVEIT K M, KURE E H. Plasma microRNAs predicting clinical outcome in metastatic colorectal cancer patients receiving first-line oxaliplatin-based treatment[J]. Mol Oncol, 2014, 8: 59–67. DOI: 10.1016/j.molonc.2013.09.001 |
| [21] | CHEN C H, CHEN M F, HUANG S J, HUANG C Y, WANG H K, HSIEH W C, et al. Saikosaponin A induces apoptosis through mitochondria-dependent pathway in hepatic stellate cells[J]. Am J Chin Med, 2017, 45: 351–368. DOI: 10.1142/S0192415X17500227 |
 2017, Vol. 38
2017, Vol. 38


