2. 解放军184医院神经外科, 鹰潭 335000;
3. 中国科学院上海生物科学研究所健康科学研究所, 上海 200032
2. Department of Neurosurgery, No. 184 Hospital of PLA, Yingtan 335000, Jiangxi, China;
3. Institute of Health Sciences, Shanghai Institute for Biological Sciences, Chinese Academy of Sciences, Shanghai 200032, China
二代测序也称大规模并行测序,目前已成功地应用于部分疾病的临床诊断、发病风险评估、针对性治疗选择及产前检查等[1]。在肿瘤中进行二代测序分析有助于提高对肿瘤生物学的认识,并可能为肿瘤的基因诊断及治疗提供策略。与一代测序如Sanger测序法相比,二代测序在文库构建、测序通量、测序周期及测序成本等方面具有明显优势,同时具有高覆盖率、高灵敏度等特征[2],因此,基于多个测序技术平台的二代测序技术将为肿瘤基因组的研究带来革命性进展。
1 二代测序简介二代测序根据原理可分为连接酶合成测序及聚合酶合成测序,其过程主要包括基因组DNA片段化处理、片段两端特定接头连接、接头与反应基质锚定桥接、大规模并行PCR信号放大、单碱基延伸测序及数据分析等。目前商业化较成熟的二代测序平台主要有以DNA簇及可逆性末端终结为核心的Illumina公司Solexa测序技术、以寡核苷酸连接和双碱基编码为核心的ABI公司SOLiD测序技术,以及以微乳液PCR(emPCR)和焦磷酸测序为核心的Roche公司454测序技术,此3种测序技术的比较见表 1[3]。
2 二代测序在胶质瘤基础研究中的应用
二代测序不仅能通过全基因组、全外显子组、全转录组测序,准确发现单核苷酸变异、基因缺失或嵌入、拷贝数异常、染色体重排及微生物感染等体细胞变异[4],还能用于研究DNA与蛋白质的相互作用及表观遗传学的改变,如基因组DNA甲基化等[5]。基于该技术的多样性,近年来二代测序在肿瘤包括胶质瘤中的应用也逐步展开。
2.1 全基因组测序全基因组测序是为个人基因组及肿瘤基因组提供终极全长基因的研究,可在一次测序分析中描绘出基因组变异谱,包括单核苷酸变异、插入或缺失、复杂的结构重排及复制数量异常等[6];同时还可发现许多经典方法检测不出的基因组改变,如启动子、增强子、内含子、非编码RNA及非标注序列等非编码区的体细胞突变。此外,对于特殊类型的变异包括重复序列的重排以及最新研究发现的可能参与肿瘤发生的反转录子活化等,全基因组测序也可能为该领域提供更多相关研究信息[7-8]。
2008年第1例应用于肿瘤(急性髓性白血病)基因组的全基因组测序结果发布,标志着肿瘤基因组学研究进入了高速发展期[9]。随即肺癌、乳腺癌等多种肿瘤获得完整全基因组测序结果,并且研究的肿瘤种类及数量仍在不断扩大[10-13]。弥漫性低级别胶质瘤(low-grade glioma,LGG)是一类治疗难度高的脑肿瘤,在儿童脑肿瘤患者中最为常见[14]。一项基于39例儿童LGG及低级别胶质神经元肿瘤的全基因组测序发现了多个新的基因突变,包括BRAF、RAF1、FGFR1、MYB、MYBL1及与组蛋白功能相关的H3F3A、ATRX等基因,并观察到62%的患者仅有1种非沉默体细胞基因突变,同时还定义出WHO Ⅱ级弥漫性胶质瘤中最常见的变异是编码FGFR1的部分序列在基因内的重复及MYB的重排,且这两种变异以相互排斥的方式存在于肿瘤中[14]。胶质母细胞瘤(glioblastoma,GBM)是脑胶质瘤中发生比例及恶性程度最高的肿瘤,其发生和发展均存在极其复杂的基因改变[15]。有研究通过全基因组测序结合癌症基因组图谱(the cancer genome atlas,TCGA) DNA复制数据对GBM基因结构重排的机制进行深入分析发现,基因融合及重排等伴随在基因内断裂点富集的区域,而改变的基因结构(如凋亡调节基因FAF1) 可干扰基因表达并导致相应蛋白缺失从而促进胶质瘤细胞生长[16],此研究揭示了之前未受重视的基因失调的基因组机制。用TCGA对42例GBM样本的全基因组分析同样发现了一些基因的特殊变异,如EGFR与周围基因的重排及其基因座周围的结构变异,并观测到TERT启动子突变与TERT mRNA表达升高相关,推测其在端粒酶再激活过程中起重要作用,继而导致GBM发生[17]。
然而,全基因组测序产生的数据量极大,数据的分析、解释及存储等需较昂贵的费用,对大部分临床或科研中心而言均会存在资金压力[18]。此外,在海量的全基因组测序信息中,目前仅少数可被解释,相当一部分可能包含新颖或未知的临床相关信息暂仍不能被阐述。基于此,更具靶向性的基因组测序在先行的临床技术应用方面更具可行性。
2.2 外显子组测序外显子包括蛋白编码基因序列及RNA编码基因序列,其在基因组中所占比例约为1.8%[19],可见外显子捕获测序具有更高的兴趣区域覆盖率、更低的价格及更简便的数据分析。外显子组测序的高覆盖率使测序更易发现肿瘤的基因突变,尤其是对肿瘤细胞中低比例突变的致癌基因和抑癌基因的诊断分析更有意义。
目前全外显子组测序联合毛细管测序法等已在多种肿瘤中发挥重要作用,如GBM中IDH1突变的研究发现,该突变主要存在于部分年轻GBM患者及大部分继发性GBM患者中,且突变状态预示着相对更好的总生存时间[20]。先前的研究还表明IDH1/2突变多存在于WHO Ⅱ、Ⅲ级胶质瘤中,并可能是LGG最初发生的一类致瘤基因事件[21],然而IDH突变似乎动态改变着细胞的表观遗传学形态,其下游变化机制尚未被证实。全外显子组测序可帮助证明额外的基因突变可能共同参与IDH突变,从而引起LGG的发生过程。对两组LGG的全外显子组测序及聚焦重测序结果发现,ATRX基因具有高频突变,而ATRX编码蛋白具有端粒生物学活性并构成染色质重组复合物的核心部分,其突变可能引起端粒的选择性延长,是基因不稳定性的可能诱因。同时ATRX突变完全发生在IDH突变的肿瘤中,并与TP53突变及星形细胞分化密切相关,而与1p/19q缺失相互排斥。由此推断,ATRX突变是IDH突变胶质瘤的一个决定性分子因素[22]。
1p/19q同源性缺失同样与低级别少突胶质瘤关系密切,但目前对这种染色体缺失所引起的基因特性和功能的改变知之甚少。目前研究者正试图通过对1p/19q同源性缺失的少突胶质瘤行外显子组测序,来寻找其中与肿瘤发生相关的关键变异基因。基于二代测序,一实验组在16例1p/19q同源性缺失的少突胶质瘤中发现19q 13.2存在较高频率(13/16) 的CIC基因体细胞突变和嵌入或缺失变异,而这16例样本还具有IDH1(14/16) 或IDH2(2/16) 突变,随后在另外13例1p/19q同源性缺失的少突胶质瘤和60例1p/19q未缺失的星形胶质瘤、少突胶质瘤中行深度测序验证,结果显示CIC基因变异率分别为69%(20/29) 和2%(1/60)[23]。由此可见CIC基因变异与少突胶质瘤病理、1p/19q同源性缺失和IDH1/2突变高度相关,并可能参与少突胶质瘤发生和发展。
外显子组测序还可以只对较少的感兴趣外显子区域DNA靶向捕捉并富集后进行高通量测序。一项包含有132个GBM相关基因的外显子区域测序发现,靶向外显子测序检测有超过1 300个SNP,其中与乳腺癌相关的SART1等位基因rs660118 SNP在GBM中的频率约为乳腺癌的2倍,并在独立对照组中行Sanger测序验证正确;而与抗原相关的SNP在20/21例GBM中被检测到,提示基因变异可能与抗原的免疫原性有关联[24]。
然而由于测序基因占全基因组的比例较低,外显子组测序也存在诸多限制。首先,探针设计的原因使得只有92%外显子组可以被捕获,在此基础上仅有90%外显子组的基因变异可在充足的测序深度下被精确发现;其次,该测序技术普遍不具有对染色体易位及其他结构变异和外显子之外的区域如启动子、增强子及转录因子结合位点等的检测能力;其在基因复制数量变异方面也存在劣势[19]。
2.3 转录组测序转录组包括了总RNA、从mRNA反转录而来的cDNA、微RNA(microRNA,miRNA)及长链非编码RNA等,基于转录组的复杂性,仅根据基因组的测序结果并不能使我们完全认识到肿瘤的生物学特性等。例如,某个基因尽管存在突变,但其可能并不在肿瘤组织中表达或被甲基化或组蛋白化的等位基因沉默;而对于miRNA等可直接通过转录调节降解或控制基因表达水平,其特异性基因表达水平的改变可加强肿瘤的生物活性[25];此外还有肿瘤中RNA编辑等[12]。转录组测序可提供更丰富的数据,使我们能更全面更深入地了解肿瘤。
促癌及抑癌miRNA表达异常已被证明与肿瘤发生密切相关[26],但其调节机制尚不明确,利用全基因组测序研究miRNA的成熟过程,有助于在转录水平阐明其失调的原因。研究发现miRNA成熟形式与前体形式的比例由正常脑组织到LGG再到高级别胶质瘤(high-grode glioma, HGG)逐渐增加,并且miRNA生物合成的3个主要步骤(核加工、核质转运、胞质加工)所对应基因的表达水平在胶质瘤组织中具有系统性改变[27],由此证明了miRNA生物合成途径的失调是胶质瘤发生和发展的标志事件。
转录组测序对基因融合包括促使致瘤基因激活的框内融合等具有高度敏感性及有效性,使得新颖的肿瘤融合基因更易被发现[4]。此前实验已证明胶质瘤中野生型FGFR3并不具有致瘤性,而转录组测序发现5%~8% GBM存在FGFR3-TACC3融合基因,体外及动物模型均证实FGFR3-TACC3融合基因可促进细胞增殖及肿瘤进展[28],从而开辟了GBM病理起源机制研究的新领域。
此外,转录组测序还可应用于基因表达谱的分析、低水平或新式转录子的识别及非人类转录子的寻找等方面[29]。如Hua等[30]研究发现875个代表GBM特征的miRNA及miRNA*和811个正常脑组织中的miRNA及miRNA*,并用RT-PCR等技术验证了转录组深度测序检测miRNA表达谱的能力,这是目前GBM最大的miRNA库,其中GBM和正常脑组织中新发现有18个miRNA和16个miRNA*。
此外,由于正常细胞并不一定表达肿瘤相同基因,转录组测序对体细胞突变的检测无相应对照,因此不具有较高的检出率。另外,由于肿瘤细胞中复杂的RNA组成,测序将产生十分丰富的数据,并需要多种不同类型的分析及关联研究,这也对生物信息工作者提出了严峻考验。
2.4 表观遗传组学测序表观遗传学主要包括DNA甲基化修饰、组蛋白修饰和转录调控等,其在肿瘤发生、发展过程中的作用也逐渐被发现[31]。如DNA甲基化具有潜在的诊断及预后判断作用[32],而在胶质瘤中单个基因MGMT甲基化可用于预测患者对DNA烷化剂的治疗反应,已被纳入胶质瘤患者临床管理策略中[33]。
ChIP-Seq是将染色体免疫共沉淀与二代测序相结合的技术,可在整个基因组水平分析已知或未知的ChIP富集DNA包含的信息,全面了解蛋白与DNA的相互作用及探测组蛋白修饰改变等[34]。其中MeDIP-Seq、MethyIC-Seq、Bisulfite-Seq等技术也可在全基因组水平对DNA甲基化进行检测[35-37]。值得关注的是,近期ChIP-Seq联合Bisulfite-Seq已在表观遗传学水平阐述了一类小儿高级别胶质瘤(pediatric high-grode glioma, pHGG)的发生机制,研究指出在组蛋白H3.3-K27M突变类型的pHGG中,K27M突变可使抑制性组蛋白调节H3K27me3水平全面下降,且使DNA呈低甲基化状态,而这两种表观遗传学改变可共同建立一个特异性转录调控机制,激活K27M突变的pHGG中的基因表达,从而导致pHGG的发生[38]。
目前表观遗传学已获得了较大发展,但二代测序技术在全面描绘出表观遗传学图谱前,仍面临多重技术挑战,如ChIP-Seq等需要的测序原料数量极大,细胞数需达到500万个;组织尤其是肿瘤组织的细胞存在异型性,表观遗传测序结果的可解释归纳性受到影响等[39]。
3 二代测序在胶质瘤个体化治疗中的应用二代测序不仅可以识别个体基因中普遍的及新的变异,而且能寻找肿瘤信号通路中关键基因的改变,确定潜在的治疗靶点,为肿瘤患者的个体化治疗制定可行性方案,目前已被应用于大肠癌、恶性黑素瘤、胃癌、多发骨髓瘤及非小细胞肺癌等肿瘤的治疗中,并使患者获益[40-43]。
然而,胶质瘤具有治疗抵抗、高度复发、恶性侵袭等特性,以替莫唑胺联合放疗为标准方案的术后辅助治疗对延长胶质瘤患者的生存时间具有较大局限性[44],GBM(WHO Ⅳ级)和间变性胶质细胞瘤(WHO Ⅲ级)患者的中位生存时间分别只有12~15个月和2~5年[15, 45]。此外,由于胶质瘤在细胞及分子水平的高度异质性[46],近年来新发展的靶向治疗如分子靶向治疗等模式依然未能明显延长非选择性胶质瘤患者的总生存期[47]。二代测序正致力于寻找对当前或将来的靶向治疗策略有效的胶质瘤基因变异。一项对44例GBM样本进行的二代测序分析发现,80% GBM含有66个突变,包括最常见的TP53、PTEN、EGFR、PIK3CA等突变,而其中69% GBM的体细胞突变具有临床治疗可行性,该研究突出了靶向基因重测序在确定基因突变中的潜能,为个体化治疗的选择及判断预后提供了充足的信息[48]。二代测序不仅能确定预期基因的突变状态,同时还能明确未知的突变基因,寻找潜在有活性的分子靶点。FGFR1编码酪氨酸激酶的序列在基因内重复即通过全基因组测序在儿童LGG中发现,其可能是新生星形胶质细胞瘤进展为高级别星形胶质细胞瘤的重要基因事件,进一步研究还发现具有基因内扩增的FGFR1产物可诱导FGFR1蛋白磷酸化激活,并上调MAPK/ERK和PI3K信号通路从而导致肿瘤的发生和发展,而此通路可被特异性抑制剂阻断,表明其可作为潜在的治疗靶点对具有FGFR1基因内扩增的弥漫性LGG患者进行个体化治疗[14]。另有研究通过全外显子组测序在一类有极大治疗难度的脑干胶质瘤中检测出肿瘤特异性突变基因PPM1D,PPM1D突变与TP53突变以相互排斥的方式存在,并减弱p53激活,其在外显子6上发生截短突变可增强PPM1D活性,抑制放射诱导的DNA损伤反应关卡蛋白CHK2的激活,并消除细胞周期关卡[49]。因此,针对功能获得性突变PPM1D的脑干胶质瘤患者,PPM1D抑制剂有提供个体化靶向治疗的潜能,其小分子抑制剂研究正在展开[50]。此外,未来研究还可将PPM1D的突变状态纳入到患者对于放疗反应的疗效预测中。
总之,基于二代测序技术的胶质瘤基因谱分析已获得潜在肿瘤治疗靶点和生物标志物,对于高度异质性的胶质瘤,以此对患者进行分层管理(图 1)[51],实行个体靶向治疗将有望使胶质瘤患者获得临床最大化利益。
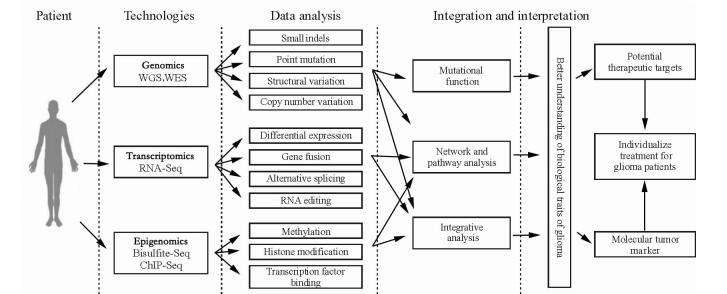
|
图 1 二代测序技术在胶质瘤研究及临床应用中的工作模式[51] Fig 1 Workflow of next-generation sequencing in glioma research and clinical application[51] WGS: Whole-genome; WES: Whole-exome technologies |
| [1] | KOBOLDT D C, STEINBERG K M, LARSON D E, WILSON R K, MARDIS E R. The next-generation sequencing revolution and its impact on genomics[J]. Cell, 2013, 155: 27–38. DOI: 10.1016/j.cell.2013.09.006 |
| [2] | SHENDURE J, JI H. Next-generation DNA sequencing[J]. Nat Biotechnol, 2008, 26: 1135–1145. DOI: 10.1038/nbt1486 |
| [3] | METZKER M L. Sequencing technologies-the next generation[J]. Nat Rev Genet, 2010, 11: 31–46. DOI: 10.1038/nrg2626 |
| [4] | MEYERSON M, GABRIEL S, GETZ G. Advances in understanding cancer genomes through second-generation sequencing[J]. Nat Rev Genet, 2010, 11: 685–696. DOI: 10.1038/nrg2841 |
| [5] | 李庆岗, 陶立. 高通量测序技术及其在生命科学中的应用[J]. 畜牧与饲料科学, 2012, 33: 25–28. DOI: 10.3969/j.issn.1672-5190.2012.12.009 |
| [6] | MELDRUM C, DOYLE M A, TOTHILL R W. Next-generation sequencing for cancer diagnostics:a practical perspective[J]. Clin Biochem Rev, 2011, 32: 177–195. |
| [7] | BECK C R, COLLIER P, MACFARLANE C, MALIG M, KIDD J M, EICHLER E E, et al. LINE-1 retrotransposition activity in human genomes[J]. Cell, 2010, 141: 1159–1170. DOI: 10.1016/j.cell.2010.05.021 |
| [8] | HUANG C R, SCHNEIDER A M, LU Y, NIRANJAN T, SHEN P, ROBINSON M A, et al. Mobile interspersed repeats are major structural variants in the human genome[J]. Cell, 2010, 141: 1171–1182. DOI: 10.1016/j.cell.2010.05.026 |
| [9] | LEY T J, MARDIS E R, DING L, FULTON B, McLELLAN M D, CHEN K, et al. DNA sequencing of a cytogenetically normal acute myeloid leukaemia genome[J]. Nature, 2008, 456: 66–72. DOI: 10.1038/nature07485 |
| [10] | PLEASANCE E D, CHEETHAM R K, STEPHENS P J, MCBRIDE D J, HUMPHRAY S J, GREENMAN C D, et al. A comprehensive catalogue of somatic mutations from a human cancer genome[J]. Nature, 2010, 463: 191–196. DOI: 10.1038/nature08658 |
| [11] | PLEASANCE E D, STEPHENS P J, O'MEARA S, MCBRIDE D J, MEYNERT A, JONES D, et al. A small-cell lung cancer genome with complex signatures of tobacco exposure[J]. Nature, 2010, 463: 184–190. DOI: 10.1038/nature08629 |
| [12] | SHAH S P, MORIN R D, KHATTRA J, PRENTICE L, PUGH T, BURLEIGH A, et al. Mutational evolution in a lobular breast tumour profiled at single nucleotide resolution[J]. Nature, 2009, 461: 809–813. DOI: 10.1038/nature08489 |
| [13] | DING L, ELLIS M J, LI S, LARSON D E, CHEN K, WALLIS J W, et al. Genome remodelling in a basal-like breast cancer metastasis and xenograft[J]. Nature, 2010, 464: 999–1005. DOI: 10.1038/nature08989 |
| [14] | ZHANG J, WU G, MILLER C P, TATEVOSSIAN R G, DALTON J D, TANG B, et al. Whole-genome sequencing identifies genetic alterations in pediatric low-grade gliomas[J]. Nat Genet, 2013, 45: 602–612. DOI: 10.1038/ng.2611 |
| [15] | WEN P Y, KESARI S. Malignant gliomas in adults[J]. N Engl J Med, 2008, 359: 492–507. DOI: 10.1056/NEJMra0708126 |
| [16] | ZHENG S, FU J, VEGESNA R, MAO Y, HEATHCOCK L E, TORRES-GARCIA W, et al. A survey of intragenic breakpoints in glioblastoma identifies a distinct subset associated with poor survival[J]. Genes Dev, 2013, 27: 1462–1472. DOI: 10.1101/gad.213686.113 |
| [17] | BRENNAN C W, VERHAAK R G, McKENNA A, CAMPOS B, NOUSHMEHR H, SALAMA S R, et al. The somatic genomic landscape of glioblastoma[J]. Cell, 2013, 155: 462–477. DOI: 10.1016/j.cell.2013.09.034 |
| [18] | MARDIS E R. The $1, 000 genome, the $100, 000 analysis?[J]. Genome Med, 2010, 2: 84. DOI: 10.1186/gm205 |
| [19] | MARDIS E R. Applying next-generation sequencing to pancreatic cancer treatment[J]. Nat Rev Gastroenterol Hepatol, 2012, 9: 477–486. DOI: 10.1038/nrgastro.2012.126 |
| [20] | PARSONS D W, JONES S, ZHANG X, LIN J C, LEARY R J, ANGENENDT P, et al. An integrated genomic analysis of human glioblastoma multiforme[J]. Science, 2008, 321: 1807–1812. DOI: 10.1126/science.1164382 |
| [21] | YAN H, PARSONS D W, JIN G, McLENDON R, RASHEED B A, YUAN W, et al. IDH1 and IDH2 mutations in gliomas[J]. N Engl J Med, 2009, 360: 765–773. DOI: 10.1056/NEJMoa0808710 |
| [22] | KANNAN K, INAGAKI A, SILBER J, GOROVETS D, ZHANG J, KASTENHUBER E R, et al. Whole-exome sequencing identifies ATRX mutation as a key molecular determinant in lower-grade glioma[J]. Oncotarget, 2012, 3: 1194–1203. DOI: 10.18632/oncotarget.v3i10 |
| [23] | YIP S, BUTTERFIELD Y S, MOROZOVA O, CHITTARANJAN S, BLOUGH M D, AN J, et al. Concurrent CIC mutations, IDH mutations, and 1p/19q loss distinguish oligodendrogliomas from other cancers[J]. J Pathol, 2012, 226: 7–16. DOI: 10.1002/path.v226.1 |
| [24] | KELLER A, HARZ C, MATZAS M, MEDER B, KATUS H A, LUDWIG N, et al. Identification of novel SNPs in glioblastoma using targeted resequencing[J]. PLoS One, 2011, 6: e18158. DOI: 10.1371/journal.pone.0018158 |
| [25] | IORIO M V, CROCE C M. MicroRNA dysregulation in cancer:diagnostics, monitoring and therapeutics. A comprehensive review[J]. EMBO Mol Med, 2012, 4: 143–159. DOI: 10.1002/emmm.201100209 |
| [26] | SANA J, HAJDUCH M, MICHALEK J, VYZULA R, SLABY O. MicroRNAs and glioblastoma:roles in core signalling pathways and potential clinical implications[J]. J Cell Mol Med, 2011, 15: 1636–1644. DOI: 10.1111/jcmm.2011.15.issue-8 |
| [27] | MOORE L M, KIVINEN V, LIU Y, ANNALA M, COGDELL D, LIU X, et al. Transcriptome and small RNA deep sequencing reveals deregulation of miRNA biogenesis in human glioma[J]. J Pathol, 2013, 229: 449–459. DOI: 10.1002/path.4109 |
| [28] | PARKER B C, ANNALA M J, COGDELL D E, GRANBERG K J, SUN Y, JI P, et al. The tumorigenic FGFR3-TACC3 gene fusion escapes miR-99a regulation in glioblastoma[J]. J Clin Invest, 2013, 123: 855–865. |
| [29] | MORRISSY A S, MORIN R D, DELANEY A, ZENG T, McDONALD H, JONES S, et al. Next-generation tag sequencing for cancer gene expression profiling[J]. Genome Res, 2009, 19: 1825–1835. DOI: 10.1101/gr.094482.109 |
| [30] | HUA D, MO F, DING D, LI L, HAN X, ZHAO N, et al. A catalogue of glioblastoma and brain microRNAs identified by deep sequencing[J]. OMICS, 2012, 16: 690–699. DOI: 10.1089/omi.2012.0069 |
| [31] | JONES P A, BAYLIN S B. The epigenomics of cancer[J]. Cell, 2007, 128: 683–692. DOI: 10.1016/j.cell.2007.01.029 |
| [32] | SHARMA S, KELLY T K, JONES P A. Epigenetics in cancer[J]. Carcinogenesis, 2010, 31: 27–36. DOI: 10.1093/carcin/bgp220 |
| [33] | HEGI M E, DISERENS A C, GORLIA T, HAMOU M F, DE TRIBOLET N, WELLER M, et al. MGMT gene silencing and benefit from temozolomide in glioblastoma[J]. N Engl J Med, 2005, 352: 997–1003. DOI: 10.1056/NEJMoa043331 |
| [34] | HOFFMAN B G, JONES S J. Genome-wide identification of DNA-protein interactions using chromatin immunoprecipitation coupled with flow cell sequencing[J]. J Endocrinol, 2009, 201: 1–13. DOI: 10.1677/JOE-08-0526 |
| [35] | LISTER R, PELIZZOLA M, DOWEN R H, HAWKINS R D, HON G, TONTI-FILIPPINI J, et al. Human DNA methylomes at base resolution show widespread epigenomic differences[J]. Nature, 2009, 462: 315–322. DOI: 10.1038/nature08514 |
| [36] | MEISSNER A, GNIRKE A, BELL G W, RAMSAHOYE B, LANDER E S, JAENISCH R. Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis[J]. Nucleic Acids Res, 2005, 33: 5868–5877. DOI: 10.1093/nar/gki901 |
| [37] | TAIWO O, WILSON G A, MORRIS T, SEISENBERGER S, REIK W, PEARCE D. Methylome analysis using MeDIP-seq with low DNA concentrations[J]. Nat Protoc, 2012, 7: 617–636. DOI: 10.1038/nprot.2012.012 |
| [38] | BENDER S, TANG Y, LINDROTH A M, HOVESTADT V, JONES D T, KOOL M, et al. Reduced H3K27me3 and DNA hypomethylation are major drivers of gene expression in K27M mutant pediatric high-grade gliomas[J]. Cancer Cell, 2013, 24: 660–672. DOI: 10.1016/j.ccr.2013.10.006 |
| [39] | SARDA S, HANNENHALLI S. Next-generation sequencing and epigenomics research:a hammer in search of nails[J]. Genomics Inform, 2014, 12: 2–11. DOI: 10.5808/GI.2014.12.1.2 |
| [40] | ROYCHOWDHURY S, IYER M K, ROBINSON D R, LONIGRO R J, WU Y M, CAO X, et al. Personalized oncology through integrative high-throughput sequencing:a pilot study[J]. Sci Transl Med, 2011, 3: 111r. DOI: 10.1126/scitranslmed.3003161 |
| [41] | HOLBROOK J D, PARKER J S, GALLAGHER K T, HALSEY W S, HUGHES A M, WEIGMAN V J, et al. Deep sequencing of gastric carcinoma reveals somatic mutations relevant to personalized medicine[J]. J Transl Med, 2011, 9: 119. DOI: 10.1186/1479-5876-9-119 |
| [42] | HALLBERG B, PALMER R H. Crizotinib-latest champion in the cancer wars?[J]. N Engl J Med, 2010, 363: 1760–1762. DOI: 10.1056/NEJMe1010404 |
| [43] | CHAPMAN M A, LAWRENCE M S, KEATS J J, CIBULSKIS K, SOUGNEZ C, SCHINZEL A C, et al. Initial genome sequencing and analysis of multiple myeloma[J]. Nature, 2011, 471: 467–472. DOI: 10.1038/nature09837 |
| [44] | ATHANASSIOU H, SYNODINOU M, MARAGOUDAKIS E, PARASKEVAIDIS M, VERIGOS C, MISAILIDOU D, et al. Randomized phase Ⅱ study of temozolomide and radiotherapy compared with radiotherapy alone in newly diagnosed glioblastoma multiforme[J]. J Clin Oncol, 2005, 23: 2372–2377. DOI: 10.1200/JCO.2005.00.331 |
| [45] | CHAMBERLAIN M C. Temozolomide:therapeutic limitations in the treatment of adult high-grade gliomas[J]. Expert Rev Neurother, 2010, 10: 1537–1544. DOI: 10.1586/ern.10.32 |
| [46] | BONAVIA R, INDA M M, CAVENEE W K, FURNARI F B. Heterogeneity maintenance in glioblastoma:a social network[J]. Cancer Res, 2011, 71: 4055–4060. DOI: 10.1158/0008-5472.CAN-11-0153 |
| [47] | van MEIR E G, HADJIPANAYIS C G, NORDEN A D, FURNARI F B. Exciting new advances in neuro-oncology:the avenue to a cure for malignant glioma[J]. CA Cancer J Clin, 2010, 60: 166–193. DOI: 10.3322/caac.20069 |
| [48] | TABONE T, ABUHUSAIN H J, NOWAK A K, Australian Genomics and Clinical Outcome of Glioma (AGOG) Network, ERBER W N, McDONALD K L. Multigene profiling to identify alternative treatment options for glioblastoma:a pilot study[J]. J Clin Pathol, 2014, 67: 550–555. DOI: 10.1136/jclinpath-2014-202173 |
| [49] | ZHANG L, CHEN L H, WAN H, YANG R, WANG Z, FENG J, et al. Exome sequencing identifies somatic gain-of-function PPM1D mutations in brainstem gliomas[J]. Nat Genet, 2014, 46: 726–730. DOI: 10.1038/ng.2995 |
| [50] | YAGI H, CHUMAN Y, KOZAKAI Y, IMAGAWA T, TAKAHASHI Y, YOSHIMURA F, et al. A small molecule inhibitor of p53-inducible protein phosphatase PPM1D[J]. Bioorg Med Chem Lett, 2012, 22: 729–732. DOI: 10.1016/j.bmcl.2011.10.084 |
| [51] | SHYR D, LIU Q. Next generation sequencing in cancer research and clinical application[J]. Biol Proced Online, 2013, 15: 4. DOI: 10.1186/1480-9222-15-4 |
 2017, Vol.
2017, Vol. 


