丙型肝炎病毒(hepatitis C virus,HCV)是引起人类慢性肝脏疾病的主要病原体之一[1]。我国的丙型肝炎感染率约为3.2%,患者可发展成肝硬化、肝癌[2]。当前临床推荐的标准治疗方案是聚乙二醇干扰素联合利巴韦林抗病毒治疗[3]。因此,对HCV载量的检测尤为重要,目前主要有免疫学和核酸检测两种方法[4-5],其中核酸检测对HCV感染患者的早发现、早诊断和早治疗大有帮助。核酸检测中的核酸提取方法主要有TRIzol法和磁珠法,TRIzol法多用于国产试剂盒,成本相对较低,样本需要量小[6];磁珠法以其高灵敏度成为HCV RNA载量检测的首选方法,多见于国外试剂盒,但成本高、样本需要量大[7]。本研究分别用TRIzol法和磁珠法提取HCV RNA,并采用qPCR对HCV进行核酸定量检测,分析两种方法用于HCV RNA载量定量检测的差异。
1 材料和方法 1.1 研究对象HCV感染者共117例,均来自2015年6月至2016年6月福建医科大学附属第二医院门诊和住院患者,其中男性68例、女性49例,平均年龄(37.1±10.2) 岁。患者的HCV基因分型为1a型13例(11.1%)、1b型51例(43.6%)、2a型36例(30.8%)、3a型5例(4.3%)、4a型2例(1.7%)、5a型2例(1.7%)和6a型8例(6.8%)。采集所有患者的血清适量冻存于-80 ℃,分析前室温溶解备用。本研究经福建医科大学附属第二医院伦理委员会同意并批准。
1.2 主要仪器与试剂磁力架由卡南吉医药科技(上海)有限公司制备;7500 qPCR仪购自美国ABI公司。TRIzol RNA提取试剂盒购自Gibco BRL公司;磁珠和配套裂解液、缓冲液由卡南吉医药科技(上海)有限公司制备;HCV核酸扩增qPCR检测试剂盒购自上海科华生物工程股份有限公司。
1.3 HCV RNA的提取TRIzol法提取:吸取100 μL血清样品,严格按照TRIzol试剂盒说明书操作提取HCV RNA。磁珠法提取:取500 μL血清样品、10 μL裂解液、100 μL缓冲液VL置入离心管中,混匀后56 ℃温浴10 min;加入500 μL缓冲液VB,20 μL磁珠,混匀后室温温浴10 min;用磁力架分离磁珠,分别采用700 μL缓冲液VW1和700 μL缓冲液VW2洗涤磁珠2次,加入50 μL洗脱液于离心管中得到HCV RNA。
1.4 核酸扩增检测HCV检测体系和反应条件严格按照试剂盒说明书进行,使用ABI配套软件定量检测结果,每次检测设置阴性、弱阳性和强阳性标准品的质控。
1.5 统计学处理用SPSS 17.0和GraphPad Prism 5.0软件进行数据处理和分析。两组间差异分析采用Bland-Altman检验;采用线性回归行相关性分析。检验水准(α)为0.05。
2 结果 2.1 两种提取方法的相关性分析分别采用TRIzol法和磁珠法提取117例HCV感染患者的血清HCV RNA,并进行HCV病毒载量检测,两种方法的检测结果取对数值进行线性回归分析,结果(图 1) 显示TRIzol法和磁珠法具有良好的线性相关性:y=0.978x+0.063 (R2=0.973)。
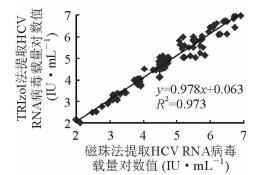
|
图 1 TRIzol法和磁珠法提取丙型肝炎病毒(HCV) RNA的相关性 |
2.2 两种提取方法的差异性分析
采用Bland-Altman检验分析两组间差异,结果(图 2) 显示,TRIzol法提取的HCV RNA的病毒载量的检测结果对数值的平均值略低于磁珠法,但两种方法提取的HCV RNA的病毒载量检测结果对数值的平均值差值仅为(0.03±0.54) IU/mL,差异无统计学意义(P > 0.05)。
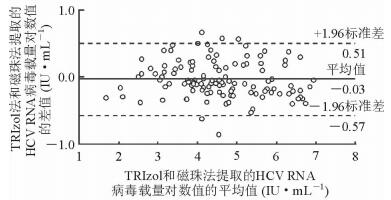
|
图 2 TRIzol法和磁珠法提取的丙型肝炎病毒(HCV)RNA病毒载量的差异分析 |
2.3 不同基因型中两种提取方法的差异性分析
比较不同基因型血清样本中TRIzol法与磁珠法提取HCV RNA的水平差异。结果(图 3) 显示,1a、1b、2a、3a、6a基因型中提取方法差异均无统计学意义,而4a和5a基因型差异有统计学意义(P < 0.05),但因为例数(n=2) 太少,分析偏差较大。
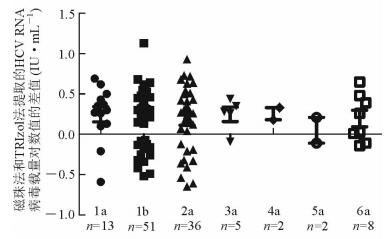
|
图 3 不同基因型(1a、1b、2a、3a、4a、5a、6a)中TRIzol法和磁珠法的差异分析 |
3 讨论
HCV是一种严重危害人类健康的重要病原体,可引起慢性肝炎、肝硬化甚至肝癌。其感染呈世界流行趋势[8]。病毒载量检测对HCV感染者的早期诊断、疗效观察和预后极为重要。在HCV RNA检测中,TRIzol法是核酸提取的常用方法,但由于新兴的磁珠法在国际上的应用,TRIzol法的关注度下降。然而,磁珠法多被整合到国际化商品化试剂盒中应用于病毒核酸全自动提取与检测平台,因价格昂贵、仪器平台检测成本过高、标本需要量大、仪器维护复杂等因素而无法在全国普及;TRIzol法成本低、样本需要量小,所需关键仪器仅为微量恒温器和常见的qPCR仪,仪器维护简单。本研究通过qPCR检测,比较了两种提取方法对HCV RNA的提取效率,旨在分析TRIzol法和磁珠法的灵敏度,评价磁珠法是否有更高的检测效率。
本实验用TRIzol法和磁珠法分别对117例HCV感染患者的血清样本进行核酸提取,并经qPCR检测定量,结果显示,两者所得的HCV RNA病毒载量具有很好的相关性(y=0.978x+0.063,R2=0.973),说明TRIzol法可以得到和磁珠法相似的结果。为进一步明确每个样本中两种检测方法的差异,本研究用Bland-Altman方法进行统计分析,可见虽然TRIzol法提取的HCV RNA病毒载量对数值的平均值比磁珠法低(0.03±0.54) IU/mL (95%置信区间),但差异无统计学意义,还不能判断TRIzol法是否比磁珠法更灵敏。本研究的相关性分析和差异性分析结果均表明,在相同的核酸检测体系下,TRIzol法的检测结果堪比磁珠法。有文献报道的磁珠法灵敏度高[9],不排除是由于加入的样本量大导致qPCR模板量大、配套检测体系更优化、检测平台更先进的原因,不能简单归结于磁珠法提取的灵敏度高。
目前为止,人类发现的HCV主要有6个型别,每种基因型又分别可以分为多种基因亚型,各型核酸序列之间相差31%~35%,而亚型序列之间相差20%~25%[10-11]。我国流行的HCV基因型主要有1、2、3、6型,其演变规律以1b型为主,其次为2a型,北方2a型是仅次于1b型的主要流行基因型,而在南方3、6型高于全国平均水平[12]。谢尧等[13]研究发现,我国主要基因型中1b型占76.64%,2a型占18.22%;西南地区的3b型(5.14%)[13]和广东等地区的6型[14]等型别较少见。不同型别HCV对药物的反应不同,如1型病毒感染者经48周标准抗病毒治疗后的好转程度明显低于2型或3型病毒感染患者24周疗程[15]。本研究比较了不同基因型中TRIzol法与磁珠法的异同,结果发现TRIzol法与磁珠法对1a、1b、2a、3a、6a型HCV的检测差异均无统计学意义,4a、5a型差异有统计学意义,但例数(n=2) 太少、分析偏差较大。由于4a和5a型HCV都不是中国人群的优势型别,所以能收集到的标本例数偏少,需要通过更多的标本对这两种型别进行检测。本研究中TRIzol法与磁珠法在优势型别1b和2a型的检测中差异无统计学意义,可推测在中国人的不同型别HCV检测中,TRIzol法与磁珠法的检测结果相当。
综上所述,在相同qPCR体系和扩增条件下,TRIzol法的灵敏度与磁珠法相比差异无统计学意义,对我国绝大部分HCV患者的检测影响相对较小。此外,由于其具有患者样本需要量小、检测成本低、仪器通用的优势,更易于普及,是一种可以在国内广泛使用的HCV RNA提取方法,同时可以更大范围地应用于试剂盒开发和HCV RNA的临床检测。
| [1] | PERUMPAIL R B, WONG R J, SCANDLING J D, HA LE D, TODO T, BONHAM C A, et al. HCV infection is associated with lower survival in simultaneous liver kidney transplant recipients in the United States[J]. Clin Transplant, 2015, 29: 920–926. DOI: 10.1111/ctr.12598 |
| [2] | YANG D R, ZHU H Z. Hepatitis C virus and antiviral innate immunity:who wins at tug-of-war?[J]. World J Gastroenterol, 2015, 21: 3786–3800. DOI: 10.3748/wjg.v21.i13.3786 |
| [3] | HILGENFELDT E G, SCHLACHTERMAN A, FIRPI R J. Hepatitis C:treatment of difficult to treat patients[J]. World J Hepatol, 2015, 7: 1953–1963. DOI: 10.4254/wjh.v7.i15.1953 |
| [4] | KIM Y K, KIM B H, JIN E S, NAM K D, JANG J Y, KIM N H, et al.[Positive predictability and predictive factors of the third generation anti-hepatitis C virus (HCV) ELISA test for HCV infection][J]. Korean J Gastroenterol, 2005, 45:181-188. |
| [5] | KIM M H, LEE H J, PARK S Y, LEE Y S, SUH J T.[Usefulness of anti-HCV ELISA test and HCV reverse transcriptase-PCR for the diagnosis of hepatits C viral infection][J]. Korean J Lab Med, 2006, 26:418-423. |
| [6] | BENEDET M, ADACHI D, WONG A, WONG S, PABBARAJU K, TELLIER R, et al. The need for a sequencing-based assay to supplement the Abbott m2000 RealTime HCV Genotype Ⅱ assay:a 1 year analysis[J]. J Clin Virol, 2014, 60: 301–304. DOI: 10.1016/j.jcv.2014.04.005 |
| [7] | BUTCHER A, ASLAM S, HEMYARI P, COWEN U, HEILEK G. HCV RNA detection in HCV antibody-positive patients with the COBAS AmpliPrep/COBAS TaqMan HCV test, v2.0 in comparison with FDA-approved nucleic acid tests[J]. J Clin Virol, 2014, 60: 336–340. DOI: 10.1016/j.jcv.2014.04.018 |
| [8] | ATTAR B M, VAN THIEL D. A new twist to a chronic HCV infection:occult hepatitis C[J/OL]. Gastroenterol Res Pract, 2015, 2015:579147. doi:10.1155/2015/579147. |
| [9] | 范公忍, 陈天宝, 李冰, 胡学玲, 曹建彪. 两种核酸提取方法对丙型肝炎病毒RNA检测效果的比较及应用评价[J]. 检验医学与临床, 2015, 12: 48–50. DOI: 10.3969/j.issn.1672-9455.2015.01.018 |
| [10] | CHOO Q L, RICHMAN K H, HAN J H, BERGER K, LEE C, DONG C, et al. Genetic organization and diversity of the hepatitis C virus[J]. Proc Nat Acad Sci USA, 1991, 88: 2451–2455. DOI: 10.1073/pnas.88.6.2451 |
| [11] | SIMMONDS P, BUKH J, COMBER C, DELÉAGE G, ENOMOTO N, FEINSTONE S, et al. Consensus proposals for a unified system of nomenclature of hepatitis C virus genotypes[J]. Hepatology, 2005, 42: 962–973. DOI: 10.1002/(ISSN)1527-3350 |
| [12] | 饶慧瑛, 李洪, 魏来, 尚佳, 陈红, 李军, 等. 中国汉族人群慢性丙型肝炎患者病毒基因型和传播高危因素的演变[C]. 中华医学会第十六次全国病毒性肝炎及肝病学术会议论文汇编, 2013. |
| [13] | 谢尧, 赵辉, 鸥蔚妮, 闫杰, 杜邵财, 徐道振, 等. 不同感染途径慢性丙型肝炎患者HCV基因型分布的差异[J]. 中华实验和临床病毒学杂志, 2004, 18: 247–250. DOI: 10.3760/cma.j.issn.1003-9279.2004.03.013 |
| [14] | 肖桂娥. 广州地区丙型肝炎患者的HCV基因型相关研究[D]. 广州: 广州医科大学, 2014. |
| [15] | WANG M, ZHANG Y, LI Z, ZHANG H, ZHANG Z, YUE D, et al. Hepatitis C virus (HCV) genotype 2a has a better virologic response to antiviral therapy than HCV genotype 1b[J]. Int J Clin Exp Med, 2015, 8: 7446–7456. |
 2017, Vol. 38
2017, Vol. 38


