大肠癌的发生是一个多因素、多阶段、多基因的过程,涉及大量肿瘤相关基因结构改变与表达异常。近年来研究已证实部分大肠癌的发生与促胃液素(gastrin,GAS)表达异常有关,部分大肠癌细胞能够通过自主产生和分泌GAS并作用于自身细胞膜受体发挥生物学效应,但其具体分子机制仍不清楚[1-4]。研究发现微RNA(microRNA, miRNA)参与调控大肠癌的发生、发展、侵袭、转移和血管生成等过程[5-6]。本研究运用酶联免疫吸附实验(ELISA)检测2014年1月—2015年12月我科71例患者大肠癌组织中的GAS含量,并利用miRNA芯片技术检测GAS高表达组和GAS阴性组miRNA的表达差异,探讨相关差异表达miRNA在GAS调节大肠癌生长中的作用及其相互关系。
1 材料和方法 1.1 标本来源收集大肠癌手术切除标本共71例,均经病理检查确诊,手术前未行放化疗。其中男38例,女33例;年龄32~71岁,平均(52±10) 岁。肿瘤部位:结肠癌39例、直肠癌32例。大体分型:溃疡型37例、隆起型28例、浸润型6例。分化程度:高分化腺癌25例、中分化腺癌27例、低分化腺癌19例。组织学类型:乳头状腺癌19例、管状腺癌33例、黏液腺癌8例、印戒细胞癌5例、未分化腺癌6例。Dukes分期:A、B期32例,C、D期39例。本研究通过皖南医学院弋矶山医院伦理委员会批准,所有患者均签署知情同意书。
1.2 试剂及仪器人GAS ELISA试剂盒购自美国R&D公司,TRIzol试剂购自美国Invitrogen公司,RNeasy微型试剂盒购自德国QIAGEN公司,miRCURYTM芯片、miRCURY Array Power标记试剂盒和缓冲液试剂盒购自丹麦Exiqon公司。GenePix 4000B芯片扫描仪、GenePix pro V6.0荧光信号处理软件均为美国Axon公司产品。
1.3 ELISA检测大肠癌组织中GAS的含量取新鲜大肠癌组织及癌旁组织各400 mg,其中200 mg液氮保存用于后期RNA的提取。另200 mg加400μL单去污剂裂解液(含PMSF)于匀浆器中进行匀浆,提取上清液于-20 ℃保存,按照ELISA试剂盒操作说明检测71例大肠癌组织中GAS的含量。GAS含量≥50.00 pg/g为阳性表达,>200.00 pg/g为强阳性表达。
1.4 样本总RNA的提取及miRNA芯片杂交 1.4.1 样本总RNA的提取和纯化选择GAS表达强阳性(高表达组)和GAS表达阴性(对照组)大肠癌患者的癌组织及相应的癌旁组织标本各4例,取50 mg冻存组织,用干净的剪刀尽可能将其碎解后放置于匀浆器中的1~2 mL的球形部位,切取过程严格按照无RNA酶的标准执行。使用生物粉碎机(美国BioSpec公司)碎解预先冻存的组织,再使用Mini-Bead-Beater-16研磨珠均质器(美国Biopec公司)进行均质化处理,然后使用RNeasy微型试剂盒纯化得到总RNA。使用NanoDrop-1000全波长紫外/可见光扫描分光光度计(美国Thermo公司)测定各标本总RNA浓度及纯度,各项指标合格后选用甲醛变性琼脂糖凝胶电泳法观察到5S、18S及28S RNA无明显降解,说明抽提RNA的质量和纯度符合基因芯片的检测要求。
1.4.2 芯片杂交按照miRCURY Array Power本标记试剂盒使用说明制备标记反应液并进行miRNA芯片杂交。本研究采用的是丹麦Exiqon公司提供的第6代芯片, 该芯片含1 891个捕获探针,涵盖人类、小鼠的所有miRNA以及这些物种有关的病毒miRNA,此外该芯片还包含miRBase数据库未收录的66个miRNA。
1.4.3 miRNA序列扫描及分析使用GenePix 4000B芯片扫描仪对所有16例标本的miRNA进行扫描,利用扫描仪配套的GenePix pro V6.0荧光信号处理软件对原始数据进行运算,取每种探针重复4次的平均值与本底荧光强度相减,通过中位数标准法获得方便分析的标准数据,计算出包括标准化信号值及样本间miRNA表达量比值等数据。最后筛选出不同组别间表达上调或下调超过3倍的miRNA,所得结果按基因表达相似程度进行分层聚类。
1.5 qPCR验证差异表达的miRNA随机选择GAS高表达组大肠癌组织中表达上调或下调3倍以上的miRNA各3个运用qPCR验证其表达水平,根据试剂盒说明书进行引物合成以及各样品的目的基因和管家基因的qPCR反应,经过与内参基因的比较及转换得到验证基因的差异倍数,分析qPCR验证结果与芯片结果是否一致。
1.6 统计学处理应用SPSS 10.0软件进行数据分析,计数资料的比较采用χ2检验。芯片检测倍数与qPCR验证倍数的比较采用配对t检验。检验水准(α)为0.05。
2 结果 2.1 大肠癌组织中的GAS含量71例大肠癌组织标本中,GAS含量≥50.00 pg/g(阳性表达)者17例(23.9%),其中>200.00 pg/g者5例,50.00~200.00 pg/g者12例。大肠癌组织中GAS表达水平与分化程度、组织学类型及Dukes分期关系密切(P均<0.05),见表 1。
|
|
表 1 大肠癌组织中促胃液素(GAS)表达与临床病理的关系 |
2.2 GenePix 4000B芯片扫描分析结果
MiRNA杂交后表达谱GenePix 4000B芯片扫描结果符合标准,信号强度高,背景均一,未见明显缺陷。芯片结果示差异表达miRNA共236个,其中与对照组相比在GAS高表达组中表达上调的miRNA 159个,表达下调的miRNA 77个。筛选出GAS高表达大肠癌组织中差异表达3倍以上的miRNA 24个,其中上调17个、下调7个(表 2、图 1)。
|
|
表 2 促胃液素(GAS)高表达组与对照组表达差异3倍以上的miRNA |

|
图 1 促胃液素(GAS)高表达组与对照组差异表达3倍以上的miRNA分层聚类图 9A: GAS高表达大肠癌组织;27A:GAS阴性表达大肠癌组织(对照组);9C:GAS高表达癌旁正常大肠黏膜组织;27C:GAS阴性表达癌旁正常大肠黏膜组织 |
2.3 qPCR验证芯片初筛结果
随机选取表达上调或下调3倍以上的miRNA各3个采用qPCR进行验证,经过与内参基因的比较及转换得到验证miRNA的差异倍数,与芯片分析结果基本一致(图 2)。
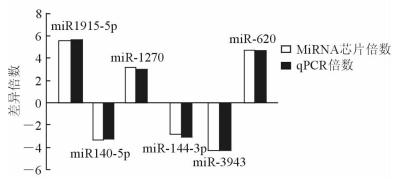
|
图 2 qPCR验证miRNA的差异倍数对比 |
3 讨论
本研究运用ELISA测得71例大肠癌患者癌组织中有17例GAS表达阳性,占总例数的23.9%;且GAS阳性表达程度与分化程度、组织学类型及Dukes分期有关,Dukes C、D期中GAS表达阳性者14例(82.4%), 中低分化癌中GAS表达阳性者有15例(88.2%),黏液腺癌、印戒细胞癌和未分化腺癌中GAS表达阳性者10例(58.8%),说明分化较差及分期较晚的大肠癌患者中GAS表达阳性的比例大,这与我们前期研究结果基本一致[7-8]。
研究证实不同类型的miRNA在大肠癌的发生、发展中扮演着不同的角色[9-10]。MiRNA芯片技术是一种快速有效地分析miRNA表达谱的方法。目前已有学者应用miRNA芯片技术筛选出众多与大肠癌生物学行为和临床特征相关的miRNA[9-10]。Ng等[11]研究发现miR-17-3p和miR-92在结直肠癌患者中表达明显增加, 且miR-92的表达水平可用来鉴别结直肠癌和炎症性肠病或正常人,敏感度89%,特异度70%。由此可见,结直肠癌患者血浆中miR-92的表达水平可作为结直肠癌早期诊断的预警指标。Yamakuchi等[12]研究发现miR-34a可通过SIRT1 (silent information regulator 1) 的3′UTR中miR-34a的结合区域抑制SIRT1表达,导致其乙酰化p53增加,p21和p53上调凋亡调控因子(PUMA)表达增加,从而调节细胞周期和凋亡。在大肠癌组织中miR-34a低表达导致细胞增殖失控。Huang等[13]对10对结肠癌组织和癌旁组织进行miRNA芯片分析发现49个表达差异的miRNA,其中miR-95有最强的促进结肠癌细胞增殖的能力,其功能主要是通过抑制下游的靶基因SNX1实现。上述结果提示,miRNA及其靶基因的异常表达与大肠癌的发生密切相关,特异性miRNA的表达模式可能与肿瘤的特性及预后有关。
本研究结果显示,GAS高表达与GAS表达阴性的大肠癌组织中miRNA的表达存在差异,表达上调的miRNA有159个,表达下调的miRNA共77个。筛选出GAS高表达且与大肠癌发病相关的3倍以上差异miRNA 24个,其中上调17个、下调7个。经检索发现,关于这些miRNA与大肠癌相关关系的研究并不多见。Pellatt等[14]通过miRNA芯片筛选发现miR-663b在人结肠癌和直肠癌中均发挥重要作用,Zhai等[15]则发现miR-140-5p可通过抑制Smad2途径和自噬抑制结直肠癌细胞的存活和侵袭,但这两项研究并未探讨相关作用是否与GAS表达水平有关。Song等[16]研究发现,miR-148b可通过下调胆囊收缩素2型受体(CCK2R)的表达阻断GAS与CCK2R结合,从而抑制大肠癌HCT-116和HT-29细胞的增殖。然而本研究并未发现miR-148b在GAS高表达与GAS表达阴性患者中存在差异表达,具体原因仍有待进一步探讨。本研究还发现了一些先前研究中尚未报道过的差异表达miRNA, 如表达上调的miR-5680和下调的miR-3943, 这些miRNA在GAS高表达的大肠癌组织中发挥何种作用值得进一步探讨。
综上所述,GAS高表达大肠癌组织中miRNA的表达存在差异,部分miRNA可能参与了GAS对大肠癌生长的调控,然而其具体机制仍不清楚。今后,我们拟建立GAS高表达大肠癌细胞模型,在此基础上进一步筛选与GAS对大肠癌发病调控相关的miRNA,并探讨其分子机制。如果能在miRNA水平有效地干预GAS对大肠癌发病的调控,将会为GAS高表达的大肠癌患者的综合治疗寻找到新的途径和潜在靶点。
| [1] | HAN Y M, PARK J M, PARK S H, HAHM K B, HONG S P, KIM E H. Gastrin promotes intestinal polyposis through cholecystokinin-B receptor-mediated proliferative signaling and fostering tumor microenvironment[J]. J Physiol Pharmacol, 2013, 64: 429–437. |
| [2] | ZYGULSKA A L, FURGALA A, KRZEMIENIECKI K, KASZUBA-ZWOINSKA J, THOR P. Enterohormonal disturbances in colorectal cancer patients[J]. Neoplasma, 2017, 64: 421–429. DOI: 10.4149/neo_2017_313 |
| [3] | MARSHALL K M, LAVAL M, ESTACIO O, HUDSON D F, KALITSIS P, SHULKES A, et al. Activation by zinc of the human gastrin gene promoter in colon cancer cells in vitro and in vivo[J]. Metallomics, 2015, 7: 1390–1398. DOI: 10.1039/C5MT00147A |
| [4] | PONNUSAMY S, LATTMANN E, LATTMANN P, THIYAGARAJAN T, PADINJARETHALAKAL B N, NARAYANAN R. Novel, isoform-selective, cholecystokinin A receptor antagonist inhibits colon and pancreatic cancers in preclinical models through novel mechanism of action[J]. Oncol Rep, 2016, 35: 2097–2106. |
| [5] | ZHANG X, XU J, JIANG T, LIU G, WANG D, LU Y. MicroRNA-195 suppresses colorectal cancer cells proliferation via targeting FGF2 and regulating Wnt/β-catenin pathway[J]. Am J Cancer Res, 2016, 6: 2631–2640. |
| [6] | KIJIMA T, HAZAMA S, TSUNEDOMI R, TANAKA H, TAKENOUCHI H, KANEKIYO S, et al. MicroRNA-6826 and -6875 in plasma are valuable non-invasive biomarkers that predict the efficacy of vaccine treatment against metastatic colorectal cancer[J]. Oncol Rep, 2017, 37: 23–30. |
| [7] | MAO J D, WU P, XIA X H, WU J, HUANG H. Correlation between expression of gastrin, somatotatin and cell apoptosis regulation gene bcl-2/bas in large intestine carcinoma[J]. World J Gastroenterol, 2005, 11: 721–725. DOI: 10.3748/wjg.v11.i5.721 |
| [8] | MAO J D, WU P, YANG Y L, HU J Q, HUANG W B, XU G Q. Relationship between expression of gastrin, somatostatin, Fas/FasL and caspases in large intestinal carcinoma[J]. World J Gastroenterol, 2008, 14: 2802–2809. DOI: 10.3748/wjg.14.2802 |
| [9] | XIE T, HUANG M, WANG Y, WANG L, CHEN C, CHU X. MicroRNAs as regulators, biomarkers and therapeutic targets in the drug resistance of colorectal cancer[J]. Cell Physiol Biochem, 2016, 40(1/2): 62–76. |
| [10] | FENG L, MA H, CHANG L, ZHOU X, WANG N, ZHAO L, et al. Role of microRNA-141 in colorectal cancer with lymph node metastasis[J]. Exp Ther Med, 2016, 12: 3405–3410. |
| [11] | NG E K, CHONG W W, JIN H, LAM E K, SHIN V Y, YU J, et al. Differential expression of microRNAs in plasma of patients with colorectal cancer: a potential marker for colorectal cancer screening[J]. Gut, 2009, 58: 1375–1381. DOI: 10.1136/gut.2008.167817 |
| [12] | YAMAKUCHI M, FERLITO M, LOWENSTEIN C J. miR-34a repression of SIRT1 regulates apoptosis[J]. Proc Natl Acad Sci USA, 2008, 105: 13421–13426. DOI: 10.1073/pnas.0801613105 |
| [13] | HUANG Z, HUANG S, WANG Q, LIANG L, NI S, WANG L, et al. MicroRNA-95 promotes cell proliferation and targets sorting Nexin 1 in human colorectal carcinoma[J]. Cancer Res, 2011, 71: 2582–2589. DOI: 10.1158/0008-5472.CAN-10-3032 |
| [14] | PELLATT D F, STEVENS J R, WOLFF R K, MULLANY L E, HERRICK J S, SAMOWITZ W, et al. Expression profiles of miRNA subsets distinguish human colorectal carcinoma and normal colonic mucosa[J/OL]. Clin Transl Gastroenterol, 2016, 7: e152. doi: 10.1038/ctg.2016.11. |
| [15] | ZHAI H, FESLER A, BA Y, WU S, JU J. Inhibition of colorectal cancer stem cell survival and invasive potential by hsa-miR-140-5p mediated suppression of Smad2 and autophagy[J]. Oncotarget, 2015, 6: 19735–19746. DOI: 10.18632/oncotarget |
| [16] | SONG Y, XU Y, WANG Z, CHEN Y, YUE Z, GAO P, et al. MicroRNA-148b suppresses cell growth by targeting cholecystokinin-2 receptor in colorectal cancer[J]. Int J Cancer, 2012, 131: 1042–1051. DOI: 10.1002/ijc.v131.5 |
 2017, Vol. 38
2017, Vol. 38




