2. 第二军医大学海军医学系学员5队, 上海 200433;
3. 第二军医大学学员旅10队, 上海 200433
2. The 5thTeam of Student Brigade, Faculty of Navy Medicine, Second Military Medical University, Shanghai 200433, China;
3. The 10thTeam of Student Brigade, Second Military Medical University, Shanghai 200433, China
丙型肝炎病毒(hepatitis C virus, HCV)是引起人类肝病的重要病原体,超过70%的急性HCV感染者不能自然清除病毒而发展为慢性感染状态。慢性HCV感染是导致脂肪肝、肝硬化、肝癌的重要原因,并和糖尿病、淋巴瘤等疾病的发生密切相关[1]。目前尚没有可预防HCV感染的疫苗上市。干扰素联合利巴韦林曾是治疗丙型肝炎的标准方案,其疗效与所感染的HCV基因型有关,病毒清除率约为50%~70%[2-3]。近年来,一些新的针对HCV NS5A(如ledipasvir)、NS5B(如sofosbuvir)的直接抗病毒药物(direct-acting antiviral agent, DAA)陆续上市,对各个基因型HCV感染均有显著疗效[4-5]。这些药物价格极其昂贵,仅在欧美发达国家和少数发展中国家有售,其耐药性和远期不良反应尚待观察[5]。最近报道,共感染乙型肝炎病毒(hepatitis B virus, HBV)的丙型肝炎患者应用DAA治疗可能会激活HBV的复制[6]。因此,筛选、研发新的抗HCV药物仍十分必要。
我们前期研究发现HCV感染高度依赖于宿主分子硫氧还蛋白互作蛋白(thioredoxin-interacting protein, TXNIP),肝细胞内TXNIP的表达水平与HCV复制水平呈正相关[7]。已有研究发现,被临床广泛用于治疗高血压的钙通道阻断剂维拉帕米(verapamil)能抑制胰腺β细胞内TXNIP的表达[8-9]。本研究拟观察维拉帕米能否通过下调肝细胞内TXNIP的表达而抑制HCV感染,探讨其作为丙型肝炎治疗药物的可能性。
1 材料和方法 1.1 细胞、质粒与试剂人肝癌细胞系Huh7.5.1由中国科学院上海巴斯德研究所钟劲研究员馈赠;用于制备细胞培养产生的HCV (cell culture derived HCV,HCVcc)的J6/JFH-1嵌合全长基因组转录质粒由美国洛克菲勒大学Charles Rice教授馈赠;人胚肾HEK 293T细胞由本实验室保存;TXNIP真核表达质粒pLenti-TXNIP和TXNIP启动子报告基因表达质粒pTXNIP-EGFP由本实验室构建,pLenti-TXNIP由人巨细胞病毒(CMV)启动子调控TXNIP基因表达,pTXNIP-EGFP由人TXNIP启动子调控增强型绿色荧光蛋白(EGFP)表达;TXNIP siRNA序列为:5′-GUC UCU AGG GUU AAG CCA A(dTdT)-3′,非靶向宿主分子的对照siRNA(NC siRNA)序列为5′-UUG GCU UAA CCC UAG AGA C(dTdT)-3′,由韩国Bioneer公司合成。
维拉帕米干粉购自美国Selleck公司;转染试剂Lipofectamine 3000、辣根过氧化物酶(HRP)标记的羊抗兔或羊抗小鼠IgG、Alexa Fluor® 488标记的羊抗人IgG购自美国Invitrogen公司;GAPDH兔单克隆抗体购自美国Cell Signaling Technology公司;TXNIP小鼠单抗、HCV核心蛋白单抗购自英国Abcam公司;RNA抽提试剂RNAiso Plus、PrimeScriptTM RT Master Mix (Perfect Real Time)试剂盒、SYBR Premix Ex Taq试剂盒购自日本TaKaRa公司;CCK-8试剂盒购自上海碧云天生物技术有限公司;HCV感染者血清由第二军医大学长海医院检验科提供。
1.2 Huh7.5.1细胞的siRNA转染和HCVcc感染Huh7.5.1细胞用含有1%青霉素-链霉素、10%胎牛血清、2 mmol/L非必需氨基酸的DMEM完全培养液于37 ℃、5% CO2细胞培养箱中培养。将Huh7.5.1细胞接种于24孔板,每孔接种2×104个细胞,用Lipofectamine 3000转染TXNIP siRNA以及NC siRNA,每孔40 pmol siRNA,8 h后更换新的DMEM完全培养液,48 h后提取细胞总蛋白及总RNA检测TXNIP的表达。
J6/JFH-1嵌合HCVcc的制备方法参见文献[10],将感染滴度调整为105 FFU/mL感染Huh7.5.1细胞。Huh7.5.1细胞转染siRNA 48 h后用HCVcc感染,感染复数(MOI)为1。HCVcc感染8 h后弃去培养液,PBS洗3次,加DMEM完全培养液继续培养48 h后进行相关检测。
1.3 Huh7.5.1细胞过表达TXNIP用HEK 293T细胞包装含TXNIP基因的慢病毒,收集慢病毒上清感染接种于24孔板中的Huh7.5.1细胞(感染时细胞汇合度为40%)。感染慢病毒上清3 d后,进行相关实验。
1.4 维拉帕米处理维拉帕米干粉直接溶解于适量DMEM培养液至浓度为20 mmol/L。将Huh7.5.1细胞接种于24孔板,6 h后加入维拉帕米溶液至不同浓度梯度,用DMEM培养液补充各孔至等体积。对照孔(Mock组)加等体积DMEM培养液。各浓度维拉帕米处理细胞48 h后用CCK-8试剂盒检测Huh7.5.1细胞增殖情况。
1.5 qPCR分析用RNAiso Plus试剂抽提细胞总RNA,RNA沉淀溶解于DEPC处理水,取适量行琼脂糖凝胶电泳检查RNA的完整性,用多功能酶标仪测定RNA浓度和纯度。然后用PrimeScriptTM RT Master Mix试剂反转录获得cDNA,随后用SYBR Premix Ex Taq试剂对cDNA行qPCR。qPCR引物序列见表 1。
|
|
表 1 qPCR引物序列 Tab 1 Primer sequences of qPCR |
1.6 蛋白质印迹法分析与免疫荧光检测
用RIPA裂解液裂解细胞,抽提细胞总蛋白,用蛋白质印迹法检测细胞中TXNIP、GAPDH和HCV核心蛋白的表达,一抗为目标蛋白特异性单抗,二抗为HRP标记的抗鼠或抗兔IgG,用化学发光法采集蛋白印迹结果。
Huh7.5.1细胞接种于96孔板,HCVcc感染细胞48 h后,用免疫荧光检测细胞中的HCV蛋白,一抗为HCV抗体阳性的丙肝患者血清,二抗为Alexa Fluor® 488标记的羊抗人IgG,用DAPI复染细胞核。用Bioteck细胞成像系统计数荧光阳性细胞。
1.7 统计学处理应用GraphPad Prism 5.0软件进行数据处理和统计学分析。实验数据以x±sx表示,两组间比较采用Student’s t检验,多组比较采用单因素方差分析(one-way ANOVA)。检验水准(α)为0.05。
2 结果 2.1 下调TXNIP表达抑制HCV对Huh7.5.1细胞的感染用TXNIP siRNA转染Huh7.5.1细胞,然后用HCVcc感染。蛋白质印迹法和qPCR结果显示,转染TXNIP siRNA 48 h后,TXNIP蛋白和mRNA表达较转染NC siRNA的细胞相比均下调(P < 0.05,图 1A~1C)。用免疫荧光检测HCV感染的阳性细胞(图 1D),TXNIP siRNA转染的Huh7.5.1细胞HCV阳性克隆数为NC siRNA转染的7.3%(P < 0.05),与TXNIP mRNA的降低幅度相似。

|
图 1 下调TXNIP表达对HCV感染的影响 Fig 1 Effect of down-regulating TXNIP expression on HCV infection A, B: Western blotting analysis of TXNIP protein expression; C: qPCR analysis of TXNIP mRNA expression; D: Immunofluorescence staining of HCV infection. TXNIP: Thioredoxin-interacting protein; HCV: Hepatitis C virus; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. *P < 0.05. n=3, x±sx. Original magnification: ×400 (D) |
2.2 维拉帕米可下调Huh7.5.1细胞中TXNIP的表达
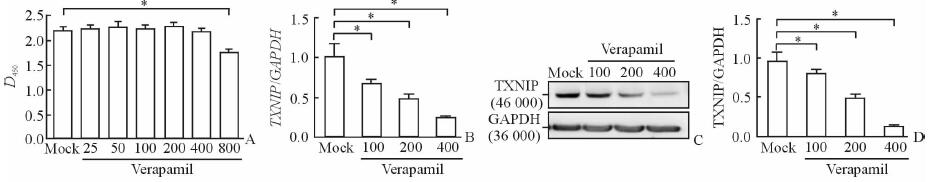
为了探讨维拉帕米能否通过下调肝细胞内TXNIP的表达从而抑制HCV感染,我们首先检测维拉帕米的细胞毒性,用25、50、100、200、400、800 μmol/L维拉帕米处理Huh7.5.1细胞48 h,随后用CCK-8试剂盒检测细胞增殖情况。结果显示,与Mock组相比,维拉帕米浓度≤400 μmol/L时,对Huh7.5.1细胞无明显毒性(图 2A)。因此,用100、200、400 μmol/L的维拉帕米处理Huh7.5.1细胞48 h,检测TXNIP的表达。qPCR结果显示,维拉帕米可明显下调细胞内TXNIP mRNA水平,且呈现浓度依赖性(P < 0.05,图 2B)。蛋白质印迹分析结果进一步证明维拉帕米能下调TXNIP的表达(P < 0.05,图 2C、2D)。
|
图 2 维拉帕米(μmol/L)下调Huh7.5.1细胞中TXNIP的表达 Fig 2 Verapamil (μmol/L) down-regulated TXNIP expression in Huh7.5.1 cells A: Proliferation ability of Huh7.5.1 cells treated with verapamil using CCK-8 assay; B: qPCR analysis of TXNIP mRNA expression; C, D: Western blotting analysis of TXNIP protein expression. TXNIP: Thioredoxin-interacting protein; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. *P < 0.05. n=3, x±sx |
2.3 维拉帕米能抑制HCV感染
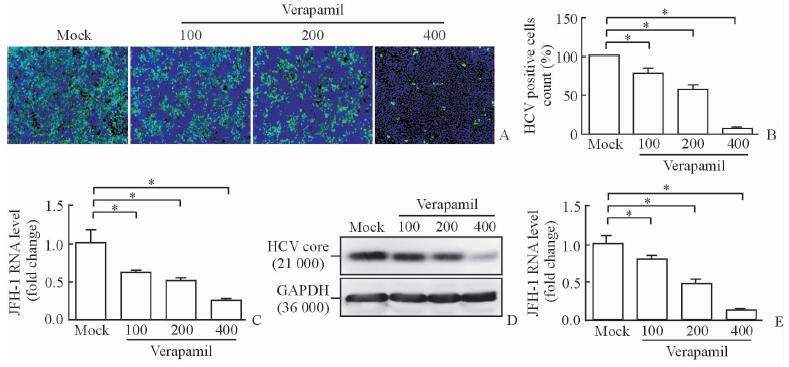
分别给予Huh7.5.1细胞100、200、400 μmol/L维拉帕米处理48 h后,用免疫荧光检测HCV感染的阳性细胞。结果显示,经各浓度维拉帕米处理后,HCV感染的阳性细胞数较Mock组均明显减少,特别是当浓度达到400 μmol/L时,HCV感染的细胞数降低至Mock组的0.85%(P < 0.05,图 3A、3B)。qPCR结果显示,细胞内的JFH-1 RNA水平随着维拉帕米浓度的增加而降低(P < 0.05,图 3C)。蛋白质印迹分析结果显示维拉帕米能抑制HCV核心蛋白的表达(图 3D)。qPCR检测细胞培养上清中的JFH-1 RNA,结果显示维拉帕米处理可明显降低HCVcc的产生(P < 0.05,图 3E)。
|
图 3 维拉帕米(μmol/L)抑制HCV感染Huh7.5.1细胞 Fig 3 Verapamil (μmol/L) inhibited HCV infection in Huh7.5.1 cells A: Immunofluorescence staining of HCV infection in Huh7.5.1 cells treated with different concentrations of verapamil; B: HCV positive cells count relative to Mock group; C: JFH-1 RNA levels relative to Mock group in Huh7.5.1 cells; D: Western blotting analysis of HCV core protein expression; E: JFH-1 RNA levels relative to Mock group in cell culture supernatants. HCV: Hepatitis C virus; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. Original magnification: ×400 (A). *P < 0.05. n=3, x±sx |
2.4 维拉帕米通过下调TXNIP表达抑制HCV感染
用TXNIP慢病毒感染Huh7.5.1细胞过表达TXNIP,随后用400 μmol/L维拉帕米处理、HCVcc(MOI=2) 感染,48 h后,行qPCR分析TXNIP mRNA和JFH-1 RNA。结果显示,维拉帕米处理对过表达TXNIP的Huh7.5.1细胞(Huh7.5.1-TXNIP)中TXNIP的mRNA表达无明显影响(图 4A),对HCV的感染也无明显影响(图 4B)。

|
图 4 过表达TXNIP对维拉帕米(400 μmol/L)抑制HCV感染的影响 Fig 4 Effect of TXNIP over-expression on verapamil (400 μmol/L) inhibiting HCV infection A: The expression of TXNIP mRNA; B: JFH-1 RNA level. TXNIP: Thioredoxin-interacting protein; HCV: Hepatitis C virus; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. *P < 0.05. n=3, x±sx |
2.5 维拉帕米能抑制TXNIP启动子的转录活性
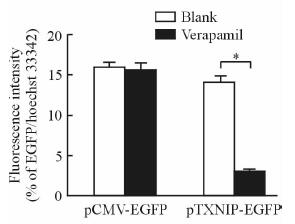
为了进一步明确维拉帕米是否抑制TXNIP启动子的转录活性,我们用含TXNIP启动子的EGFP报告基因表达质粒pTXNIP-EGFP和含人CMV启动子的EGFP报告基因表达质粒pCMV-EGFP分别转染Huh7.5.1细胞,转染8 h后加入维拉帕米,使其终浓度为400 μmol/L,继续培养细胞48 h,用多功能酶标仪检测细胞荧光强度。结果显示,维拉帕米能抑制TXNIP启动子调控的EGFP表达(P < 0.05),但对CMV启动子调控的EGFP表达无影响(图5)。
|
图 5 维拉帕米(400 μmol/L)抑制TXNIP启动子的转录活性 Fig 5 Verapamil (400 μmol/L) inhibited transcriptional activity of TXNIP promoter TXNIP: Thioredoxin-interacting protein; CMV: Cytomegalovirus; EGFP: Enhanced green fluorescent protein. *P < 0.05. n=3, x±sx |
3 讨论
HCV感染是全球范围的严重公共卫生问题,尽管近年来新一代DAA的上市使丙型肝炎的治疗取得巨大进展,但众多医药企业和科研机构仍致力于研发易于普及、疗效确切、不良反应小的抗HCV药物[1-4]。在抗病毒药物研发方面,老药新用近年来备受瞩目,如最近陆续有报道在寨卡病毒药物筛选方面取得成功[11-12]。从已上市的药物中筛选出的抗病毒药物,其安全性、不良反应、药代动力学等因素明确,且已规模化大批量生产,因此,老药新用能尽快得到有效、廉价的抗病毒药物,潜力巨大,与筛选研发新的抗病毒药物相比优势明显。
我们前期通过基因芯片和深度测序技术鉴定出HCV感染诱导肝细胞上调TXNIP的表达[7],该分子除在细胞中与硫氧还蛋白相互作用调节细胞氧化还原反应外[13-15],还对细胞自噬、凋亡、炎症小体激活、糖和脂类代谢等发挥重要调控作用[16-21],其促进HCV感染的作用机制有待进一步探讨。本研究中,用siRNA下调Huh7.5.1细胞中TXNIP的表达,然后用HCVcc感染,结果可见HCV感染水平随TXNIP表达水平的降低而明显下降,二者之间呈现相似的下调比例,提示HCV感染对TXNIP分子的高度依赖。
有研究发现TXNIP的表达上调可导致胰腺β细胞凋亡,与糖尿病发生密切相关,而钙通道阻滞剂维拉帕米能抑制TXNIP的表达从而促进β细胞存活,在小鼠模型中能防止链脲佐菌素诱导的糖尿病发生[8]。为探讨维拉帕米这种临床广泛用于治疗高血压的药物能否用于治疗丙型肝炎,我们以Huh7.5.1细胞为模型观察了维拉帕米的抗HCV活性。结果显示,在未产生细胞毒性的浓度范围内,维拉帕米可浓度依赖性下调TXNIP的表达和抑制HCV感染。用TXNIP慢病毒感染Huh7.5.1细胞,使其过表达TXNIP,维拉帕米对TXNIP表达和HCV感染的抑制作用不再呈现,因慢病毒载体中TXNIP启动子是人CMV启动子,维拉帕米对该启动子的转录无抑制作用。用启动子-EGFP报告基因质粒进行检测进一步证实维拉帕米是通过抑制TXNIP启动子的转录活性而下调该基因的表达。
大量报道显示,TXNIP的高表达与2型糖尿病、脂肪肝的发生高度相关[8, 17-18],而HCV感染常引发这两种疾病[1],因此,维拉帕米作为一种潜在的抗HCV药物,不仅能通过下调TXNIP的表达抑制HCV感染,还可能有助于延缓HCV感染引发的代谢性疾病。当然,对于血压正常的丙型肝炎患者,用维拉帕米治疗是否会引起低血压,需引起重视。
本研究结果显示维拉帕米可作为一种潜在的抗HCV药物。参考目前丙型肝炎治疗的联合用药方案,我们下一步拟观察维拉帕米与利巴韦林这两种低价的临床常用药物联合应用是否具有协同作用,进一步探讨维拉帕米应用于丙型肝炎临床治疗的价值。
| [1] | ROSEN H R. Clinical practice. Chronic hepatitis C infection[J]. New Engl J Med, 2011, 364: 2429–2438. DOI: 10.1056/NEJMcp1006613 |
| [2] | LAU J Y, TAM R C, LIANG T J, HONG Z. Mechanism of action of ribavirin in the combination treatment of chronic HCV infection[J]. Hepatology, 2002, 35: 1002–1009. DOI: 10.1053/jhep.2002.32672 |
| [3] | DAVIS G L, ESTEBAN-MUR R, RUSTGI V, HOEFS J, GORDON S C, TREPO C, et al. Interferon alfa-2b alone or in combination with ribavirin for the treatment of relapse of chronic hepatitis C. International Hepatitis Interventional Therapy Group[J]. N Engl J Med, 1998, 339: 1493–1499. DOI: 10.1056/NEJM199811193392102 |
| [4] | CHARLTON M, EVERSON G T, FLAMM S L, KUMAR P, LANDIS C, BROWN R S Jr, et al. Ledipasvir and sofosbuvir plus ribavirin for treatment of HCV infection in patients with advanced liver disease[J]. Gastroenterology, 2015, 149: 649–659. DOI: 10.1053/j.gastro.2015.05.010 |
| [5] | NKUIZE M, SERSTÉ T, BUSET M, MULKAY J P. Combination ledipasvir-sofosbuvir for the treatment of chronic hepatitis C virus infection:a review and clinical perspective[J]. Ther Clin Risk Manag, 2016, 12: 861–872. |
| [6] | KAWAGISHI N, SUDA G, ONOZAWA M, KIMURA M, MAEHARA O, ITO J, et al. Hepatitis B virus reactivation during hepatitis C direct-acting antiviral therapy in patients with previous HBV infection[J/OL]. J Hepatol, 2017. doi:10.1016/j.jhep.2017.04.008.[Epub ahead of print] |
| [7] | 唐紫薇, 王世杰, 王文博, 赵平, 戚中田. 宿主细胞硫氧还蛋白互作蛋白与丙型肝炎病毒感染的相互作用研究[J]. 中华传染病杂志, 2016, 34: 597–602. DOI: 10.3760/cma.j.issn.1000-6680.2016.10.006 |
| [8] | XU G, CHEN J, JING G, SHALEV A. Preventing β-cell loss and diabetes with calcium channel blockers[J]. Diabetes, 2012, 61: 848–856. DOI: 10.2337/db11-0955 |
| [9] | LEDWITCH K V, BARNES R W, ROBERTS A G. Unravelling the complex drug-drug interactions of the cardiovascular drugs, verapamil and digoxin, with P-glycoprotein[J/OL]. Biosci Rep, 2016, 36(2). pii:e00309. doi:10.1042/BSR20150317. |
| [10] | GUAN M, WANG W, LIU X, TONG Y, LIU Y, REN H, et al. Three different functional microdomains in the hepatitis C virus hypervariable region 1(HVR1) mediate entry and immune evasion[J]. J Biol Chem, 2012, 287: 35631–35645. DOI: 10.1074/jbc.M112.382341 |
| [11] | SACRAMENTO C Q, DE MELO G R, DE FREITAS C S, ROCHA N, HOELZ L V, MIRANDA M, et al. The clinically approved antiviral drug sofosbuvir inhibits Zika virus replication[J/OL]. Sci Rep, 2017, 7:40920. doi:10.1038/srep40920. |
| [12] | ZHOU H, WANG F, WANG H, CHEN C, ZHANG T, HAN X, et al. The conformational changes of Zika virus methyltransferase upon converting SAM to SAH[J]. Oncotarget, 2017, 8: 14830–14834. |
| [13] | HWANG J, SUH H W, JEON Y H, HWANG E, NGUYEN L T, YEOM J, et al. The structural basis for the negative regulation of thioredoxin by thioredoxin-interacting protein[J/OL]. Nat Commun, 2014, 5:2958. doi:. 10.1038/ncomms3958. |
| [14] | HANSCHMANN E M, GODOY J R, BERNDT C, HUDEMANN C, LILLIG C H. Thioredoxins, glutaredoxins, and peroxiredoxins-molecular mechanisms and health significance:from cofactors to antioxidants to redox signaling[J]. Antioxid Redox Signal, 2013, 19: 1539–1605. DOI: 10.1089/ars.2012.4599 |
| [15] | LEE S, KIM S M, LEE R T. Thioredoxin and thioredoxin target proteins:from molecular mechanisms to functional significance[J]. Antioxid Redox Signal, 2013, 18: 1165–1207. DOI: 10.1089/ars.2011.4322 |
| [16] | BOMPADA P, ATAC D, LUAN C, ANDERSSON R, OMELLA J D, LAAKSO E O, et al. Histone acetylation of glucose-induced thioredoxin-interacting protein gene expression in pancreatic islets[J]. Int J Biochem Cell Biol, 2016, 81(Pt A): 82–91. |
| [17] | MANDALA A, DAS N, BHATTACHARJEE S, MUKHERJEE B, MUKHOPADHYAY S, ROY S S. Thioredoxin interacting protein mediates lipid-induced impairment of glucose uptake in skeletal muscle[J]. Biochem Biophys Res Commun, 2016, 479: 933–939. DOI: 10.1016/j.bbrc.2016.09.168 |
| [18] | SINGH L P. Thioredoxin interacting protein (TXNIP) and pathogenesis of diabetic retinopathy[J/OL]. J Clin Exp Ophthalmol, 2013, 4. doi:10.4172/2155-9570.1000287. |
| [19] | FORRED B J, NEUHARTH S, KIM D I, AMOLINS M W, MOTAMEDCHABOKI K, ROUX K J, et al. Identification of redox and glucose-dependent Txnip protein interactions[J/OL]. Oxid Med Cell Longev, 2016, 2016:5829063. doi:10.1155/2016/5829063. |
| [20] | DOTIMAS J R, LEE A W, SCHMIDER A B, CARROLL S H, SHAH A, BILEN J, et al. Diabetes regulates fructose absorption through thioredoxin-interacting protein[J/OL]. Elife, 2016, 5. pii:e18313. doi:10.7554/eLife.18313. |
| [21] | MAHMOOD D F, ABDERRAZAK A, EL HADRI K, SIMMET T, ROUIS M. The thioredoxin system as a therapeutic target in human health and disease[J]. Antioxid Redox Signal, 2013, 19: 1266–1303. DOI: 10.1089/ars.2012.4757 |
 2017, Vol. 38
2017, Vol. 38



