2. 第二军医大学长海医院检验科, 上海 200433
2. Department of Clinical Laboratory, Changhai Hospital, Second Military Medical University, Shanghai 200433, China
肝癌是最常见的恶性肿瘤之一,患病率居恶性肿瘤前5位[1]。早期肝癌患者常无特异性症状而使临床诊断较为困难,中晚期肝癌患者在确诊时常已失去了最佳治疗时机。因此,寻找诊断肝癌转移和病情监测的生物学标志物能为探索肝癌发生、发展、转移、复发等机制提供研究思路,为肝癌的诊断、治疗、预后诊治提供更多依据。
长链非编码RNA(long non-coding RNA,lncRNA)是一类长度大于200 nt的非编码RNA,起初被认为没有意义,是基因组转录的噪音,但近年来被发现在人类基因组的表观遗传调控方面具有十分重要的作用,如调节DNA甲基化、组蛋白修饰和染色质重塑[2-3]。通过表观遗传学、转录和转录后调控,现已发现多种特异性lncRNA可以作为疾病诊断和治疗的标志物[4-6]。且lncRNA已被证明具有致癌或抑癌活性[7],最近研究显示肿瘤相关lncRNA在循环血中能被稳定检测,循环lncRNA是潜在的无创肿瘤标志物[8-9]。
LncRNA尿路上皮癌相关1(urothelial carcinoma-associated 1,UCA1) 最初是在膀胱癌中发现的lncRNA癌基因,参与调控胚胎发育,膀胱癌侵袭、进展和耐药[10]。研究发现UCA1可调节HBx蛋白的表达从而调控肝癌进展[11]。因此,本研究通过检测UCA1在肝癌肝内转移患者肝癌组织和血浆中的表达,分析其表达水平与临床病理因素的相关性,为寻找肝癌肝内转移患者的临床特征提供参考。
1 资料和方法 1.1 研究对象收集2015年1月至2016年1月第二军医大学长海医院确诊为肝癌肝内转移并手术切除的20例患者的肿瘤组织和术前、术后血浆样本,20例肝癌患者的肿瘤组织和血浆样本,以及20例健康受试者的血浆样本。所有肝癌及肝癌肝内转移患者均通过影像学检查及手术病理结果共同诊断确诊。所有患者均未接受过放化疗、免疫治疗、介入治疗及肝移植治疗,所有患者均不合并其他肿瘤及肝癌远处转移。所有患者和参与研究的健康受试者均签署知情同意书。本研究符合上海市医学会标准,经长海医院伦理委员会批准。
1.2 qPCR检测肿瘤组织和血浆样本中UCA1 mRNA的表达所有肿瘤组织经手术切除后于10 min内快速置入液氮冷冻,-80 ℃冰箱保存。肿瘤组织和血浆总RNA分别采用TRIzol试剂盒(美国Invitrogen公司)和mirVanaTM PARIS试剂盒(美国Ambion公司)提取。反转录获得cDNA。以cDNA为模板、GAPDH为内参照基因,采用SYBR PrimeScriptTM RT-PCR试剂盒[宝生物工程(大连)有限公司]进行qPCR反应检测UCA1 mRNA的表达。引物设计:UCA1正向5′-CTC TCC TAT CTC CCT TCA CTG A-3′,反向5′-CTT TGG GTT GAG GTT CCT GT-3′;GAPDH正向5′-GGT GGT CTC CTC TGA CTT CAA-3′,反向5′-GTT GCT GTA GCC AAA TTC GTT GT-3′。qPCR反应条件:95 ℃ 5 s,60 ℃ 31 s;循环40次。每个样本重复3次。UCA1 mRNA表达量采用2-ΔΔCt公式计算。
1.3 统计学处理应用SPSS 20.0软件进行数据分析,用GraphPad Prism 6.0软件绘图。计量资料用x±s表示,多组间比较采用方差分析,两组间比较采用t检验;计数资料用百分比表示,组间比较采用χ2检验或Fisher精确概率法。检验水准(α)为0.05。
2 结果 2.1 UCA1 mRNA在肝癌肝内转移患者血浆中高表达检测血浆中UCA1 mRNA的表达,结果显示肝癌肝内转移患者血浆中UCA1 mRNA的表达(0.037±0.008) 高于健康受试者(0.011±0.002) 和肝癌患者(0.020±0.006),差异有统计学意义(P<0.01)。
2.2 肝癌肝内转移患者术后血浆UCA1 mRNA表达下调与手术前相比,肝癌肝内转移患者术后血浆中UCA1 mRNA表达下降,差异有统计学意义(0.037±0.008 vs 0.015±0.005,P<0.05)。
2.3 肝癌和肝癌肝内转移患者肿瘤组织UCA1 mRNA的表达结果显示肝癌肝内转移患者原发灶肿瘤组织中UCA1 mRNA的表达高于肝癌患者(6.85±1.27 vs 4.85±1.46, P<0.01)。
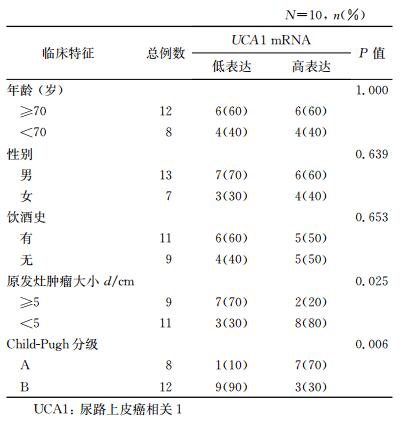
2.4 肝癌肝内转移患者原发灶肿瘤组织中UCA1 mRNA表达与临床病理特征的关系以UCA1 mRNA的中位表达水平(0.036) 为界,将20例肝癌肝内转移患者分为高表达组(n=10) 和低表达组(n=10)。肝癌肝内转移患者原发灶肿瘤组织中UCA1 mRNA的表达与原发灶肿瘤大小(P=0.025) 和Child-Pugh分级(P=0.006) 有关,与患者年龄、性别、饮酒史均无相关性,见表 1。
|
|
表 1 肝癌肝内转移患者原发灶肿瘤组织中UCA1 mRNA表达与临床病理特征的关系 |
3 讨论
研究发现lncRNA与多种肿瘤的发生、发展和转移密切相关,其在肿瘤中的重要作用日益受到关注,通过lncRNA序列寻找基因组的表观遗传调控的方式是一个新兴模式,将为多种疾病的发展提供新的解释,同时为寻找到疾病的发病机制增加了可能性,研究者也试图寻找特异性lncRNA作为疾病诊断和治疗的标志物。与其他疾病相比,迄今寻找到的与肝癌相关的lncRNA较多,但是这些lncRNA在疾病中的作用仍不十分明了,而能在人体内检测到的可能与肝癌转移密切相关的lncRNA更是鲜有报道。转移是影响肝癌及其他肿瘤患者预后的关键因素,而与肿瘤转移相关的lncRNA有稳定的特性,使其能够在组织及血液中被检测出来[9, 12],这为我们探寻肝癌转移过程中的分子机制提供了新的思路。
由于肝癌的发展过程伴随着多种肝脏代谢紊乱,因此也为评估这些基因相关的功能带来了巨大的挑战,探寻与肝癌及肝癌转移相关的lncRNA并确定其在疾病发展中的病理生理机制具有重大意义。随着肿瘤研究的深入,许多与肿瘤发生相关的病因也逐渐为人们所认知,但转移是肿瘤治疗过程中的瓶颈,一直未见突破性进展,许多肿瘤转移在晚期才被发现,也因此错过了最好的治疗时机。本研究将肝癌肝内转移的患者作为研究对象,以lncRNA UCA1作为突破口,研究UCA1 mRNA的表达情况及其表达水平与肝癌肝内转移患者病理特征的关系,以明确UCA1作为检测肝癌肝内转移与发展的诊断标志物的可能性,希望为早日阐明肝癌的发展和转移机制提供思路。
UCA1基因全长1 442 bp,有加尾信号ATTAAA和poly A尾,定位于19号染色体短臂的1区3带1亚带,含3个外显子和2个内含子,UCA1基因可能存在5个开放阅读框,靠近5′端,均小于300 nt[13]。有研究发现,UCA1表达可促进癌细胞增殖、侵袭、转移和抑制细胞凋亡[14],且在膀胱癌、结直肠癌、黑素瘤和食管癌等恶性肿瘤组织中表达上调,特别是在分期较晚的肿瘤中[15-16]。然而,它们在肝癌肝内转移过程中的作用仍知之甚少。本研究检测了肝癌肝内转移患者、肝癌患者及健康受试者血浆UCA1 mRNA表达,结果显示肝癌肝内转移患者血浆中UCA1 mRNA表达高于肝癌患者及健康受试者,且术后表达低于术前;此外,肝癌肝内转移患者原发灶肿瘤组织中UCA1 mRNA表达高于肝癌患者,提示肿瘤组织及血浆中UCA1 mRNA的表达均与肝癌发展存在密切关系。通过分析肝癌肝内转移患者原发灶UCA1 mRNA的表达与临床病理特征之间的关系,发现UCA1 mRNA在肝癌肝内转移患者原发灶肿瘤组织中的表达水平与原发灶肿瘤大小和Child-Pugh分级有关,而与年龄、性别、饮酒史无关,提示UCA1在肝癌的发展、侵袭、转移等方面可能具有重要作用,UCA1可能是肝癌转移和病情监测的诊断学标志物。低氧诱导因子1α(HIF-1α)和C/EBPα可调控UCA1表达,它们往往在肿瘤细胞中表达上调[17],这可能是导致肿瘤组织中UCA1高表达的原因之一。UCA1高表达可阻止BRG1结合到靶启动子上,抑制p21和p27的表达,进而促进肿瘤细胞生长、转移、侵袭和抑制细胞凋亡[18]。有研究发现UCA1能通过促进STAT3激活和抑制miR-143表达上调己糖激酶2转录,增强肿瘤细胞糖异生,从而在肿瘤发展过程中发挥作用[19]。
综上所述,UCA1 mRNA表达升高可能在肝癌发展、侵袭、转移等方面发挥了作用,并且其表达水平与肝癌发展的多个临床因素存在一定的关系。在临床实践中,外周血检测具有创伤小、可重复等优点,而且循环血lncRNA能被稳定地检测出来,因此循环UCA1有望成为诊断肝癌转移及监测病情发展的重要标志物。但因本研究样本量较少,血浆UCA1能否用于诊断和病情监测仍需大规模样本的验证。
| [1] | 陈东风, 孙文静, 刘凯军, 文良志. 非酒精性脂肪性肝病相关性肝癌的流行病学现状与发病机制[J]. 中华肝脏病杂志, 2017, 25: 111–114. DOI: 10.3760/cma.j.issn.1007-3418.2017.02.006 |
| [2] | LI W, ZHENG J, DENG J, YOU Y, WU H, LI N, et al. Increased levels of the long intergenic non-protein coding RNA POU3F3 promote DNA methylation in esophageal squamous cell carcinoma cells[J]. Gastroenterology, 2014, 146: 1714–1726. DOI: 10.1053/j.gastro.2014.03.002 |
| [3] | LING H, SPIZZO R, ATLASI Y, NICOLOSO M, SHIMIZU M, REDIS R S, et al. CCAT2, a novel noncoding RNA mapping to 8q24, underlies metastatic progression and chromosomal instability in colon cancer[J]. Genome Res, 2013, 23: 1446–1461. DOI: 10.1101/gr.152942.112 |
| [4] | GUTTMAN M, DONAGHEY J, CAREY B W, GARBER M, GRENIER J K, MUNSON G, et al. LincRNAs act in the circuitry controlling pluripotency and differentiation[J]. Nature, 2011, 477: 295–300. DOI: 10.1038/nature10398 |
| [5] | XIANG J F, YIN Q F, CHEN T, ZHANG Y, ZHANG X O, WU Z, et al. Human colorectal cancer-specific CCAT1-L lncRNA regulates long-range chromatin interactions at the MYC locus[J]. Cell Res, 2014, 24: 513–531. DOI: 10.1038/cr.2014.35 |
| [6] | HUANG J L, ZHENG L, HU Y W, WANG Q. Characteristics of long non-coding RNA and its relation to hepatocellular carcinoma[J]. Carcinogenesis, 2014, 35: 507–514. DOI: 10.1093/carcin/bgt405 |
| [7] | PRENSNER J R, CHINNAIYAN A M. The emergence of lncRNAs in cancer biology[J]. Cancer Discov, 2011, 1: 391–407. DOI: 10.1158/2159-8290.CD-11-0209 |
| [8] | TONG Y S, WANG X W, ZHOU X L, LIU Z H, YANG T X, SHI W H, et al. Identification of the long non-coding RNA POU3F3 in plasma as a novel biomarker for diagnosis of esophageal squamous cell carcinoma[J]. Mol Cancer, 2015, 14: 3. |
| [9] | REN S, WANG F, SHEN J, SUN Y, XU W, LU J, et al. Long non-coding RNA metastasis associated in lung adenocarcinoma transcript 1 derived miniRNA as a novel plasma-based biomarker for diagnosing prostate cancer[J]. Eur J Cancer, 2013, 49: 2949–2959. DOI: 10.1016/j.ejca.2013.04.026 |
| [10] | YANG C, LI X, WANG Y, ZHAO L, CHEN W. Long non-coding RNA UCA1 regulated cell cycle distribution via CREB through PI3-K dependent pathway in bladder carcinoma cells[J]. Gene, 2012, 496: 8–16. DOI: 10.1016/j.gene.2012.01.012 |
| [11] | HU J J, SONG W, ZHANG S D, SHEN X H, QIU X M, WU H Z, et al. HBx-upregulated lncRNA UCA1 promotes cell growth and tumorigenesis by recruiting EZH2 and repressing p27Kip1/CDK2 signaling[J]. Sci Rep, 2016, 6: 23521. DOI: 10.1038/srep23521 |
| [12] | WANG F, LI X, XIE X, ZHAO L, CHEN W. UCA1, a non-protein-coding RNA up-regulated in bladder carcinoma and embryo, influencing cell growth and promoting invasion[J]. FEBS Lett, 2008, 582: 1919–1927. DOI: 10.1016/j.febslet.2008.05.012 |
| [13] | 谢小娟, 李旭, 王帆, 陈葳. 非编码RNA UCA1基因的细胞定位和组织表达谱分析[J]. 南方医科大学学报, 2010, 30: 57–60,69. |
| [14] | WANG X, GONG Y, JIN B, WU C, YANG J, WANG L, et al. Long non-coding RNA urothelial carcinoma associated 1 induces cell replication by inhibiting BRG1 in 5637 cells[J]. Oncol Rep, 2014, 32: 1281–1290. |
| [15] | LI J Y, MA X, ZHANG C B. Overexpression of long non-coding RNA UCA1 predicts a poor prognosis in patients with esophageal squamous cell carcinoma[J]. Int J Clin Exp Pathol, 2014, 7: 7938–7944. |
| [16] | SRIVASTAVA A K, SINGH P K, RATH S K, DALEL D, GOEL M M, BHATT M L. Appraisal of diagnostic ability of UCA1 as a biomarker of carcinoma of the urinary bladder[J]. Tumour Biol, 2014, 35: 11435–11442. DOI: 10.1007/s13277-014-2474-z |
| [17] | LI X, WU Y, LIU A, TANG X. Long non-coding RNA UCA1 enhances tamoxifen resistance in breast cancer cells through a miR-18a-HIF1α feedback regulatory loop[J]. Tumour Biol, 2016, 37: 14733–14743. DOI: 10.1007/s13277-016-5348-8 |
| [18] | XUE M, LI X, LI Z, CHEN W. Urothelial carcinoma associated 1 is a hypoxia-inducible factor-1α-targeted long noncoding RNA that enhances hypoxic bladder cancer cell proliferation, migration, and invasion[J]. Tumour Biol, 2014, 35: 6901–6912. DOI: 10.1007/s13277-014-1925-x |
| [19] | LI Z, LI X, WU S, CHEN W. Long non-coding RNA UCA1 promotes glycolysis by upregulating hexokinase 2 through the mTOR-STAT3/microRNA143 pathway[J]. Cancer Sci, 2014, 105: 951–955. DOI: 10.1111/cas.2014.105.issue-8 |
 2017, Vol. 38
2017, Vol. 38