乳腺癌是影响全世界妇女健康的主要恶性肿瘤,发病率位居女性恶性肿瘤之首。研究表明,诊断乳腺癌时的肿瘤分期是影响乳腺癌预后的重要因素;Ⅰ、Ⅱ、Ⅲ、Ⅳ期乳腺癌患者的5年生存率分别约98%、85%、60%和20%,其中Ⅰ期乳腺癌患者的10年无病生存率是进展期乳腺癌的9倍以上[1]。早期发现和确诊对乳腺癌患者的预后有决定性的影响[2]。目前,我国筛查发现的无症状患者逐渐增多,早期乳腺癌的检出率逐渐升高,保乳比例不断增加,患者的5年生存率也明显提高。但由于我国筛查体系不健全、民众预防意识不足、诊断延迟导致晚期病例比例偏高等情况,使我国乳腺癌患者的生存率与发达国家相比仍存在差距。因此寻找特异性和灵敏度高的肿瘤标志物对乳腺癌的诊断和治疗具有重要意义。
近年来乳腺癌在基因组学和转录组学水平上被研究并与临床紧密结合[3],而其蛋白质组学的相关特征少有报道。蛋白质作为生物功能的直接执行者,更多反映了肿瘤的微环境及肿瘤的特异性翻译后修饰,更适合作为肿瘤标志物[4]。血清蛋白质组学为寻找有效的临床肿瘤标志物提供了有效方法。本研究应用定量蛋白质组学(quantitative proteomics)串联质量标签(tandem mass tag, TMT)标记技术筛选了Ⅱ期乳腺癌患者血清中发生变化的关键差异蛋白,并进行了信息分析和初步实验验证,为后续研究乳腺癌发生、发展进程中的分子机制及寻找临床乳腺癌诊断和治疗的潜在靶点提供依据。
1 材料和方法 1.1 标本来源与采集血清标本取自2014年1月至2015年12月第二军医大学长海医院乳腺外科收治的乳腺癌患者及同期在体检中心行体格检查的健康女性。乳腺癌患者68例均为美国癌症联合委员会(AJCC)乳腺癌TNM Ⅱ期乳腺浸润性导管癌,年龄21~64岁,平均(40.4±2.6) 岁。健康女性62例,均自愿参与本研究,年龄31~67岁,平均(48.2±2.2) 岁。本研究经第二军医大学长海医院伦理委员会审查通过,患者采血前均签署知情同意书。所有研究对象在采血前均未接受过任何治疗。血清采集及保存:清晨采集空腹静脉血4 mL,4 ℃静置1 h,1 336×g离心5 min后分离血清,-80 ℃冰箱保存备用。所有血清标本在行质谱检测前避免反复冻融。
1.2 主要试剂与仪器BCA蛋白定量试剂盒、TMT试剂购于美国Thermo Fisher Scientific公司,十二烷基磺酸钠购于美国Amresco公司,乙腈购于美国J.T.Baker公司,胰酶购于美国Gibco公司,测序级胰蛋白酶购于美国Promega公司,HRP/TRITC/FITC标记二抗购于美国SouthernBiotech公司,0.22 μm滤膜与超滤管(截留相对分子质量为3 000) 购自美国Millipore公司。其余试剂至少为国产分析纯级别。1.5 mL FRESCO 21型离心机购于美国Thermo Fisher Scientific公司,1.5、2 mL离心管均购于美国Axygen公司;Model 680型酶标仪购于美国Bio-Rad公司;PRO25型电动匀浆器购自美国Kimble Chase公司。
1.3 蛋白提取取每例Ⅱ期乳腺癌患者的血清各2 μL混合均匀,健康女性血清做相同处理;稀释后分别测定蛋白浓度。取96孔板,每孔加入标准品或待测样品10 μL,再加入工作液80 μL,振荡器混匀。37 ℃摇床中孵育30 min,冷却至室温后酶标仪检测570 nm波长处的光密度(D)值。绘制标准曲线并根据标准曲线计算待测样品的浓度。
取250 μg蛋白对应的血清量悬空滴于超滤管(截留相对分子质量为10 000) 中,加入200 μL尿素后13 000×g离心30 min。弃去上清液,膜上加200 μL尿素,13 000×g离心25 min。弃去上清液后加100 μL碘乙酰胺(尿素溶解),振荡摇匀,避光室温静置2 h,13 000×g离心25 min。加100 μL尿素,13 000×g离心25 min,重复1次;加100 μL NH4HCO3溶液,13 000×g离心25 min,重复1次。将超滤管移入新离心管,加胰酶(NH4HCO3溶液稀释),37 ℃摇床200 r/min酶解过夜,离心收集肽段。滤膜用60 μL 0.5 mol/L NaCl浸泡,离心,再次收集肽段,重复1~2次。加入0.1% CF3COOH酸化肽段,肽段脱盐,抽干并冷冻保存。
1.4 TMT标记使用100 mmol/L三乙胺硼烷溶解上述样品(pH≈8.5)。每管TMT试剂分别用41 μL的乙腈溶解,取TMT-126标记试剂加入健康女性血清肽段中、TMT-128标记试剂加入乳腺癌患者血清肽段中,混匀,摇床室温孵育4 h。加入8 μL的5%羟胺(hydroxylamine,HDX)终止反应,摇床室温孵育20 min。等量混合,酸化样品后脱盐、抽干。
1.5 LC-LTQ-MS/MS(液相色谱质谱联用)分析以及质谱数据检索取TMT标记的样品用10 μL 2%甲酸溶解,13 800×g离心5 min,上样进行LC-MS/MS分析。HPLC反向柱层析系统选择300 nL/min流速、5%~32%乙腈梯度洗脱分离样品。分离样品经LTQ Orbitrap XL质谱检测,从一级谱图中选取信号最强的3个母离子行高能碰撞诱导裂解(higher-energy collisional dissociation,HCD),所得数据使用Proteome Discoverer 1.4蛋白质组学软件进行检索分析。
1.6 生物信息学分析对从数据库检索得到的所有蛋白质进行分析,筛选表达变化在1.5倍以上(fold change大于1.5为上调,小于0.67为下调)的蛋白质为差异蛋白,在GeneCoDis3中对差异蛋白进行基因本体论(gene ontology,GO)注释分析和功能聚类分析,利用在线工具STRING (http://www.string-db.org)对差异蛋白进行蛋白相互作用分析。在UniProt数据库中检索筛选所得的差异蛋白质,并对其进行功能分类与分析。
1.7 蛋白质印迹法验证差异蛋白使用裂解液[50 mmol/L Tris (pH 7.4),150 mmol/L NaCl,1% Triton X-100,1%脱氧胆酸钠,0.1% SDS,1 mmol/L正钒酸钠,150 mmol/L氟化钠,0.5 mol/L EDTA,1 mmol/L亮抑蛋白酶肽]裂解样品,测定蛋白浓度后,取20~30 μg蛋白行SDS-PAGE,然后将蛋白转至聚偏二氟乙烯膜,5%脱脂奶粉封闭,加入一抗孵育,洗膜,再加入辣根过氧化物酶(HRP)标记的二抗孵育。采用化学发光法显示蛋白条带。
1.8 qPCR检测差异表达基因选取差异表达基因,在7300型定量PCR仪(ABI公司)上采用SYBR GreenⅠ法进行qPCR验证。反应体系(20 μL):SYBR Green Real-time PCR Master Mix 10 μL,ROX参比染料0.4 μL,cDNA (50 ng/μL) 1 μL,正、反向引物(10 μmol/L)各0.4 μL,ddH2O 7.8 μL。扩增条件:95 ℃预变性10 s;95 ℃变性15 s,60℃退火30 s,72 ℃延伸30 s,共40个循环。反应结束后由PCR扩增曲线得到样品的Ct值,采用ΔΔCt法计算目的基因转录水平的变化,并以β-actin作为内参。引物序列如下:RAF1上游5′-CCG AAC AAG CAA AGA ACA GTG-3′,下游5′-GAC GCA GCA TCA GTA TTC CAA T-3′;VIM上游5′-AGT CCA CTG AGT ACC GGA GAC-3′,下游5′-CAT TTC ACG CAT CTG GCG TTC-3′。
1.9 统计学处理应用GraphPad Prism 5软件进行数据分析,数据用x±s表示,组间比较采用t检验。检验水准(α)为0.05。
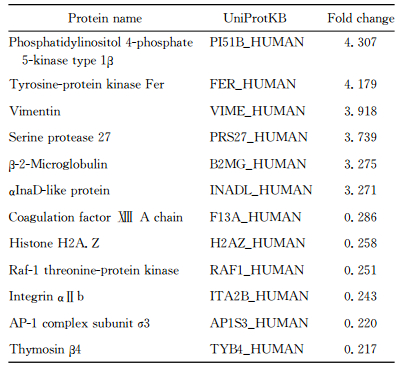
2 结果 2.1 乳腺癌患者的血清总蛋白及差异蛋白利用MaxQuant软件在IPI (International Protein Index)数据库中检索质谱鉴定所得的数据,结果在Ⅱ期乳腺癌患者中共定量得359种蛋白,其中筛选到26种蛋白表达上调,41种表达下调,部分差异蛋白见表 1。
|
|
表 1 Ⅱ期乳腺癌患者血清中的部分差异表达蛋白 Tab 1 Several differentially expressed proteins in patientswith stage Ⅱ breast cancer |
2.2 差异蛋白的GO功能注释与显著性富集分析
GO功能注释与显著性富集分析结果(图 1)显示,筛选得到的差异蛋白主要为血液凝固、细胞骨架结合蛋白,位于细胞外区、细胞质,且富集程度较高。

|
图 1 Ⅱ期乳腺癌患者血清中差异蛋白的显著性富集分析 Fig 1 Significant enrichment analysis of differentially expressed proteins in patients with stage Ⅱ breast cancer The vertical axis is the gene ontology (GO) category and the horizontal axis is the-lg (P value) of these GO terms. A: Biological process (BP); B: Molecular function (MF); C: Cellular component (CC); D: Signal pathway. GTP: Guanosine triphosphate; ECM: Extracellular matrix |
2.3 差异蛋白相互作用分析
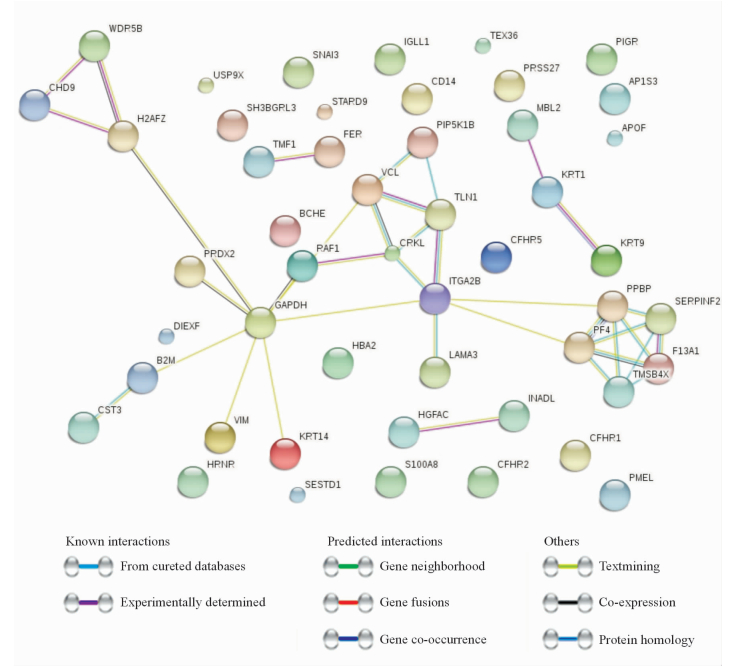
结果(图 2)显示,部分差异蛋白之间具有相互作用。F13A1、TMSB4X、SERPINF2、PPBP、PF4蛋白形成主要的相互作用网络,其功能聚类于分子结合方面。此外,VCL、GAPDH、RAF1蛋白处于相互作用网络中的关键位置,VIM基因也参与到复杂的调控网络中。
|
图 2 差异蛋白间相互作用分析 Fig 2 Interaction analysis of differentially expressed proteins |
2.4 蛋白质印迹法验证差异蛋白
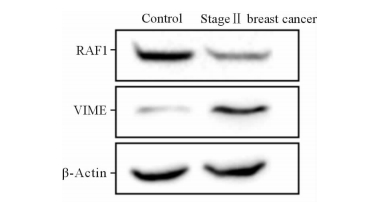
选取变化倍数显著的波形蛋白(VIME)和RAF1蛋白行蛋白质印迹分析,结果(图 3)显示RAF1在Ⅱ期乳腺癌患者血清中的表达水平低于健康对照女性,而VIME表达高于健康对照女性,与质谱鉴定结果一致。
|
图 3 蛋白质印迹法检测VIME和RAF1蛋白的表达 Fig 3 VIME and RAF1 expressions detected by Western blotting VIME: Vimentin; RAF1: Raf-1 threonine-protein kinase |
2.5 qPCR验证差异蛋白
qPCR结果(图 4)表明,VIM基因在Ⅱ期乳腺癌患者血清中的表达较健康对照女性上调(P<0.01),而RAF1基因表达较健康对照女性下调(P<0.01),与蛋白质印迹法验证结果及质谱鉴定结果一致。

|
图 4 qPCR检测VIM和RAF1 mRNA的表达水平 Fig 4 Expressions of VIM and RAF1 mRNA detected by qPCR VIM: Vimentin; RAF1: Raf-1 threonine-protein kinase. **P < 0.01 vs control. n=3, x±s |
3 讨论
基因组学揭示了人类遗传学图谱的基本特点,为人类认识基因和遗传物质带来了根本性的变革。然而基因组学并不能揭示所有基因的功能及其调控机制。因为绝大部分基因的功能都依赖于其编码的蛋白质,所以对细胞或者机体蛋白质进行研究有助于揭示细胞的代谢过程和机体生命活动的规律。定量蛋白质组学可以实现在一次实验中对成百上千种蛋白质的定量和分析,为规模化发现和验证生物标志物、发现新的治疗靶标提供了重要手段[5]。
VIME是中间丝状体家族的重要组成部分,属于支架和连接蛋白[6-7]。VIME在维持细胞结构、细胞质的完整性及细胞支架的相互连接中显示出不同作用,此外它还参与调控细胞迁移、增殖、信号转导,且在许多肿瘤中表达增高,但其在肿瘤进展中的机制尚未阐明[8]。本研究中VIME蛋白在乳腺癌患者血清中的表达明显上调(为健康对照女性的3.918倍),与研究显示VIME蛋白在乳腺癌肿瘤组织中表达升高[9]的结论一致。VIME蛋白上调是上皮间质转化(EMT)标志,而EMT与肿瘤细胞的分化、生长、浸润及转移相关[10-11]。此外,还有研究表明VIME蛋白的表达水平与乳腺癌细胞的迁移能力呈正相关[12]。
RAF1基因最初发现于小鼠肉瘤和禽的癌病毒中,其在导管上皮高表达并且在不同级别的乳腺肿瘤中呈不同程度的表达,对肝癌的发生、发展有抑制作用[13]。本研究中,RAF1蛋白在乳腺癌患者血清中的表达显著下调(为健康对照女性的0.251),而有研究表明RAF1蛋白在存在淋巴结转移的乳腺癌肿瘤组织中表达下调,表明其表达下调可能是乳腺癌淋巴结转移的生物学标志[14]。RAF1调控肿瘤的机制可能是阻断磷酸化作用和促有丝分裂蛋白激酶(MAPK)的激活[15-16],也可能是在肿瘤坏死因子(TNF)α和白介素1β的刺激下,通过阻断核因子kappa B((NF-κB)的激活来调节肿瘤的淋巴结转移[17]。然而RAF1并不是在所有肿瘤中均表达下调,也有报道称RAF1过表达与鳞状细胞癌和肺癌的进展相关[18]。本课题组前期使用免疫组化法研究发现RAF1在预后较差的基底样型乳腺癌中过表达,且RAF1表达增高对患者的腋窝淋巴结转移和总生存率具有较差的预后影响,分析其原因可能是RAF1对Ras/Raf/MEK/ERK经典信号通路具有激活作用[19]。RAF1在不同肿瘤中表达不同,甚至在不同类型的相同肿瘤中表达也不相同,其机制有待进一步探讨。RAF1蛋白是与肿瘤发生和转移相关的重要蛋白,在今后研究中应阐明其机制,为肿瘤发生、发展及抗癌药物敏感性预测提供潜在的生物学标志物。
本研究的蛋白质相互作用关联图显示,乳腺癌患者有几个生物学进程的基因呈现明显变化:(1) 血液凝固进程,在高严谨筛选条件下共有2种蛋白(PLF4、CXCL7) 上调为健康对照女性的1.92~2.08倍,7种蛋白(A2AP、ITA2B、VINC、FLNA、F13A、TLN1、TYB4) 下调为健康对照女性的0.22~0.66。其中PLF4可刺激DNA合成、有丝分裂、糖酵解、细胞内营积累,呈上调趋势;TYB4参与了细胞-基质黏附和细胞-细胞黏附,调节细胞表面钙黏蛋白的表达,并通过钙黏蛋白复合体增强传导作用,呈下调趋势。(2) 细胞连接装配进程,有2种蛋白(INAD、LAMA3) 上调为健康对照女性的2.47~3.27倍,2种蛋白(FLNA、K1C14) 下调为健康对照女性的0.25~0.43。其中FLNA在血管生成过程中具有调节细胞间通信、黏附和连接的作用,呈下调趋势。
生物信息学分析(蛋白质组学)可以从众多差异蛋白中筛选可能有意义的蛋白,而对这些蛋白进行验证是筛选目的蛋白的第一步。本研究根据差异蛋白鉴定分析结果显示VIME蛋白和RAF1蛋白的变化倍数较显著,且RAF1与其他蛋白关联较多,结合GO注释分析和功能聚类分析与文献检索示这2种蛋白与肿瘤相关,蛋白质印迹法和qPCR结果显示RAF1和VIME蛋白的表达与蛋白质组学质谱鉴定结果一致,验证了蛋白质组学结果,表明其在乳腺癌发生、发展中的作用值得研究。
综上所述,定量蛋白质组学技术能有效筛选出乳腺癌患者血清中的差异蛋白,对其相应的生物学分析有助于了解它们参与的乳腺癌发生、发展相关生物学过程和代谢通路,为乳腺癌的研究提供了新方法和研究思路。
| [1] | CHIARI D, ORSENIGO E, GUARNERI G, BAIOCCHI G L, MAZZA E, ALBARELLO L, et al. Effect of neoadjuvant chemotherapy on HER-2 expression in surgically treated gastric and oesophagogastric junction carcinoma:a multicentre Italian study[J]. Updates Surg, 2017, 69: 35–43. DOI: 10.1007/s13304-017-0423-2 |
| [2] | GONZÁLEZ-REYMÚNDEZ A, DE LOS CAMPOS G, GUTIÉRREZ L, LUNT S Y, VAZQUEZ A I. Prediction of years of life after diagnosis of breast cancer using omics and omic-by-treatment interactions[J/OL]. Eur J Hum Genet, 2017. doi:10.1038/ejhg.2017.12.[Epubaheadofprint] |
| [3] | CHEN Y, BRITTON D, WOOD E R, BRANTLEY S, MAGLIOCCO A, PIKE I, et al. Quantitative proteomics of breast tumors:tissue quality assessment to clinical biomarkers[J/OL]. Proteomics, 2017. doi:10.1002/pmic.201600335.[Epubaheadofprint] |
| [4] | JIANG Z, SHEN H, TANG B, CHEN H, YU Q, JI X, et al. Identification of diagnostic markers involved in the pathogenesis of gastric cancer through iTRAQ-based quantitative proteomics[J]. Data Brief, 2017, 11: 122–126. DOI: 10.1016/j.dib.2016.12.023 |
| [5] | SIMICEVIC J, DEPLANCKE B. Transcription factor proteomics-tools, applications, and challenges[J/OL]. Proteomics, 2017.doi:10.1002/pmic.201600317.[Epubaheadofprint] |
| [6] | JONES C, MACKAY A, GRIGORIADIS A, COSSU A, REIS-FILHO J S, FULFORD L, et al. Expression profiling of purified normal human luminal and myoepithelial breast cells:identification of novel prognostic markers for breast cancer[J]. Cancer Res, 2004, 64: 3037–3045. DOI: 10.1158/0008-5472.CAN-03-2028 |
| [7] | VAN DE RIJN M, PEROU C M, TIBSHIRANI R, HAAS P, KALLIONIEMI O, KONONEN J, et al. Expression of cytokeratins 17 and 5 identifies a group of breast carcinomas with poor clinical outcome[J]. Am J Pathol, 2002, 161: 1991–1996. DOI: 10.1016/S0002-9440(10)64476-8 |
| [8] | SATELLI A, LI S. Vimentin in cancer and its potential as a molecular target for cancer therapy[J]. Cell Mol Life Sci, 2011, 68: 3033–3046. DOI: 10.1007/s00018-011-0735-1 |
| [9] | DOWLING P, MELEADY P, DOWD A, HENRY M, GLYNN S, CLYNES M. Proteomic analysis of isolated membrane fractions from superinvasive cancer cells[J]. Biochim Biophys Acta, 2007, 177: 93–101. |
| [10] | JOHANN D J, RODRIGUEZ-CANALES J, MUKHERJEE S, PRIETO D A, HANSON J C, EMMERT-BUCK M, et al. Approaching solid tumor heterogeneity on a cellular basis by tissue proteomics using laser capture microdissection and biological mass spectrometry[J]. J Proteome Res, 2009, 8: 2310–2318. DOI: 10.1021/pr8009403 |
| [11] | ULIRSCH J, FAN C, KNAFL G, WU M J, COLEMAN B, PEROU C M, et al. Vimentin DNA methylation predicts survival in breast cancer[J]. Breast Cancer Res Treat, 2013, 137: 383–396. DOI: 10.1007/s10549-012-2353-5 |
| [12] | VUORILUOTO K, HAUGEN H, KIVILUOTO S, MPINDI J P, NEVO J, GJERDRUM C, et al. Vimentin regulates EMT induction by Slug and oncogenic H-Ras and migration by governing Axl expression in breast cancer[J]. Oncogene, 2011, 30: 1436–1448. DOI: 10.1038/onc.2010.509 |
| [13] | TAN W J, LAI J C, THIKE A A, LIM J C, TAN S Y, KOH V C, et al. Novel genetic aberrations in breast phyllodes tumours:comparison between prognostically distinct groups[J]. Breast Cancer Res Treat, 2014, 145: 635–645. DOI: 10.1007/s10549-014-2982-y |
| [14] | HAGAN S, AL-MULLA F, MALLON E, OIEN K, FERRIER R, GUSTERSON B, et al. Reduction of Raf-1 kinase inhibitor protein expression correlates with breast cancer metastasis[J]. Clin Cancer Res, 2005, 11: 7392–7397. DOI: 10.1158/1078-0432.CCR-05-0283 |
| [15] | REDDY K B, NABHA S M, ATANASKOVA N. Role of MAP kinase in tumor progression and invasion[J]. Cancer Metastasis Rev, 2003, 22: 395–403. DOI: 10.1023/A:1023781114568 |
| [16] | JERIC I, MAURER G, CAVALLO A L, RAGUZ J, DESIDERI E, TARKOWSKI B, et al. A cell-autonomous tumour suppressor role of RAF1 in hepatocarcinogenesis[J]. Nat Commun, 2016, 7: 13781. DOI: 10.1038/ncomms13781 |
| [17] | YEUNG K C, ROSE D W, DHILLON A S, YAROS D, GUSTAFSSON M, CHATTERJEE D, et al. Raf kinase inhibitor protein interacts with NF-kappaB-inducing kinase and TAK1 and inhibits NF-kappaB activation[J]. Mol Cell Biol, 2001, 21: 7207–7217. DOI: 10.1128/MCB.21.21.7207-7217.2001 |
| [18] | XU Z H, HANG J B, HU J A, GAO B L. RAF1-MEK1-ERK/AKT axis may confer NSCLC cell lines resistance to erlotinib[J]. Int J Clin Exp Pathol, 2013, 6: 1493–1504. |
| [19] | WU P, LI X, WU Y, HU W, WANG Y, GAO L, et al. Overexpression of Raf-1 in basal-like carcinoma of the breast:correlation with clinicopathology and prognosis[J]. Contemp Oncol (Pozn), 2014, 18: 391–395. |
 2017, Vol. 38
2017, Vol. 38