2. 第二军医大学长海医院病理科, 上海 200433
2. Department of Pathology, Changhai Hospital, Second Military Medical University, Shanghai 200433, China
根据2015年《中国癌症统计》的估算,我国癌症患者已高达429.2万,死亡癌症患者达281.4万[1]。然而,卫生服务需求与医疗卫生资源之间的矛盾使得传统医疗手段和看病模式不能满足社会发展需要。大数据和云计算支撑的人工智能(artificial intelligence)飞速发展,有望成为技术变革的核心[2-3]。目前,人工智能已经影响了医疗行业的工作模式,在新药研发、疾病诊断、中医理论、中药辨识和病理诊断等方面取得了卓越成绩。迄今,IBM公司的“沃森”是全球领先的医疗人工智能系统,它将人工智能的数据整合、分析与判断能力与医师的诊疗经验相结合,提供辅助医疗的处理逻辑[4]。
1 病理图像是人工智能的绝佳应用场景 1.1 深度学习的数据特征人工智能的核心是深度学习[5]。深度学习源于人工神经网络的研究,其通过组合低层特征形成更加抽象的高层表示属性类别或特征,以发现数据的分布式特征表示。深度学习颠覆了语音识别、图像分类、文本理解等众多领域的算法设计思路,渐渐形成了一种从训练数据出发,经过一个端到端(end-to-end)的模型,然后直接输出得到最终结果的一种新模式。深度学习中的每一层都可以为了最终任务来进行自我调整,从而实现各层之间的通力合作,大大提高任务的准确度。随着大数据时代的到来和图形处理器(graphics processing unit, GPU)等各种更强大的计算设备的发展,深度学习可以充分利用各种海量数据(标注数据、弱标注数据或仅仅数据本身),完全自动地学习抽象的知识表达,即将原始数据浓缩成某种知识[6]。只要有大规模结构化的数据作为训练集,人工智能就能识别数据中难以肉眼辨别和(或)难以用生物学统计学方法提取的特征。CT、MRI和病理图片等都属于结构化的数据,是人工智能学习的最佳材料。
1.2 病理医学的窘境和解决方案病理切片是二维医学图像,是一种典型的结构化数据,非常适合人工智能的深度学习[7-8]。我国病理医师数量少,且收入普遍偏低、工作环境恶劣,导致人才流失严重。病理人才的匮乏进一步加重了医师的负担和责任,增加了误诊和漏诊概率。目前,优秀的病理人才多集中于大城市的三甲医院,基层医院病理医师水平参差不齐。三甲医院病理医师工作、科研和教学业务繁忙,可接触更多病例,能力不断提升,但大部分为重复劳动;而基层病理医师较为空闲、接触病例少、能力提升缓慢。此外,三甲医院病理科每天产生上千张病理切片,尽管大部分可能没有阳性结果,但仍必须对每张切片进行仔细审阅,耗费大量精力。人工智能有助于改善目前的病理困境,其识别病理切片准确性可靠、速度快、没有地域限制、诊断标准一致、不会疲劳,可辅助病理医师筛查病理照片,使病理医师将更多精力用于研究疑难杂症、提升自身水平[3, 9-10]。
2 病理人工智能的学习过程实践 2.1 胃癌和胆管癌人工智能识别模型的构建流程准确性和特异性高的计算模型必须有大量的病理切片数据支持,依靠这些数据进行后续的深度学习。本团队在构建胃癌和胆管癌人工智能识别模型的实践中总结了深度学习的流程,主要分为以下5个阶段。
(1) 数据标注。算法团队与医师团队共同设计项目类别、病理切片数量、标注平台和标注方式等细节。医师团队收集病理切片,并数字化所有切片。算法团队开发标注工具,医师团队使用该工具对图像进行分类,标定肿瘤细胞和特殊结构(如神经、癌栓和淋巴细胞等)所在的位置,详细说明各种结构的特点,录入数据库。
(2) 数据预处理。算法团队将标注好的图片进行预处理,对图片进行反转、模糊化、调节曝光度等操作,让神经网络在训练中能更好地学习病理疾病呈现出的本质特征,去掉人为和环境的干扰,从而增强算法的鲁棒性。数据处理完毕后,重新将图片数据进行随机排列,并按照6:2:2的比例分为训练集、验证集和测试集;将前2个集合提供给算法团队进行数据训练,最后1个集合将交由医疗团队进行测试,以保证算法的效果真实性。
(3) 算法设计。神经网络的设计对学习病理疾病的特征极为重要,算法团队运用多个在图像识别分类上有效的基本模型来对系统进行基准训练。实验中根据文献设计一系列算法模型,将训练集合输入到各个神经网络中进行训练,跟踪算法的收敛速度和准确性,并使用验证集来内部验证算法的效果[11-13]。
(4) 算法训练。比较后确定2~3个最优基本模型,在基本模型的基础上进一步微调神经网络的结果和网络参数,提高算法的准确性。在取得阶段的训练成果后,医疗团队使用测试集对算法进行测试,比较没有训练过的图像能否被算法正确地识别。
(5) 算法测试。最后通过受试者工作特征(receiver operating characteristic, ROC)曲线客观评价算法的性能,其通过构建敏感性(特异性曲线)进行衡量,其中敏感性代表正确识别恶性病变的能力,特异性代表正确识别良性病变的能力。
2.2 精准标注是高质量人工智能的先决条件病理人工智能的核心是算法,但基础和必要条件是有效数据。病理人工智能如果源于不完整或有偏移的数据基础,最终决策会产生偏差甚至错误,进而导致人们失去对人工智能的信心。同其他领域的人工智能一样,简洁、完整和准确的数据是医学领域人工智能的必要前提。然而,与其他图像相比,病理图像还有自己独特的特点。第一是高质量图像的获取和合适的图片尺寸;与显微镜和数字切片扫描仪合作可以解决图像质量问题,而对于大尺寸病理切片的图片处理则需要根据算法来不断训练和调整。第二就是数据的标注置信度;病理人工智能算法精准与否完全取决于标注的可靠程度,但数字病理切片的标注十分烦琐,工作量大,而且特殊病理需要多个专家和特殊染色来审核和辅助标注;此外,病理诊断病种繁多,每个病种可能都需要一个模型,由于疾病的亚专科化,每位专家可能仅对部分病种特别熟悉,因此标注过程中每个病种都要有专门的专家团队以及标准化的流程[14]。
病理标注方式因算法而异,但原则一致,即准确、完整地标记出所有的肿瘤细胞。而这也是标注的难点,因为不同种类肿瘤细胞的表现形式多样,即使同一种肿瘤,其肿瘤细胞也繁杂多样[15]。本团队近年来参与了胃癌、胆管癌和乳腺癌的标注,总结标注原则如下。
2.2.1 标注团队的权威性病理标注团队必须有一定的医学背景和深厚的专业知识,团队成员包括2~4名具有医学背景的青年医师、1名固定的病理专家、由不同领域病理医师组成的专家团以及1位病理技术人员。青年医师负责标注,但在标注某种疾病之前必须经过专家团成员的培训和指导。固定的病理专家负责定期和最终审核,适时修正遗漏或错误的标注;如果还不能确定,咨询专家团中2位相关专家予以指导。如果需要特殊染色如免疫组化,则由病理技术人员予以支持。
2.2.2 标注工具的简洁化目前市场的图像软件如Photoshop、ImageJ、NDP.view均能够标记肿瘤细胞,但像素和方便程度均不理想。不同团队均自行研发了标注工具,有的简单、有的复杂,但最终都简洁化,如本团队自行研发的标注软件,标注类型主要为不连续性线型,线的起点与终点能自我连接,粗细和颜色也可以调节,并附带自定义功能,大大提升了标注速度和标注准确性。
2.2.3 标注序列的程序化确定行某个肿瘤的人工智能之后,团队成员先行讨论宏观和细节,宏观包括分析肿瘤细胞的特点并进行分类,确定每种类型需要多少病例、每个病例抽取多少张图片。以胃癌为例,胃癌有10余种类型,就要建立10余个序列,每个序列含有一种类型的上百甚至上千张病理切片,然后用统一的扫描工具数字化所有照片,利用相应的标注工具切取特定像素的图片,而后由标注人员标注,专家审核和补充。
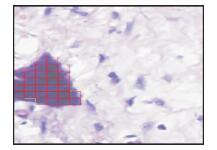
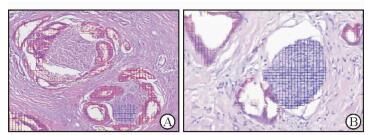
2.2.4 制片流程的标准化低质量的图片算法可能会被误认为是肿瘤,如图 1所示病理切片中的苏木精结晶被认为是肿瘤。伊红的染色时间过长或过短也会产生偏移,本团队在进行人工智能识别病理图像时发现,伊红染色过浓时病理图像中的神经可能不能完全识别(图 2A),而正常染色则能完全识别(图 2B)。伊红染色过浓会导致不同组织结构之间的反差减少,不利于分辨。解决办法一般有以下两种:(1)增加不同染色质量切片的样本量用于深度学习;(2)建立全自动的制片和染色设备和流程,提供标准化和数字化的病理图像。显然标准化制片流程的可行性更强。
|
图 1 病理切片中的杂质被人工智能识别为肿瘤 Fig 1 Impurities in pathological section were identified as tumors by artificial intelligence H-E staining. Original magnification: ×400 |
|
图 2 人工智能识别肿瘤中的神经侵犯 Fig 2 Neural invasion identified by artificial intelligence A: Artificial intelligence can not accurately identify the nerve in tumor due to intense staining; B: Artificial intelligence can identify the nerve. H-E staining. Original magnification: ×200 |
2.2.5 标注流程的规范性
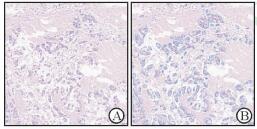
熟练的医师标注时使用方便的标注工具也要耗费大量时间,如对一个分化程度较低的腺癌的2 048×2 048像素、放大200倍的图片,可能需要上百个标记(图 3),而对一张印戒细胞癌的病理照片,则需要上千个标记。故为了防止返工,标注前必须建立规范化的标注流程:(1)明确标记方式,即专家截取图片后,根据算法确定标注方式(标记细胞核还是癌巢或腺体);(2)明确标记范围,即要确定标记线是否需紧贴腺体,存在空隙是否影响算法等;(3)明确标记组织,肿瘤组织成分较多,有些成分对于指导治疗和评估预后十分关键,要事先决定是否需要标记;(4)合理安排时间;(5)疑难病例及时汇报。
|
图 3 标注前(A)、标注后(B)低分化腺癌的临床病理切片 Fig 3 Clinical pathological sections of low-differentiated adenocarcinoma before (A) and after (B) labeling H-E staining. Original magnification: ×100 |
2.2.6 疑难病理的解决方案
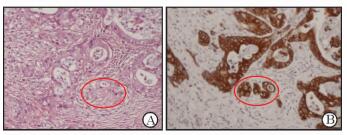
有些细胞很难确定是否为肿瘤细胞,尤其是侵袭到间质或淋巴结内的微小转移灶。免疫组化能准确显示侵袭到间质内的散在分布的癌细胞。本团队在标注过程中发现,肿瘤组织中一般有分化成熟的癌细胞和分化不成熟的癌细胞,肉眼难以分辨(图 4A)。行免疫组化染色后,所有癌细胞均得以显示,H-E染色中难以分辨的癌细胞也容易识别(图 4B)。此外,测序技术也可为区分肿瘤的分类提供更加精确的诊断。
|
图 4 免疫组化发现散在分布的肿瘤细胞 Fig 4 Scattered tumor cells identified by immunohistochemistry Various types of tumor cells (in red circle) in pathological images were hardly identified by H-E staining (A) and easily identified by immunohistochemistry (B). Original magnification: ×200 |
3 人工智能在肿瘤病理中的应用进展
病理人工智能已用于乳腺癌[16]、胃癌[17]、前列腺癌[18]和肠癌[19-20]等多种肿瘤中,应用范围集中于良恶性诊断、疾病分级、染色分析和早期肿瘤筛查等方面。
3.1 乳腺癌病理领域人工智能在乳腺癌的筛查、诊断前哨淋巴结是否有癌细胞和人表皮生长因子受体2(human epidermal growth factor receptor-2, HER2)染色评分等方面均取得了显著成绩。Zadeh Shirazi等[21]根据乳腺癌患者的乳腺肿块形状、边缘、密度、年龄和乳腺影像报告数据系统(breast imaging-reporting and data system, BI-RADS)评分,采用深度学习方法对822例乳腺癌患者进行了分析和验证,验证阶段疾病的检出率高达95%。Araújo等[22]运用卷积神经网络对乳腺癌的组织图像进行了分类,四分类(正常组织、良性疾病、原位癌和侵袭癌)的准确度为77.8%,二分类(癌和非癌组织)的准确度为83.3%,肿瘤检出的敏感度为95.6%。
乳腺癌的HER2检测和评估对于指导用药和评估预后意义明确。2016年在Nottingham举办的PathSoc大会上组织了基于人工智能的自动HER2评分比赛,结果证明人工智能的HER2评分优于病理专家[23]。另一项研究对71例乳腺癌切除标本进行了自动HER2评分,结果显示83%与病理医师诊断结果相符,但有12例结果不一致,重新分析后修正了8例的诊断。分析原因是HER2染色异质性造成医师对HER2表达的结果判断出现了偏差[24]。
3.2 胃癌病理领域胃癌分类和胃癌早期筛查是指导胃癌治疗和提高胃癌患者生存率的重要手段。Sharma等[25]采用多种深度学习方法对胃癌切片进行分类,准确性较高的算法对胃癌分类的准确度达0.699 0。Yoshida等[17]对3 062个胃镜样本进行了人工智能分析,该研究从三分类(癌或可疑癌、腺瘤或可疑肿瘤性病变和非肿瘤)和二分类(阴性或非阴性)角度比较了人工智能系统与病理专家之间的吻合度。三分类的总体吻合度为55.6% (1 702/3 062),阴性活组织检查标本中吻合度为90.6% (1 033/1 140),阳性活组织检查标本中吻合度 < 50%。而对于二分类,该系统的敏感度、特异度、阳性预测值和阴性预测值分别为89.5% (95%CI:87.5%~91.4%)、50.7% (95%CI:48.5%~52.9%)、47.7% (95%CI:45.4%~49.9%)和90.6% (95%CI:88.8%~92.2%)。在上海胃癌人工智能比赛中,截至2017年11月4日,人工智能对于胃癌诊断的准确度高达89.7%以上(http://www.datadreams.org/)。
4 展望计算机辅助算法在病理分析中的广泛应用不但能提升病理诊断的准确度、减轻病理医师负担,还能为患者提供个性化的治疗意见和疾病预后判断,但病理人工智能依然处于研发阶段,短时间内难以进入临床实践。
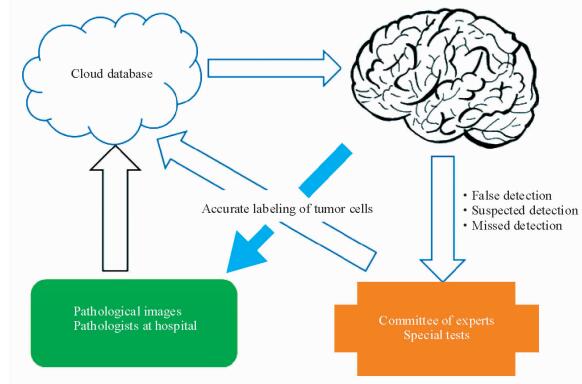
首先,病理诊断是最终诊断,不允许出错,而目前病理人工智能的准确度还不能达到100%。因此要增加医疗数据,尤其是疑难病理数据,通过病理专家的深度参与将人工智能与人类智慧和实验室手段相结合,提高病理诊断的准确性;此外,还要整合免疫组化、分子检测等先进的医学技术手段和临床病例与治疗信息,从多个角度减少误诊率和漏诊率,进一步提高人工智能诊断的准确性(图 5)。其次,病理学是一门经验性医学,人工智能只有学到足够知识才能提高准确性,然而优秀的病理资源主要掌握在三甲医院病理科,共享意识淡漠,研发算法团队拿不到足够的病理样本,从而导致肿瘤样本中病理类型存在偏移和不足,影响人工智能的准确性。
|
图 5 提高病理人工智能深度学习能效的流程图 Fig 5 Flow chart of improving efficiency in deep learning of pathological artificial intelligence |
目前,人工智能正实现着“使计算机去做过去只有人才能做的工作”这一构想,基于统计学、信息论和控制论的计算机技术在医疗领域显示出独特优势,其对医疗大数据的处理能力比人脑更快、更准确、更具重复性。病理学可能会成为人工智能攻克的前沿阵地。如在胃癌的早期筛查工作中协同多家三甲医院参与可产生大量的数据,这些数据除比较容易诊断的肿瘤外,还可能有大量疑似病例和漏诊病例,通过对这些数据进行深入分析和学习,将会提高人工智能诊断的准确性和特异性,从而推动胃癌的早期筛查工作。病理人工智能辅助诊断已实现了形态上的定量分析和细胞学的初筛,在国内外也开始了商业化运作;而组织病理学诊断,包括疾病的预后诊断、病理学分类和良恶性判断等在实验室也取得了显著进展。随着临床医师参与度的提升以及社会资本的进入,病理人工智能必将飞速发展。
| [1] | CHEN W, ZHENG R, BAADE P D, ZHANG S, ZENG H, BRAY F, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66: 115–132. DOI: 10.3322/caac.21338 |
| [2] | MUSIB M, WANG F, TARSELLI M A, YOHO R, YU K H, ANDRÉ R M, et al. Artificial intelligence in research[J]. Science, 2017, 357: 28–30. DOI: 10.1126/science.357.6346.28 |
| [3] | RUSSELL S. Artificial intelligence:the future is superintelligent[J]. Nature, 2017, 548: 520–521. DOI: 10.1038/548520a |
| [4] | Oncologists partner with Watson on genomics[J/OL]. Cancer Discov, 2015, 5:788.doi:10.1158/2159-8290.CD-NB2015-090. |
| [5] | LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521: 436–444. DOI: 10.1038/nature14539 |
| [6] | MAMOSHINA P, VIEIRA A, PUTIN E, ZHAVORONKOV A. Applications of deep learning in biomedicine[J]. Mol Pharm, 2016, 13: 1445–1454. DOI: 10.1021/acs.molpharmaceut.5b00982 |
| [7] | GURCAN M N, BOUCHERON L E, CAN A, MADABHUSHI A, RAJPOOT N M, YENER B. Histopathological image analysis:a review[J]. IEEE Rev Biomed Eng, 2009, 2: 147–171. DOI: 10.1109/RBME.2009.2034865 |
| [8] | YING X, MONTICELLO T M. Modern imaging technologies in toxicologic pathology:an overview[J]. Toxicol Pathol, 2006, 34: 815–826. DOI: 10.1080/01926230600918983 |
| [9] | WEBSTER J D, DUNSTAN R W. Whole-slide imaging and automated image analysis:considerations and opportunities in the practice of pathology[J]. Vet Pathol, 2014, 51: 211–223. DOI: 10.1177/0300985813503570 |
| [10] | YE J J. Artificial intelligence for pathologists is not near-it is here:description of a prototype that can transform how we practice pathology tomorrow[J]. Arch Pathol Lab Med, 2015, 139: 929–935. DOI: 10.5858/arpa.2014-0478-OA |
| [11] | SENDERS J T, STAPLES P C, KARHADE A V, ZAKI M M, GORMLEY W B, BROEKMAN M L D, et al. Machine learning and neurosurgical outcome prediction:a systematic review[J/OL]. World Neurosurg, 2017. pii:S1878-8750(17) 31665-0. doi:10.1016/j.wneu.2017.09.149. |
| [12] | KORBAR B, OLOFSON A M, MIRAFLOR A P, NICKA C M, SURIAWINATA M A, TORRESANI L, et al. Deep learning for classification of colorectal polyps on whole-slide images[J/OL]. J Pathol Inform, 2017, 8:30.doi:10.4103/jpi.jpi_34_17. |
| [13] | SHIN H C, ROTH H R, GAO M, LU L, XU Z, NOGUES I, et al. Deep convolutional neural networks for computer-aided detection:CNN architectures, dataset characteristics and transfer learning[J]. IEEE Trans Med Imaging, 2016, 35: 1285–1298. DOI: 10.1109/TMI.2016.2528162 |
| [14] | CAICEDO J C, COOPER S, HEIGWER F, WARCHAL S, QIU P, MOLNAR C, et al. Data-analysis strategies for image-based cell profiling[J]. Nat Methods, 2017, 14: 849–863. DOI: 10.1038/nmeth.4397 |
| [15] | IGLESIAS J E, SABUNCU M R, AGANJ I, BHATT P, CASILLAS C, SALAT D, et al. An algorithm for optimal fusion of atlases with different labeling protocols[J]. NeuroImage, 2015, 106: 451–463. DOI: 10.1016/j.neuroimage.2014.11.031 |
| [16] | HOUSSAMI N, LEE C I, BUIST D S M, TAO D. Artificial intelligence for breast cancer screening:opportunity or hype?[J]. Breast, 2017, 36: 31–33. DOI: 10.1016/j.breast.2017.09.003 |
| [17] | YOSHIDA H, SHIMAZU T, KIYUNA T, MARUGAME A, YAMASHITA Y, COSATTO E, et al. Automated histological classification of whole-slide images of gastric biopsy specimens[J/OL]. Gastric cancer, 2017. doi:10.1007/s10120-017-0731-8. |
| [18] | KWAK J T, HEWITT S M. Multiview boosting digital pathology analysis of prostate cancer[J]. Comput Methods Programs Biomed, 2017, 142: 91–99. DOI: 10.1016/j.cmpb.2017.02.023 |
| [19] | HAJ-HASSAN H, CHADDAD A, HARKOUSS Y, DESROSIERS C, TOEWS M, TANOUGAST C. Classifications of multispectral colorectal cancer tissues using convolution neural network[J/OL]. J Pathol Inform, 2017, 8:1.doi:10.4103/jpi.jpi_47_16. |
| [20] | VU T H, MOUSAVI H S, MONGA V, RAO G, RAO U K. Histopathological image classification using discriminative feature-oriented dictionary learning[J]. IEEE Trans Med Imaging, 2016, 35: 738–751. DOI: 10.1109/TMI.2015.2493530 |
| [21] | ZADEH SHIRAZI A, SEYYED MAHDAVI CHABOK S J, MOHAMMADI Z. A novel and reliable computational intelligence system for breast cancer detection[J/OL]. Med Biol Eng Comput, 2017. doi:10.1007/s11517-017-1721-z. |
| [22] | ARAÚJO T, ARESTA G, CASTRO E, ROUCO J, AGUIAR P, ELOY C, et al. Classification of breast cancer histology images using Convolutional Neural Networks[J/OL]. PLoS One, 2017, 12:e0177544. doi:10.1371/journal.pone.0177544. |
| [23] | QAISER T, MUKHERJEE A, REDDY PB C, MUNUGOTI S D, TALLAM V, PITKAAHO T, et al. HER2 challenge contest:a detailed assessment of automated HER2 scoring algorithms in whole slide images of breast cancer tissues[J/OL]. Histopathology, 2017. doi:10.1111/his.13333. |
| [24] | VANDENBERGHE M E, SCOTT M L, SCORER P W, SODERBERG M, BALCERZAK D, BARKER C. Relevance of deep learning to facilitate the diagnosis of HER2 status in breast cancer[J/OL]. Sci Rep, 2017, 7:45938. doi:10.1038/srep45938. |
| [25] | SHARMA H, ZERBE N, KLEMPERT I, HELLWICH O, HUFNAGL P. Deep convolutional neural networks for automatic classification of gastric carcinoma using whole slide images in digital histopathology[J/OL]. Comput Med Imaging Graph, 2017. doi:10.1016/j.compmedimag.2017.06.001. |
 2017, Vol. 38
2017, Vol. 38


