尽管近年来胃癌发病率总体呈逐年下降趋势,但仍居全世界所有恶性肿瘤的第4位,死亡率居第2位[1-2]。随着各种辅助治疗手段的进步,早期胃癌患者术后的5年生存率及生活质量得到了极大提高,但晚期胃癌中位总生存期(overall survival,OS)仍不足1年[3]。因此,提高胃癌早期检出率成为减少胃癌患者死亡率的关键环节。目前胃癌常用的诊断方法有放射学检查、肿瘤标志物检查、内镜检查等,但是放射学检查的早期检出率太低;常用的肿瘤标志物检查虽然经济便捷但灵敏度和特异度均较差;内镜检查虽然分辨率高,且配合活组织检查准确率高,但是检查过程痛苦、花费高,难以广泛普及。因此探寻和鉴定可用于胃癌早期诊断的新型分子标志物对于提高胃癌的早期检出率及改善预后有重要的临床意义。
随着人类基因组计划的实施,我们认识到绝大多数哺乳动物基因组被转录产生长链非编码RNA(long non-coding RNA,lncRNA)[4]。lncRNA是长度在200~100 000 nt之间、缺乏蛋白质编码能力的RNA分子。真核生物的lncRNA可以在表观遗传学水平、转录水平和转录后水平等多个层面调控基因的表达[5],是细胞生长、凋亡、分化、侵袭和干细胞多样性的关键调节剂[6-8]。近年来,越来越多的lncRNA被发现与肿瘤的发生、发展密切相关。lncRNA-XIST是位于X染色体失活中心的XIST基因的转录产物,是哺乳动物中X染色体失活过程的关键性起始物[9]。研究发现lncRNA-XIST在肝癌、肺癌、鼻咽癌等肿瘤中表达失调,并且细胞实验证实这种失调会促进肿瘤细胞增殖和侵袭[10-13]。目前尚未见到有关lncRNA-XIST在胃癌患者血浆中表达情况及血浆lncRNA-XIST水平与胃癌发病风险关系的报道,故本研究旨在通过检测组织及血浆中lncRNA-XIST的表达情况,分析血浆lncRNA-XIST与胃癌发病风险的关系,并探讨其临床诊断价值,评估其作为胃癌相关肿瘤标志物的潜力。
1 资料和方法 1.1 病例资料收集2017年2月至7月于第二军医大学长海医院胃肠外科就诊的40例胃癌患者手术切除的肿瘤组织及相应的癌旁组织。入组标准:(1)术前经我院或其他三甲医院病理科确诊的原发性胃腺癌患者;(2)术前未经过化学治疗以及放射治疗;(3)无其他肿瘤病史;(4)近期无输血史;(5)有完整的流行病学资料及相关临床信息。另外收集同期相同入组标准的90例胃癌患者术前的静脉血液标本作为胃癌组,以同期在第二军医大学长海医院体检中心体检的无肿瘤病史的90例健康体检者的静脉血液标本作为对照组。其中胃癌组男性64例,女性26例,年龄43~82岁,中位年龄66岁,≤60岁24例,>60岁66例;对照组男性60例,女性30例,年龄48~77岁,中位年龄64岁,≤60岁27例,>60岁63例。两组性别及年龄的差异均无统计学意义(P均>0.05),研究数据具有可比性。本研究已通过第二军医大学长海医院医学伦理委员会批准,所有患者签署知情同意书。
1.2 主要试剂试验中TRIzol试剂购自Invitrogen公司,SYBR® Premix Ex TaqTMⅡ试剂盒(AK8405)、PrimeScripTM RT Master Mix试剂盒(AK4302)购自宝生物工程(大连)有限公司,血液总RNA快速提取试剂盒(离心柱型)购自北京百泰克生物技术有限公司,lncRNA-XIST与内参基因GAPDH引物由上海杰李生物技术有限公司合成。
1.3 样本收集所有肿瘤及其癌旁组织在手术切除30 min内放入含RNA保存液的冻存管后置入-80 ℃冰箱保存;收集胃癌组和对照组研究对象的清晨空腹静脉血5 mL置于EDTA抗凝管中,2 h内4 ℃ 800×g离心5 min,小心吸取上清液至无酶离心管中,然后4 ℃ 13 000×g离心10 min,吸取上清,置于-80 ℃冰箱保存备用。
1.4 组织及血浆RNA提取按照TRIzol试剂盒说明书提取胃癌及癌旁组织中总RNA, 紫外分光光度计测定RNA浓度及纯度。取上述收集血浆约1.5 mL,按照血液总RNA快速提取试剂盒说明书提取胃癌组及对照组血浆样本的总RNA,提取的RNA用30 μL DEPC水洗脱后置于-80 ℃冰箱备用。
1.5 qPCR检测首先设计2对引物:一对(lncRNA-XIST-F、lncRNA-XIST-R)用于扩增lncRNA-XIST,另一对(GAPDH-F、GAPDH-R)扩增GAPDH。序列如下:lncRNA-XIST-F 5′-GCA TTA TGT AAA TCA GGG GTC CAC-3′,lncRNA-XIST-R 5′-TAT GAA CAG CAG GCC AAA TCC AA-3′;GAPDH-F 5′-CTC TGA CTT CAA CAG CGA CAC C-3′,GAPDH-R 5′-CTG TTG CTG TAG CCA AAT TCG TT-3′。然后取组织或血浆中提取的RNA,根据PrimeScripTMRT Master Mix试剂盒操作说明进行反转录:采用20 μL反应体系,加入Master Mix试剂4 μL,反应条件为37 ℃ 15 min、85 ℃ 5 s,反转录合成cDNA。qPCR采用20 μL反应体系,加入SYBR® Premix Ex TaqTMⅡ试剂10 μL、cDNA 1 μL、引物各0.8 μL、补DEPC水至20 μL,反应条件为42 ℃ 300 s,95 ℃ 30 s、95 ℃ 5 s、60 ℃ 34 s,40个循环。所有样本均设定复孔对照。qPCR反应结束后用配套的计算机软件计算出各反应管内的Ct值,采用2-ΔCt(ΔCt=Ct目的基因-Ct内参基因)或2-ΔΔCt[ΔΔCt=(Ct实验组目的基因-Ct实验组内参基因)-(Ct对照组目的基因-Ct对照组内参基因)]法计算组织及血浆中lncRNA-XIST的相对表达水平。
1.6 肿瘤标志物的测定采集术前胃癌患者及健康体检者静脉血3 mL送第二军医大学长海医院检验科,采用ACS:180系列全自动化学发光免疫分析仪测定癌胚抗原(carcino-embryonic antigen, CEA)和糖类抗原(carbohydrate antigen, CA)19-9、CA72-4的含量。阳性界定值为:CEA>5 ng/mL, CA19-9>37 U/mL, CA72-4>9.8 U/mL。
1.7 统计学处理所有数据均应用SPSS 21.0软件进行分析。所有资料均用Shapira-Wilkinson法检验其正态性。若计量资料符合正态分布,以x±s表示,组间比较用t检验和单因素方差分析;若不符合正态分布,则以中位数(下四分位数,上四分位数)表示,组间比较采用非参数检验。通过绘制受试者工作特征(receiver operating characteristic,ROC)曲线判断血浆中lncRNA-XIST表达水平在胃癌诊断中的灵敏度和特异度。所有统计检验均为双侧检验,检验水准(α)为0.05。
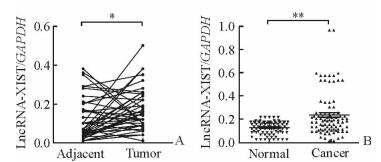
2 结果 2.1 胃癌组织及血浆中lncRNA-XIST的表达水平根据qPCR数据计算出的2-ΔCt值代表lncRNA-XIST与内参基因表达水平的相对倍数关系,2-ΔCt值越大表示lncRNA-XIST表达水平越高。结果显示,胃癌组织lncRNA-XIST的2-ΔCt值为0.150(0.094, 0.247),高于癌旁组织的0.085(0.041, 0.193),差异有统计学意义(P=0.012,图 1A)。胃癌患者术前静脉血浆lncRNA-XIST的2-ΔCt值为0.189(0.119, 0.256),与健康体检者[0.144(0.095, 0.180)]相比差异有统计学意义(P < 0.01,图 1B)。
|
图 1 LncRNA-XIST在胃癌患者组织和血浆中的表达水平 Fig 1 Expression levels of lncRNA-XIST in tissues and plasma of gastric carcinoma patients Real-time quantitative PCR results. A: Relative expression of lncRNA-XIST in gastric cancer tissues and adjacent non-tumor tissues (n=40, *P < 0.05); B: Relative expression of plasmatic lncRNA-XIST of patients with gastric cancer and healthy volunteers (n=90, **P < 0.01). lncRNA-XIST: Long non-coding RNA XIST; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase |
2.2 组织中lncRNA-XIST水平与临床病理参数的关系
组织中lncRNA-XIST的2-ΔΔCt值代表了胃癌组织与癌旁组织中lncRNA-XIST水平的相对倍数关系。2-ΔΔCt值越大,表示lncRNA-XIST在肿瘤组织中的表达对比癌旁组织升高越明显。如表 1所示,lncRNA-XIST水平与TNM分期、淋巴结转移情况、分化程度有关,TNM Ⅲ~Ⅳ期、有淋巴结转移、低分化腺癌的癌组织中lncRNA-XIST水平均升高(P < 0.05);lncRNA-XIST水平与患者性别、年龄、肿瘤大小、Ki-67无关。
|
|
表 1 胃癌患者癌组织中lncRNA-XIST水平与临床病理特征的关系 Tab 1 Correlation between levels of cancer tissues lncRNA-XIST and clinicopathological features in gastric cancer patients |
2.3 血浆中lncRNA-XIST水平与病理参数的关系
检测结果(表 2)与癌组织lncRNA-XIST的表达结果相符合,胃癌患者血浆中lncRNA-XIST的2-ΔCt值与TNM分期、淋巴结转移、分化程度有关(P < 0.05),而与患者性别、年龄、肿瘤大小、Ki-67无关。
|
|
表 2 胃癌患者血浆中lncRNA-XIST水平与临床病理特征的关系 Tab 2 Correlation between levels of plasma lncRNA-XIST and clinicopathological features in gastric cancer patients |
2.4 血浆lncRNA-XIST在胃癌诊断中的价值
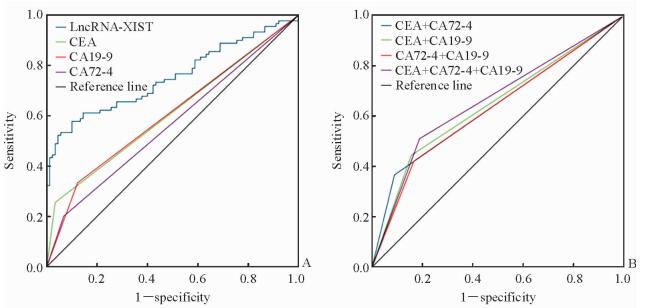
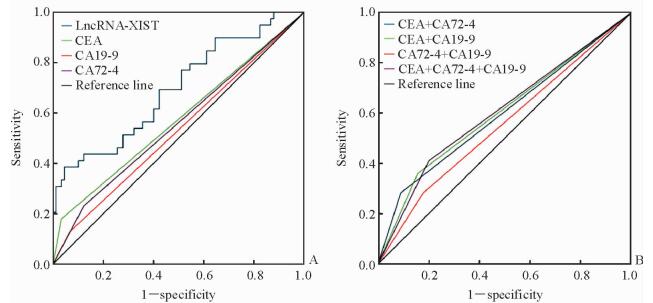
血浆lncRNA-XIST诊断胃癌时ROC曲线下面积(area under curve, AUC)为0.753(95%CI 0.681~0.825,P < 0.001)。通过约登指数(敏感度+特异度-1)确定截断值。当截断值为0.197时,其检测灵敏度为51.1%,特异度为95.6%。同时,我们检测两组的3种常用肿瘤标志物,结果显示血浆lncRNA-XIST诊断胃癌的灵敏度及特异度均高于CEA、CA19-9、CA72-4单独及联合检测(图 2,表 3)。血浆lncRNA-XIST诊断TNMⅠ~Ⅱ期胃癌时AUC为0.694(95%CI 0.592~0.796,P < 0.01),灵敏度为38.5%,特异度为95.6%,也均优于CEA、CA19-9、CA72-4单独及联合检测(表 3、图 3)。
|
图 2 血浆lncRNA-XIST及常用肿瘤标志物诊断胃癌的ROC曲线 Fig 2 ROC analysis of plasma lncRNA-XIST and commonly-used tumor markers for diagnosing gastric cancer A: ROC analysis of lncRNA-XIST, CEA, CA19-9 and CA72-4; B: ROC analysis of combined detection of CEA, CA19-9 and CA72-4. lncRNA-XIST: Long non-coding RNA XIST; ROC: Receiver operating characteristic; CEA: Carcino-embryonic antigen; CA: Carbohydrate antigen |
|
|
表 3 血清lncRNA-XIST在胃癌诊断中的效能分析 Tab 3 Efficacy analysis of plasma lncRNA-XIST in diagnosis of gastric cancer |
|
图 3 血浆lncRNA-XIST及常用肿瘤标志物诊断TNMⅠ~Ⅱ期胃癌的ROC曲线 Fig 3 ROC analysis of plasmatic lncRNA-XIST and commonly-used tumor markers for diagnosing TNM Ⅰ-Ⅱ stage gastric cancer A: ROC analysis of lncRNA-XIST, CEA, CA19-9 and CA72-4; B: ROC analysis of combined detection of CEA, CA19-9 and CA72-4. lncRNA-XIST: Long non-coding RNA XIST; ROC: Receiver operating characteristic; CEA: Carcino-embryonic antigen; CA: Carbohydrate antigen |
3 讨论
我国是胃癌高发国家,但胃镜检查率低,早期胃癌检出率不到10%,远低于日韩(均大于50%)的早期检出率[14-15]。目前临床上最常用的胃癌肿瘤标志物为CEA、CA19-9、CA72-4,但灵敏度均较低。Yang等[16]通过统计发现CEA、CA19-9、CA72-4对胃癌的诊断灵敏度仅分别为25.5%、38.7%、33%,3者联合检测虽然能将灵敏度提高到66.6%,但特异度明显下降。因此寻找灵敏度及特异度更高的新型肿瘤标志物成为目前胃癌研究的热点。LncRNA可以在多个层面上调控肿瘤相关基因的表达水平,包括表观遗传调控、转录调控以及转录后调控[17]。Chen等[18]和Ma等[19]的研究发现XIST在胃癌肿瘤组织中的表达较癌旁组织增高,且与TNM分期、淋巴结转移等病理因素密切相关,随后在细胞实验中发现使用慢病毒干扰XIST表达后可以有效抑制肿瘤细胞的增殖和侵袭,并推测XIST可以作为竞争性内源RNA(competing endogenous RNA,ceRNA)竞争性抑制微RNA(microRNA,miRNA),从而通过miR-101/EZH2、miR-497/MACC1等通路促进胃癌的发生与转移。
Arita等[20]检测了胃癌患者血浆中多种lncRNA的稳定性,发现除了高温孵化、反复冻融或长时间放置等极端条件外,血浆lncRNA水平非常稳定。即使某些lncRNA长度较长,容易断裂后以片段的形式存在外周血中,这部分片段也是十分稳定的[21],只需挑选出合适的引物进行qPCR也可以检测出原lncRNA在血浆中的真实表达水平, 这是lncRNA作为一种可靠生物标志物的前提。
在本研究中,我们通过qPCR对胃癌患者的癌及癌旁组织、血浆和健康体检者血浆中的lncRNA-XIST表达水平进行分析,发现胃癌组织中lncRNA-XIST的表达水平高于癌旁组织,胃癌组血浆中lncRNA-XIST表达高于健康体检组,且胃癌组织及血浆中的lncRNA-XIST水平与胃癌TNM分期、淋巴结转移、分化程度相关,肿瘤分期晚、有淋巴结转移、分化程度低的患者癌组织及血浆中的lncRNA-XIST水平相对较高;血浆lncRNA-XIST单独诊断胃癌及TNM Ⅰ~Ⅱ期胃癌的灵敏度和特异度均高于CEA、CA19-9、CA72-4单独检测或互相联合检测时,表明无论是对胃癌还是相对早期的TNM Ⅰ~Ⅱ期胃癌,血浆lncRNA-XIST的检测效能均优于目前临床常用的这3种肿瘤标志物,特别是有较高的灵敏度,这有利于在早期筛查出更多的肿瘤患者,从而改善预后。综合以上结果,可以考虑将lncRNA-XIST作为胃癌诊断的潜在肿瘤标志物。
由于本研究纳入的样本数量小,收集样本时间短,这导致某些分组数量少,以及lncRNA-XIST表达与患者预后的相关性未能进行分析。因此,我们将在下一步的研究中扩大样本量,完善术后随访,进一步分析血浆中lncRNA-XIST的表达水平在诊断胃癌及判断预后中的意义。
| [1] | CARCAS L P. Gastric cancer review[J/OL]. J Carcinog, 2014, 13:14.doi:10.4103/1477-3163.146506. |
| [2] | TORRE L A, BRAY F, SIEGEL R L, FERLAY J, LORTET-TIEULENT J, JEMAL A. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65: 87–108. DOI: 10.3322/caac.21262 |
| [3] | CERVANTES A, RODA D, TARAZONA N, ROSELLÍ S, PÉREZ-FIDALGO J A. Current questions for the treatment of advanced gastric cancer[J]. Cancer Treat Rev, 2013, 39: 60–67. DOI: 10.1016/j.ctrv.2012.09.007 |
| [4] | ESTELLER M. Non-coding RNAs in human disease[J]. Nat Rev Genet, 2011, 12: 861–874. |
| [5] | RINN J L, CHANG H Y. Genome regulation by long noncoding RNAs[J]. Annu Rev Biochem, 2012, 81: 145–166. DOI: 10.1146/annurev-biochem-051410-092902 |
| [6] | GUTTMAN M, DONAGHEY J, CAREY B W, GARBER M, GRENIER J K, MUNSON G, et al. lincRNAs act in the circuitry controlling pluripotency and differentiation[J]. Nature, 2011, 477: 295–300. DOI: 10.1038/nature10398 |
| [7] | LOEWER S, CABILI M N, GUTTMAN M, LOH Y H, THOMAS K, PARK I H, et al. Large intergenic non-coding RNA-RoR modulates reprogramming of human induced pluripotent stem cells[J]. Nat Genet, 2010, 42: 1113–1117. DOI: 10.1038/ng.710 |
| [8] | TSAI M C, MANOR O, WAN Y, MOSAMMAPARAST N, WANG J K, LAN F, et al. Long noncoding RNA as modular scaffold of histone modification complexes[J]. Science, 2010, 329: 689–693. DOI: 10.1126/science.1192002 |
| [9] | BROWN C J, BALLABIO A, RUPERT J L, LAFRENIERE R G, GROMPE M, TONLORENZI R, et al. A gene from the region of the human X inactivation centre is expressed exclusively from the inactive X chromosome[J]. Nature, 1991, 349: 38–44. DOI: 10.1038/349038a0 |
| [10] | FANG J, SUN C C, GONG C. Long noncoding RNA XIST acts as an oncogene in non-small cell lung cancer by epigenetically repressing KLF2 expression[J]. Biochem Biophys Res Commun, 2016, 478: 811–817. DOI: 10.1016/j.bbrc.2016.08.030 |
| [11] | SONG P, YE L F, ZHANG C, PENG T, ZHOU X H. Long non-coding RNA XIST exerts oncogenic functions in human nasopharyngeal carcinoma by targeting miR-34a-5p[J]. Gene, 2016, 592: 8–14. DOI: 10.1016/j.gene.2016.07.055 |
| [12] | ZHUANG L K, YANG Y T, MA X, HAN B, WANG Z S, ZHAO Q Y, et al. MicroRNA-92b promotes hepatocellular carcinoma progression by targeting Smad7 and is mediated by long non-coding RNA XIST[J/OL]. Cell Death Dis, 2016, 7:e2203.doi:10.1038/cddis.2016.100. |
| [13] | HUANG Y S, CHANG C C, LEE S S, JOU Y S, SHIH H M. Xist reduction in breast cancer upregulates AKT phosphorylation via HDAC3-mediated repression of PHLPP1 expression[J]. Oncotarget, 2016, 7: 43256–43266. DOI: 10.18632/oncotarget.v7i28 |
| [14] | JUNG K W, WON Y J, OH C M, KONG H J, LEE D H, LEE K H; Community of Population-Based Regional Cancer Registries. Cancer statistics in Korea:incidence, mortality, survival, and prevalence in 2014[J]. Cancer Res Treat, 2017, 49: 292–305. DOI: 10.4143/crt.2017.118 |
| [15] | HIGASHI T, MACHⅡ R, AOKI A, HAMASHIMA C, SAITO H. Evaluation and revision of checklists for screening facilities and municipal governmental programs for gastric cancer and colorectal cancer screening in Japan[J]. Jpn J Clin Oncol, 2010, 40: 1021–1030. DOI: 10.1093/jjco/hyq091 |
| [16] | YANG A P, LIU J, LEI H Y, ZHANG Q W, ZHAO L, YANG G H. CA72-4 combined with CEA, CA125 and CA19-9 improves the sensitivity for the early diagnosis of gastric cancer[J]. Clin Chim Acta, 2014, 437: 183–186. DOI: 10.1016/j.cca.2014.07.034 |
| [17] | HE Y, MENG X M, HUANG C, WU B M, ZHANG L, LÜ X W, et al. Long noncoding RNAs:novel insights into hepatocelluar carcinoma[J]. Cancer Lett, 2014, 344: 20–27. DOI: 10.1016/j.canlet.2013.10.021 |
| [18] | CHEN D L, JU H Q, LU Y X, CHEN L Z, ZENG Z L, ZHANG D S, et al. Long non-coding RNA XIST regulates gastric cancer progression by acting as a molecular sponge of miR-101 to modulate EZH2 expression[J/OL]. J Exp Clin Cancer Res, 2016, 35:142. doi:10.1186/s13046-016-0420-1. |
| [19] | MA L, ZHOU Y, LUO X, GAO H, DENG X, JIANG Y. Long non-coding RNA XIST promotes cell growth and invasion through regulating miR-497/MACC1 axis in gastric cancer[J]. Oncotarget, 2017, 8: 4125–4135. |
| [20] | ARITA T, ICHIKAWA D, KONISHI H, KOMATSU S, SHIOZAKI A, SHODA K, et al. Circulating long non-coding RNAs in plasma of patients with gastric cancer[J]. Anticancer Res, 2013, 33: 3185–3193. |
| [21] | WANG Y, LI Z, ZHENG S, ZHOU Y, ZHAO L, YE H, et al. Expression profile of long non-coding RNAs in pancreatic cancer and their clinical significance as biomarkers[J]. Oncotarget, 2015, 6: 35684–35698. |
 2017, Vol. 38
2017, Vol. 38


