2. 中国科学院大学化学与化工学院无机化学教研室, 北京 100049;
3. 国家蛋白质科学中心(上海), 上海 200031;
4. 首都医科大学附属北京世纪坛医院, 北京 100038
2. Department of Inorganic Chemistry, School of Chemistry and Chemical Engineering, University of Chinese Academy of Sciences, Beijing 100049, China;
3. National Center for Protein Science Shanghai, Shanghai 200031, China;
4. Beijing Shijitan Hospital, Capital Medical University, Beijing 100038, China
人间充质干细胞(human mesenchymal stem cells,hMSCs)具有分化为脂肪细胞、成骨细胞和神经细胞等细胞谱系的能力, 被认为是理想的工程细胞[1-2]。然而,由于缺乏对干细胞早期分化状态进行有效且可靠鉴别的生物学标志物,极大阻碍了干细胞的分化机制研究和临床应用[3]。近年来,人们开始关注干细胞分化过程中的力学性质变化,并认为力学性质可作为鉴定干细胞分化潜能的物理生物学标记[4]。例如,Chen等[5]发现羊水来源的干细胞在成骨方向分化诱导培养液中培养4周后硬度下降;Hammerick等[6]比较了人成纤维细胞和脂肪来源的诱导多能干细胞的硬度,认为分化潜能越大,细胞越硬;González-Cruz等[7]进一步确定了在分化过程中干细胞的弹性和黏弹性会发生变化。虽然以上结果表明干细胞力学性质可能与其分化状态相关,但由于细胞具有很大的异质性,即使一个细胞的不同位置,其力学性质也可能不同,这对准确测量干细胞的力学性质提出了挑战[8]。
原子力显微镜(atomic force microscopy, AFM)作为一种可以测量材料力学性质的工具被越来越多地应用于研究干细胞的力学性质与分化状态的关系[4]。然而,目前的细胞力学性质测量多是基于平均杨氏模量或局部细胞弹性模量,难以精确描述单细胞或细胞群体内的力学异质性。因此,需要一种更加灵敏的检测方法以定量测量单细胞的力学性质。峰值力定量纳米力学作图(peak force quantitative nanomechanical mapping, PF-QNM)技术是一种能够同时进行高分辨率成像和定量力学性能测量的新型AFM成像模式[9],本研究利用该技术定量测量了氯化锂诱导成骨细胞分化过程中hMSCs的力学性质,探讨其作为物理生物学标记来鉴定干细胞早期分化的可行性。
1 材料和方法 1.1 hMSCs的培养与诱导初代脐带来源的hMSCs从山东大学医学院获得,用完全培养液(含10%胎牛血清、100 U/mL青霉素和100 μg/mL链霉素的低糖DMEM,均购自美国Gibco公司)在37 ℃、5% CO2环境中培养。培养的细胞2~3 d更换1次培养液,当细胞生长至85%汇合度时1:3传代,所有实验用细胞均传到第8代。传至第8代的hMSCs以5×103/cm2密度铺于60 mm培养皿中,使用完全培养液培养3~5 d,直到细胞生长至80%汇合,然后分别加入含氯化锂的诱导培养液分别诱导48 h或72 h。由于氯化锂诱导干细胞具有剂量效应[2],本研究采用含30 mmol/L或4 mmol/L氯化锂的完全培养液作为诱导培养液。
1.2 免疫荧光成像经过处理的hMSCs用PBS洗3次,4%多聚甲醛溶液固定10 min后,用透化液(含0.1% Triton X-100的PBS)处理3~5 min;然后用封闭液(含1% BSA的PBS)封闭10 min;封闭后的细胞与OregonGreen® 488鬼笔环肽(1:30溶于封闭液中)共同孵育20 min标记干细胞骨架,用PBS洗3次,每次5 min。用DAPI(1:300溶于封闭液中)复染5 min,PBS洗3次,每次5 min。用激光共聚焦显微镜成像,并利用Image-Pro Plus 6.0软件(美国Media Cybernetics公司)进行分析。
1.3 AFM成像细胞经4%多聚甲醛溶液室温固定30 min,用PBS洗3次后在PBS液相环境下成像。成像所选用的探针为SNL-A(0.35 N/m,美国Bruker公司)。SNL-A针尖材料为硅,悬臂梁为氮化硅,针尖半径2 nm。成像前通过Ramp功能在云母表面做力曲线对针尖探针的弹性常数(k)进行校正并记录各种参数。我们利用AFM的PF-QNM模式在液相中成像,扫描速度为0.5 Hz,样品泊松常数设为0.5。所得结果使用AFM离线处理软件Nano Scope Analysis(美国Bruker公司)进行分析处理。数据拟合使用Origin 8(美国OriginLab公司)处理。
1.4 qPCR检测为考察氯化锂对干细胞命运的影响,用激活转录因子4(activating transcription factor 4, ATF4)表征干细胞成骨分化状态[10]。使用总RNA提取试剂盒(美国ZYMO公司)提取细胞总RNA,并用NanoDrop超微量紫外分光光度计对其定量。利用反转录试剂盒(美国Life Technologies公司)将干细胞总RNA反转录为cDNA。qPCR实验采用25 μL反应体系,按照qPCR试剂盒(美国Life Technologies公司)操作说明书进行qPCR。在反应体系中加入1 μL反转录模板cDNA和ATF4引物(Accession ID: NM_001675; Forward: 5′-AAG GCG GGC TCC TCC GAA TG-3′, Reverse: 5′-CCC AAC AGG GCA TCC AAG TCG A-3′)。反应程序设置为95 ℃ 10 min,然后40个循环(95 ℃ 15 s, 60 ℃ 30 s, 72 ℃ 30 s)。GAPDH(primer Accession ID: NM_001256799; Forward: 5′-GGA GCG AGA TCC CTC CAA AAT-3′, Reverse: 5′-GGC TGT TGT CAT ACT TCT CAT GG-3′)作为内参。数据用2-ΔΔCt法处理。所有实验重复3次。将得到的数据进行归一化处理,将0 h对照组的相关基因表达量设为0,以归一化后基因表达量为纵坐标作图。
1.5 统计学处理利用BioScopeTM CatalystTM AFM软件(美国Bruker公司)收集处理AFM成像图片及数据,共聚焦数据利用Leica Application Suite AF软件(德国Leica公司)进行分析,qPCR结果使用Applied Biosystems StepOneTM Real-Time PCR System软件(美国Applied Biosystems公司)进行处理及分析。使用Origin 8软件(美国OriginLab公司)进行作图及统计学分析,数据以x±s表示,组间差异比较采用t检验,检验水准(α)为0.05。
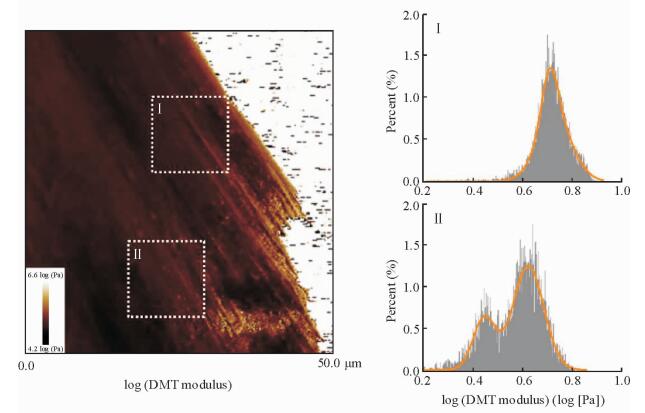
2 结果 2.1 基于PF-QNM纳米级力学谱线(纳米力谱)的干细胞成像图 1是一个典型的PF-QNM模式下干细胞力学成像图,左侧图为干细胞杨氏模量对数成像结果。将成像中每一点的力学性质进行输出,并进行高斯拟合,得到干细胞单细胞的纳米力谱。图 1右侧的直方图显示图像中各数值的相对频率。有趣的是,从直方图我们可以清楚地发现不同区域之间的力学性质是不同的。在区域Ⅰ中仅存在1个峰,弹性模量(Pa)的对数值为0.5~0.9;然而,在区域Ⅱ中2个峰值分别对应于0.46和0.63。这些结果表明,可以通过分析纳米力谱来区分细胞力学性质的异质性。
|
图 1 hMSC单细胞的纳米力谱 Fig 1 Nanomechanical spectroscopy of cultured human mesenchymal stem cell (hMSC) Atomic force microscopy images and the histograms of the values in the areas (Ⅰ and Ⅱ) marked with dashed rectangles, respectively. DMT: Derjaguin-Muller-Toropov |
2.2 干细胞分化过程中的力学表型
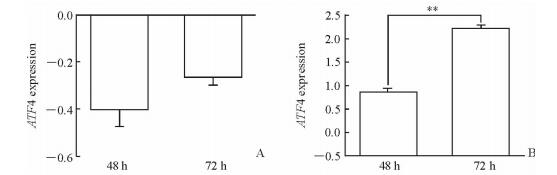
用不同浓度氯化锂处理hMSCs,用鬼笔环肽标记干细胞骨架,通过对免疫荧光染色进行观察,结果显示30 mmol/L氯化锂可以显著改变干细胞形态,表现在干细胞宽度减小(P < 0.05)但长度没有显著变化;而4 mmol/L氯化锂对干细胞形态影响不大(图 2)。用ATF4表征干细胞成骨分化状态,qPCR结果显示30 mmol/L氯化锂促进ATF4表达,而4 mmol/L氯化锂抑制ATF4表达;经过较长时间(72 h)的诱导后,30 mmol/L氯化锂的促进作用变得更为显著(P < 0.01),而4 mmol/L的抑制作用下降(图 3)。表明高浓度氯化锂可促进干细胞的成骨分化潜力,低浓度氯化锂则抑制成骨分化,特别是在诱导早期,抑制作用更明显。

|
图 2 氯化锂诱导hMSCs形态变化 Fig 2 Lithium chloride induced morphological changes of human mesenchymal stem cells (hMSCs) A: Immunofluorescence staining of F-actin (green); B, C: Statistical results of fluorescence immunoassay images showed changes in hMSCs length and width. *P < 0.05. n=3, x±s |
|
图 3 氯化锂诱导hMSCs成骨分化 Fig 3 Lithium chloride induced osteogenic differentiation of human mesenchymal stem cells (hMSCs) Quantitative real-time polymerase chain reaction (qPCR) results of the ATF4 expression after treatment with 4 mmol/L (A) and 30 mmol/L (B) lithium chloride for 48 or 72 h. **P < 0.01. n=3, x±s |
为了通过PF-QNM纳米力学测绘法评估氯化锂对细胞分化的影响,对从不同处理收集的单细胞进行成像。每个处理测量30个细胞,并对得到的结果进行统计分析,获得平均杨氏模量和平均纳米力谱(图 4A、4B)。平均杨氏模量结果表明,高浓度和低浓度的氯化锂都会增加干细胞硬度,并且氯化锂对干细胞力学性质调控具有时间效应,随着处理时间增加干细胞变得更硬(P < 0.05,P < 0.01)。氯化锂对干细胞硬度调控还具有浓度效应,低浓度(图 4A、4B)和高浓度(图 4C、4D)氯化锂都能增加干细胞硬度,但经长时间高浓度氯化锂处理后细胞变得更硬。纳米力谱结果与杨氏平均模型相似。这表明,在不同浓度氯化锂诱导下具有不同分化潜力的干细胞的力学性质会发生变化,平均杨氏模量和平均纳米力谱均可反映氯化锂对干细胞诱导的剂量效应与时间效应。

|
图 4 氯化锂引起hMSCs力学性质变化 Fig 4 Changes of mechanical properties of human mesenchymal stem cells (hMSCs) after treatment with lithium chloride Average Young's modulus (A, C) and nanomechanical spectroscopy (B, D) of hMSCs treated with 4 mmol/L (A, B) and 30 mmol/L (C, D) lithium chloride. *P < 0.05, **P < 0.01. n=3, x±s |
2.3 纳米力谱表征干细胞分化早期力学变化
为了进一步探索力学表型用于鉴别分化早期的可能性,对不同浓度氯化锂诱导干细胞48 h的力学性质进行了分析。在高浓度和低浓度处理的情况下,干细胞的平均杨氏模量(图 5A)几乎相同,难以用于区分干细胞的分化状态变化。然而,纳米力谱(图 5B、5C)则清楚地显示在低浓度处理的情况下有2个峰值,意味着存在2种不同力学性质细胞群体;在高浓度处理的条件下只有1个峰值,说明只存在1种力学性质的单细胞群体。结果表明,与平均杨氏模量相比,纳米力谱可以更敏感地反映分化早期的力学性质变化。

|
图 5 hMSCs分化早期力学表型 Fig 5 Mechanical phenotype of human mesenchymal stem cells (hMSCs) differentiation at the early stage Mean Young's modulus (A) and nanomechanical spectroscopy of hMSCs treated with 4 mmol/L (B) and 30 mmol/L (C) lithium chloride. n=3, x±s |
3 讨论
干细胞的力学性质与其细胞组成、细胞骨架结构以及外部微环境直接相关[11-12]。越来越多的证据表明力学性质的改变密切反映了生理状态下的细胞功能变化[7, 11-13]。为了准确鉴定干细胞分化阶段,除了细胞表面标志物和转基因报告标志物之外,其力学性质可以作为可行的替代标记手段,并已被应用于表征细胞分化、重编程,诊断分化过程的发展,以及监测细胞对环境刺激的反应等重要生理过程。我们的结果显示,hMSCs在受氯化锂调控后,力学性质随着分化状态的变化也发生了改变,杨氏模量升高。此外,杨氏模量的变化反映出干细胞不同的分化趋势。其中经过72 h不同浓度氯化锂诱导后,向成骨方向分化的hMSCs的平均杨氏模量高于成骨分化受抑制的细胞;但在48 h时平均杨氏模量不能将处于不同分化状态的hMSCs加以区分。
PF-QNM成像模式因其高分辨率和实时测量力学性质的特性而被广泛应用于研究细胞等生物样品的力学性质[7, 12-22]。为了进一步分析分化早期(48 h)干细胞力学性质,我们将诱导48 h后hMSCs的PF-QNM力学成像图中数据进行纳米力谱分析。结果显示在诱导48 h后,处于不同分化状态的hMSCs纳米力谱具有显著差异。在低浓度氯化锂处理的情况下,hMSCs表现为具有2种不同力学性质的细胞群体;而在高浓度处理的条件下细胞群体的力学性质变得一致,暗示着异质性降低。
虽然越来越多的证据表明细胞力学性质可以反映干细胞的分化状态[4],但细胞的力学性质与细胞功能以及与其他表型之间的关系和潜在机制仍不清楚。本研究结果表明,在氯化锂诱导后,向成骨方向分化的hMSCs具有更高的硬度。进一步的深入研究将有助于理解氯化锂对干细胞诱导分化的分子机制。AFM已成为表征干细胞等生物样品力学表型的有力工具。通常,人们选择使用平均杨氏模量作为“标记”对分化期间的细胞力学性质进行评估。然而,由于固有的细胞异质性和异步分化,平均杨氏模量识别早期分化中力学性质变化十分困难。本研究中所提出的基于PF-QNM的纳米力谱为描述干细胞分化过程中的力学性质变化提供了更灵敏的方法,有望成为潜在的物理生物学标记用于研究干细胞早期分化事件。
| [1] | KARAHUSEYINOGLU S, CINAR O, KILIC E, KARA F, AKAY G G, DEMIRALP D O, et al. Biology of stem cells in human umbilical cord stroma:in situ and in vitro surveys[J]. Stem Cells, 2007, 25: 319–331. DOI: 10.1634/stemcells.2006-0286 |
| [2] | LU L L, LIU Y J, YANG S G, ZHAO Q J, WANG X, GONG W, et al. Isolation and characterization of human umbilical cord mesenchymal stem cells with hematopoiesis-supportive function and other potentials[J]. Haematologica, 2006, 91: 1017–1026. |
| [3] | CONCONI M T, BURRA P, DI LIDDO R, CALORE C, TURETTA M, BELLINI S, et al. CD105+ cells from Wharton's jelly show in vitro and in vivo myogenic differentiative potential[J]. Int J Mol Med, 2006, 18: 1089–1096. |
| [4] | BONGIORNO T, KAZLOW J, MEZENCEV R, GRIFFITHS S, OLIVARES-NAVARRETE R, McDONALD J F, et al. Mechanical stiffness as an improved single-cell indicator of osteoblastic human mesenchymal stem cell differentiation[J]. J Biomech, 2014, 47: 2197–2204. DOI: 10.1016/j.jbiomech.2013.11.017 |
| [5] | CHEN Q, XIAO P, CHEN J N, CAI J Y, CAI X F, DING H, et al. AFM studies of cellular mechanics during osteogenic differentiation of human amniotic fluid-derived stem cells[J]. Anal Sci, 2010, 26: 1033–1037. DOI: 10.2116/analsci.26.1033 |
| [6] | HAMMERICK K E, HUANG Z, SUN N, LAM M T, PRINZ F B, WU J C, et al. Elastic properties of induced pluripotent stem cells[J]. Tissue Eng Part A, 2011, 17(3/4): 495–502. |
| [7] | GONZÁLEZ-CRUZ R D, FONSECA V C, DARLING E M. Cellular mechanical properties reflect the differentiation potential of adipose-derived mesenchymal stem cells[J/OL]. Proc Natl Acad Sci USA, 2012, 109:E1523-E1529. doi:10.1073/pnas.1120349109. |
| [8] | BAKSH D, SONG L, TUAN R S. Adult mesenchymal stem cells:characterization, differentiation, and application in cell and gene therapy[J]. J Cell Mol Med, 2004, 8: 301–316. DOI: 10.1111/jcmm.2004.8.issue-3 |
| [9] | DARLING E M, TOPEL M, ZAUSCHER S, VAIL T P, GUILAK F. Viscoelastic properties of human mesenchymally-derived stem cells and primary osteoblasts, chondrocytes, and adipocytes[J]. J Biomech, 2008, 41: 454–464. DOI: 10.1016/j.jbiomech.2007.06.019 |
| [10] | OFEK G, WILLARD V P, KOAY E J, HU J C, LIN P, ATHANASIOU K A. Mechanical characterization of differentiated human embryonic stem cells[J/OL]. J Biomech Eng, 2009, 131:061011. doi:10.1115/1.3127262. |
| [11] | PAJEROWSKI J D, DAHL K N, ZHONG F L, SAMMAK P J, DISCHER D E. Physical plasticity of the nucleus in stem cell differentiation[J]. Proc Natl Acad Sci USA, 2007, 104: 15619–15624. DOI: 10.1073/pnas.0702576104 |
| [12] | PILLARISETTI A, DESAI J P, LADJAL H, SCHIFFMACHER A, FERREIRA A, KEEFER C L. Mechanical phenotyping of mouse embryonic stem cells:increase in stiffness with differentiation[J]. Cell Reprogram, 2011, 13: 371–380. DOI: 10.1089/cell.2011.0028 |
| [13] | TAN Y, KONG C W, CHEN S, CHENG S H, LI R A, SUN D. Probing the mechanobiological properties of human embryonic stem cells in cardiac differentiation by optical tweezers[J]. J Biomech, 2012, 45: 123–128. DOI: 10.1016/j.jbiomech.2011.09.007 |
| [14] | SWEERS K, VAN DER WERF K, BENNINK M, SUBRAMANIAM V. Nanomechanical properties of α-synuclein amyloid fibrils:a comparative study by nanoindentation, harmonic force microscopy, and Peakforce QNM[J/OL]. Nanoscale Res Lett, 2011, 6:270. doi:10.1186/1556-276X-6-270. |
| [15] | SWEERS K K, VAN DER WERF K O, BENNINK M L, SUBRAMANIAM V. Atomic force microscopy under controlled conditions reveals structure of C-terminal region of α-synuclein in amyloid fibrils[J]. ACS Nano, 2012, 6: 5952–5960. DOI: 10.1021/nn300863n |
| [16] | BITLER A, DOVER R, SHAI Y. Fractal properties of macrophage membrane studied by AFM[J]. Micron, 2012, 43: 1239–1245. DOI: 10.1016/j.micron.2012.04.009 |
| [17] | ADAMS P G, CADBY A J, ROBINSON B, TSUKATANI Y, TANK M, WEN J, et al. Comparison of the physical characteristics of chlorosomes from three different phyla of green phototrophic bacteria[J]. Biochim Biophys Acta, 2013, 1827: 1235–1244. DOI: 10.1016/j.bbabio.2013.07.004 |
| [18] | KEEFER C L, DESAI J P. Mechanical phenotyping of stem cells[J]. Theriogenology, 2011, 75: 1426–1430. DOI: 10.1016/j.theriogenology.2010.11.032 |
| [19] | TOKARSKA-RODAK M, KOZIOŁ-MONTEWKA M, SKRZYPIEC K, CHMIELEWSKI T, MENDYK E, TYLEWSKA-WIERZBANOWSKA S. Analysis of nanomechanical properties of Borrelia burgdorferi spirochetes under the influence of lytic factors in an in vitro model using atomic force microscopy[J/OL]. Postepy Hig Med Dosw (Online), 2015, 69:1222-1227. doi:10.5604/17322693.1179650. |
| [20] | KHALISOV M M, ANKUDINOV A V, PENNIYAYNEN V A, DOBROTA D, KRYLOV B V. Application of atomic force microscopy for investigation of Na+, K+-ATPase signal-transducing function[J]. Acta Physiol Hung, 2015, 102: 125–130. DOI: 10.1556/036.102.2015.2.2 |
| [21] | ANKUDINOV A V, KHALISOV M M, PENNIYAINEN V A, PODZOROVA S A, KRYLOV B V. Application of atomic force microscopy for studying intracellular signalization in neurons[J]. Tech Phys, 2015, 60: 1540–1544. DOI: 10.1134/S1063784215100047 |
| [22] | SCHILLERS H, MEDALSY I, HU S, SLADE A L, SHAW J E. PeakForce Tapping resolves individual microvilli on living cells[J]. J Mol Recognit, 2016, 29: 95–101. DOI: 10.1002/jmr.v29.2 |
 2017, Vol. 38
2017, Vol. 38


