2. 第二军医大学学员旅学员8队, 上海 200433;
3. 山西农业大学动物科技学院, 晋中 030801;
4. 第二军医大学海军医学系海洋生物技术教研室, 上海 200433
2. The Eighth Student Team, Student Brigade, Second Military Medical University, Shanghai 200433, China;
3. College of Animal and Technology, Shanxi Agricultural University, Jinzhong 030801, Shanxi, China;
4. Department of Marine Biotechnology, Faculty of Naval Medicine, Second Military Medical University, Shanghai 200433, China
长链非编码RNA(long noncoding RNA, lncRNA)是一类长度大于200 nt的非编码RNA,在个体生长发育、疾病发生及发展等生命过程中发挥重要作用[1]。通过比较模式生物间、模式生物与非模式生物间甚至非模式生物间lncRNA的序列、转录位置、空间结构以及生物学功能等特征分析其保守性,并应用于新lncRNA的筛选与鉴定是近年来lncRNA研究的新策略[2]。
物种lncRNA参考数据库的构建是开展lncRNA相关研究的前提条件,其主要步骤包括样品测序、序列组装和数据库比对注释等。模式生物由于具有更为完备的基因组、转录组以及lncRNA自身参考数据库,新lncRNA筛选与鉴定研究的策略相对完善且固定[3]。而非模式生物由于可供参考的数据库信息有限,其lncRNA的筛选、鉴定及其所构建的参考数据库易出现注释不全面、不准确甚至错误等问题[4]。根据lncRNA的保守性特征开发生物信息学分析工具,通过多层次筛选可有效提高非模式生物lncRNA参考数据库构建的准确性,更好地推动新lncRNA的鉴定与功能研究进程[5]。本文就lncRNA保守性的研究现状及其如何运用于非模式生物lncRNA的筛选作一综述。
1 LncRNA的保守性LncRNA的保守性一般表现在一级结构、转录位置、二级结构以及功能与组织特异性等。借鉴蛋白质[6]和微RNA(miRNA)[7]的保守性在序列辨认、靶点预测和生物学效应研究等方面的应用,深入研究lncRNA的保守性有助于解决序列比对、分析和预测等实际问题。
1.1 序列保守性与信使RNA(mRNA)类似,新合成的lncRNA含有5′端7-甲基鸟嘌呤帽子、3′端poly A尾以及开放阅读框(opening reading frame, ORF)等基本结构,需要经过剪接加工成为成熟的lncRNA[8]。通过物种间同源基因的比较分析发现lncRNA的平均长度大约为编码基因长度的1/5(1 000 nt左右),有很大概率含有多个50~100 nt的ORFs,如有研究发现脊椎动物基因间的lncRNA平均含有2~3个外显子[5, 9]。研究选取高表达、经过精确注释的lncRNA构建数据库,分析发现lncRNA外显子的保守性低于mRNA外显子的保守性[10-12],但高于编码基因内含子、基因间序列(intergenic regions)[11, 13-15]以及lncRNA自身内含子[13]的保守性。就不同物种而言,lncRNA的保守性非常低,如研究发现人与小鼠间lncRNA的保守性仅20%,而人和鱼之间甚至降低至仅5%[5]。目前已发现的lncRNA基因座超过58 000个,但实际鉴定出的具有生物学功能的lncRNA却十分有限,其数量远不及蛋白质,可能与lncRNA具有较低的序列保守性有关。
1.2 空间结构的保守性LncRNA的空间结构对其生物学功能具有重要作用。研究表明,生物体内的lncRNA往往比mRNA具有更多的二级结构[16-17],已有实验证实部分lncRNA中的局部区域具有特定的二级结构,如肺癌转移相关转录本1 (metastasis-associated lung adenocarcinoma transcript 1,MALAT1) 3′末端的三螺旋(triple helix)结构具有类似增强子的作用[18],心肌肥厚相关表观遗传调节子(cardiac-hypertrophy-associated epigenetic regulator,CHAER)在5′端的双四环(bi-tetra-loop)结构可能是与Zeste基因增强子同源物2(enhancer of zeste homolog 2,EZH2)结合的必要条件[19]等。需要注意的是,具有相同二级结构lncRNA的基因在物种间未必是同源的[5]。目前尚无lncRNA三级、四级结构的研究报道。近期开发的dChIRP(domain-specific ChIRP)[20]技术可以在天然环境下剖析lncRNA不同结构域的功能,如研究揭示果蝇体内一种雄性果蝇剂量补偿效应所需的lncRNA roX1的三指手掌结构(three-fingered hand)可与染色质相互作用,是未来探索lncRNA高级结构对其生物学功能影响的可行方法之一。
1.3 转录位置的保守性LncRNA转录位置的保守性是指在直系同源序列中lncRNA与编码基因或其他区域相对位置的固定[21],这提示lncRNA对编码基因存在如沉默作用等某种特定的调控。研究发现非蛋白质编码RNA IGF2R反义链(antisense of IGF2R non-protein coding RNA,AIRN)的基因序列和编码基因胰岛素样生长因子2型受体(insulin-like growth factor 2 receptor,IGF2R)基因的启动子之间存在部分重叠,重叠部分转录出的RNA可沉默IGF2R,而剩余大小为118 kb的非重叠AIRN序列则与基因沉默无关[22]。同样,不同哺乳动物间的lncRNA浆细胞瘤转化迁移基因1(plasmacytoma variant translocation 1,PVT1)仅在第1个及中间2个外显子上存在保守性,但它们都位于Myc编码基因的下游[5]。转录位置的保守性是极其容易忽略的,符合这一性质的lncRNA在序列上往往并不保守,且由于其具有功能的部分片段长度较短或其转录需要特定的条件,从而导致这类保守性难以被发现。
1.4 转录和剪接模式的保守性比较lncRNA的外显子是在RNA层面研究物种lncRNA进化保守性的一种常规思路[5, 10, 23]。但研究常发现在两亲缘关系很近的物种中存在lncRNA特异性表达于单一物种的现象,提示有必要进一步从DNA层面比较lncRNA的基因座,分析lncRNA转录和剪接模式的保守性。Hezroni等[5]通过对17种脊椎动物的lncRNA研究发现>70%的lncRNA基因座已存在于5 000万年前的生物体中。但与非灵长类动物相比,人类仅保留了大约20%的lncRNA剪接过程[23],说明lncRNA具有非常高的进化效率。与其他依赖保守DNA或成熟RNA序列的蛋白质或miRNA相比,很多转录lncRNA的基因座会在进化中逐渐改变或丢失,而保留至今的lncRNA的内外显子的序列和结构往往较之前发生了较大的变化。这一方面可能是由于本身具有功能的lncRNA数量并不多,容易在进化过程中丢失[4];另一方面则可能是由于lncRNA需要与其他元件结合才能发挥功能,如与miRNA或蛋白结合发挥内源性竞争作用,未必表现出明显的保守性[24]。
1.5 组织和细胞特异性LncRNA在表观遗传调控、细胞周期调控和细胞分化调控等众多生命活动中发挥重要作用。高度保守的lncRNA往往具有组织特异性,常存在于脑、睾丸等体内重要组织器官中[5, 23, 25],发挥调控胚胎发育、精子生成以及突触传递等重要的生理功能[10]。Ulitsky等[26]在斑马鱼中发现了29条与哺乳动物同源的lncRNA,其中cyrano和megamind主要分布于神经系统中,可能与人胚胎期大脑发育有关。但总体而言,lncRNA分布的组织特异性并不高,在不同生物体内的相同特定细胞中,同源lncRNA的数量非常有限。Kutter等[27]研究发现小鼠肝脏与大鼠肝脏有60%的lncRNA同源,而人肝脏中lncRNA的同源性降低至27%。同样,在人和鼠眼(视锥、视杆和视网膜细胞)[28]、多能干细胞[29]以及胰岛朗格汉斯细胞[30]中lncRNA的同源性结果类似。
1.6 功能保守性尽管lncRNA数量众多,但仅少数具有明确的生物学功能。从保守性入手对lncRNA的序列、结构、加工过程及空间分布等进行比较,最终目的在于揭示lncRNA可能存在的保守功能,如序列上十分保守的Uc.283+A能结合miR-195前体并下调miR-195的表达[31]。LncRNA的保守功能可以体现在多个方面,如具有相似的分子功能、与相同靶基因结合甚至代替其他生物中的同源序列并发挥相似作用,有研究人员发现其他生物中的roX可以在缺失roX的果蝇体内与其相应位点结合[32]。由于目前发现的保守lncRNA数量较少,以及lncRNA活体实验技术的不成熟,发现具有保守功能的lncRNA并对其进行实验验证仍是深入研究lncRNA功能保守性的难点。
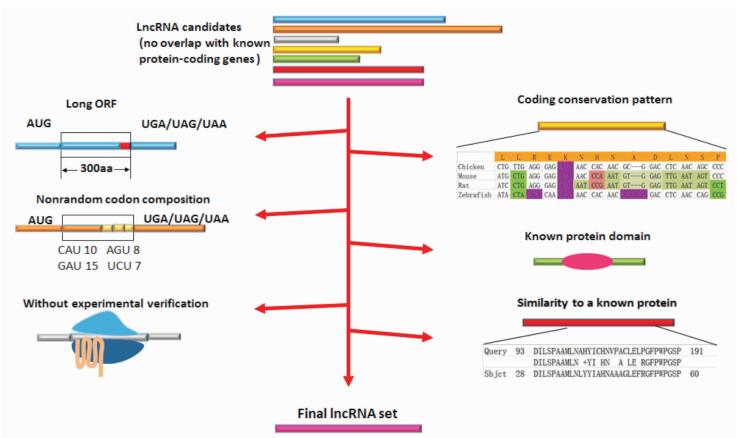
2 保守性在非模式生物lncRNA筛选中的应用非模式生物往往缺少相对应的基因组参考数据库,直接将组装好的序列与模式生物基因组比对,进行lncRNA的筛选会导致比对不精确或比对不全,有时甚至会注释出错误信息。现行的主要处理办法一是选择亲缘较近的模式生物,以求注释得到的信息更加精确,二是与数据库中所有物种进行匹配,以得到尽可能多的注释。借助不断涌现的lncRNA保守性研究成果与mRNA的相互比较,采用正向选择(针对lncRNA)和反向排除(针对mRNA)的方法,不断开发新的软件工具并进行优化和完善[2]成为目前各实验室开展非模式生物lncRNA筛选的重要方式[5, 10, 23, 33]。图 1显示了lncRNA保守性在非模式生物lncRNA筛选中的应用[21]。
|
图 1 LncRNA保守性在非模式生物lncRNA筛选中的应用示意图[21] Fig 1 Diagram of application of lncRNA conservation in screening lncRNA in non-model organisms[21] lncRNA: Long noncoding RNA; ORF: Opening reading frame |
2.1 ORF长度
ORF或外显子的长度大于300 nt是大多数mRNA的特征[9]。采用CPAT[34]和iSeeRNA[35]等工具检测ORF的数量和长度并反向排除含有大于300 nt ORF的序列是lncRNA筛选过程中的简单步骤。显然,仅根据ORF长度进行lncRNA的筛选是十分有限的[9],如目前研究较深入的Xist、Hotair、Meg3等lncRNA的部分外显子长度均大于300 nt。一般ORF长度在lncRNA筛选中并不会单独应用,但由于其操作简便,已成为lncRNA筛选中普遍使用的方法。
2.2 核苷酸或密码子的出现频率与分布LncRNA ORF的核苷酸突变频率高,位置往往呈随机分布,而编码mRNA的ORF这一特征则正好相反,这与长期的生物进化过程需要维持编码蛋白的功能稳定性是一致的。这种由生物选择压力(selective pressure)导致的lncRNA和mRNA ORF之间的核苷酸突变频率差异表明,可以通过比较ORF内不同位置上的碱基构成或测试ORF内碱基突变对其生物学功能的影响来区分lncRNA与mRNA。由于突变频率分析简单、计算快捷,其也已成为区分筛选lncRNA的常用特征参数之一,常见工具有CPC[36]、CONC[37]和CPAT[34]。其中CPC和CONC对比分析的是单个密码子及其表达的氨基酸的突变频率,而CPAT则是观察每个碱基在ORF中以及每2个密码子在整条序列中的出现频率。
2.3 功能性结构域以lncRNA ORF推测翻译的蛋白质虽然也存在小范围、不常见的保守结构域,但其数量和结构种类远少于mRNA翻译的蛋白质所含有的结构域。HMMER(http://www.hmmer.org/)可用于检验转录产物、分析其编码的蛋白质序列并计算其潜在的结构域。可参考的数据库如Pfam[38](http://pfam.xfam.org/)运用多序列对比和隐马尔可夫模型(hidden Markov model, HMM)收录了大量蛋白家族,其条目已超过16 000。GenBank和Ensembl收录的蛋白质更多,但与Pfam收录蛋白质必须经实验验证相比,其存在大量仅由计算机推测获取的蛋白质序列,导致lncRNA假阴性的概率增加。
2.4 实验检验在完成生物信息学筛选后,针对感兴趣lncRNA需进一步实验验证,其验证核心在于是否会翻译出蛋白质。由于多数转录本仅含有个别长的保守ORF,因此可以采用移码突变(frameshift mutations)的方式来干扰其翻译成多肽并探讨其对RNA功能的影响,从而判断其是否为lncRNA[26, 39]。
ORF的长度、密码子的组成与分布、序列所翻译蛋白质的结构域与相似度等特征都是lncRNA保守性运用于非模式生物筛选中的重要参数指标。非模式生物缺少完整、准确的基因组信息,而保守程度的算法也不局限于序列的保守性,还包括空间结构、转录位置以及剪接方式等,导致目前已建立的lncRNA保守性算法难以完全涵盖,实现全面、准确和高效的非模式生物lncRNA的筛选仍有待进一步优化和完善。
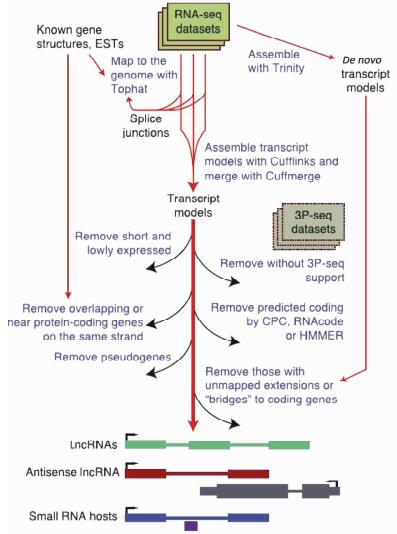
3 PLAR(pipeline for lncRNA annotation from RNA-seq data)流程在lncRNA筛选中的应用以色列Weizmann研究所的Ulitsky教授及其实验室是lncRNA保守性研究[4-5, 21, 26, 40]的顶尖团队,他们构建了一套称为PLAR[5]的包括非模式生物在内的完整的lncRNA筛选方法(图 2)。PLAR的简要过程如下:(1)转录组信息搜集,即通过测序或借助公共数据库获取研究对象完整的转录组信息;(2)转录本的组装,即使用TopHat2将测序所获得的RNA序列借助Ensembl和RefSeq数据库配对到基因组中,配对结果用Cufflinks进一步组装成转录模型,对于碎片化的短序列则使用Trinity进行从头(De novo)组装;(3)信息注释,即采用CuffMerge软件,以Ensembl为参考数据库对所建立的转录模型进行注释;(4)lncRNA的筛选,即利用CPC[36]、HMMER[41]和RNAcode[42]等方法或数据库以及pseudogene(http://pseudogene.org)排除与假基因重叠的转录本,并去除低表达的RNA序列;(5)排除有编码潜力的转录本,即设置一定的参数排除与蛋白质编码基因重叠或相近的序列,如将与编码基因外显子重叠超过50%的单外显子转录本排除;(6)将剩余lncRNA分类,包括基因区间的长链非编码RNA(long intergenic noncoding RNA,lincRNA)、antisense lncRNA以及small RNA hosts等。PLAR的详细过程、使用软件及参数、筛选数据库等信息可参见网站http://webhome.weizmann.ac.il/home/igoru/PLAR。
|
图 2 PLAR流程示意图[5] Fig 2 Diagram of PLAR flow[5] PLAR: Pipeline for lncRNA annotation from RNA-seq data; lncRNA: Long non-coding RNA; ESTs: Expressed sequence tags |
目前已建立的PLAR数据库包括人、恒河猴、斑马鱼、海胆、姥鲨等17种非模式生物及常规模式生物,并发现这些生物中的同源lincRNA在5′端外显子中具有小段保守序列,且部分与转录因子重叠;在哺乳动物中预测超过1 000条具有保守功能的人lincRNA,在非哺乳动物中也具有数百条,这些序列能在基因结构大幅度改变的情况下仅需特定片段中的一小段即可行使其功能,这大大扩展了lncRNA保守性的相关研究。PLAR在常规lncRNA建库策略的基础上加以改进,借助公认的数据库获取最全面的转录组信息(正向选择),通过保守性多层次筛选编码RNA(反向排除),在现有技术条件下可以获得较为准确、完整、高质量的lncRNA数据库,具有较高的推广价值,是目前运用保守性筛选lncRNA的代表性方法之一,可将其应用于几乎所有物种。
4 小结保守性是目前lncRNA研究的热点和难点。尽管研究发现lncRNA在一级结构、空间结构、转录与剪接模式以及分布和功能等方面都存在一定的保守性,但其保守性明显低于mRNA,且需更多的实验数据支撑。在缺少对应基因组数据库的情况下,根据lncRNA与mRNA的保守性同时采用正向选择和反向排除的方法,可以筛选出非模式生物的lncRNA并且构建相应的参考数据库。但由于该过程受到lncRNA和编码基因重叠程度、分析软件、参考数据库以及算法等多种因素的影响,其完整性和准确性在不同实验室间存在着明显差异。随着lncRNA保守性研究的不断深入和生物信息学筛选工具的不断完善,所构建的非模式生物lncRNA参考数据库将更全面和准确,也将更具参考价值。
| [1] | CHEN L L. Linking long noncoding RNA localization and function[J]. Trends Biochem Sci, 2016, 41: 761–772. DOI: 10.1016/j.tibs.2016.07.003 |
| [2] | HOUSMAN G, ULITSKY I. Methods for distinguishing between protein-coding and long noncoding RNAs and the elusive biological purpose of translation of long noncoding RNAs[J]. Biochim Biophys Acta, 2016, 1859: 31–40. DOI: 10.1016/j.bbagrm.2015.07.017 |
| [3] | IYER M K, NIKNAFS Y S, MALIK R, SINGHAL U, SAHU A, HOSONO Y, et al. The landscape of long noncoding RNAs in the human transcriptome[J]. Nat Genet, 2015, 47: 199–208. DOI: 10.1038/ng.3192 |
| [4] | ULITSKY I. Evolution to the rescue:using comparative genomics to understand long non-coding RNAs[J]. Nat Rev Genet, 2016, 17: 601–614. DOI: 10.1038/nrg.2016.85 |
| [5] | HEZRONI H, KOPPSTEIN D, SCHWARTZ M G, AVRUTIN A, BARTEL D P, ULITSKY I. Principles of long noncoding RNA evolution derived from direct comparison of transcriptomes in 17 species[J]. Cell Rep, 2015, 11: 1110–1122. DOI: 10.1016/j.celrep.2015.04.023 |
| [6] | BRAWAND D, SOUMILLON M, NECSULEA A, JULIEN P, CSÁRDI G, HARRIGAN P, et al. The evolution of gene expression levels in mammalian organs[J]. Nature, 2011, 478: 343–348. DOI: 10.1038/nature10532 |
| [7] | BEREZIKOV E. Evolution of microRNA diversity and regulation in animals[J]. Nat Rev Genet, 2011, 12: 846–860. DOI: 10.1038/nrg3079 |
| [8] | QUINN J J, CHANG H Y. Unique features of long non-coding RNA biogenesis and function[J]. Nat Rev Genet, 2016, 17: 47–62. |
| [9] | DINGER M E, PANG K C, MERCER T R, MATTICK J S. Differentiating protein-coding and noncoding RNA:challenges and ambiguities[J/OL]. PLoS Comput Biol, 2008, 4:e1000176. doi:10.1371/journal.pcbi.1000176. |
| [10] | NECSULEA A, SOUMILLON M, WARNEFORS M, LIECHTI A, DAISH T, ZELLER U, et al. The evolution of lncRNA repertoires and expression patterns in tetrapods[J]. Nature, 2014, 505: 635–640. DOI: 10.1038/nature12943 |
| [11] | GUTTMAN M, AMIT I, GARBER M, FRENCH C, LIN M F, FELDSER D, et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals[J]. Nature, 2009, 458: 223–227. DOI: 10.1038/nature07672 |
| [12] | ZHANG Y C, LIAO J Y, LI Z Y, YU Y, ZHANG J P, LI Q F, et al. Genome-wide screening and functional analysis identify a large number of long noncoding RNAs involved in the sexual reproduction of rice[J/OL]. Genome Biol, 2014, 15:512. doi:10.1186/513059-014-0512-1. |
| [13] | CABILI M N, TRAPNELL C, GOFF L, KOZIOL M, TAZON-VEGA B, REGEV A, et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses[J]. Genes Dev, 2011, 25: 1915–1927. DOI: 10.1101/gad.17446611 |
| [14] | MARQUES A C, PONTING C P. Catalogues of mammalian long noncoding RNAs:modest conservation and incompleteness[J]. Genome Biol, 2009, 10: R124. DOI: 10.1186/gb-2009-10-11-r124 |
| [15] | PONJAVIC J, PONTING C P, LUNTER G. Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs[J]. Genome Res, 2007, 17: 556–565. DOI: 10.1101/gr.6036807 |
| [16] | YANG J R, ZHANG J. Human long noncoding RNAs are substantially less folded than messenger RNAs[J]. Mol Biol Evol, 2015, 32: 970–977. DOI: 10.1093/molbev/msu402 |
| [17] | SPITALE R C, FLYNN R A, ZHANG Q C, CRISALLI P, LEE B, JUNG J W, et al. Structural imprints in vivo decode RNA regulatory mechanisms[J]. Nature, 2015, 519: 486–490. DOI: 10.1038/nature14263 |
| [18] | WILUSZ J E, JnBAPTISTE C K, LU L Y, KUHN C D, JOSHUA-TOR L, SHARP P A. A triple helix stabilizes the 3' ends of long noncoding RNAs that lack poly(A) tails[J]. Genes Dev, 2012, 26: 2392–2407. DOI: 10.1101/gad.204438.112 |
| [19] | WANG Z, ZHANG X J, JI Y X, ZHANG P, DENG K Q, GONG J, et al. The long noncoding RNA Chaer defines an epigenetic checkpoint in cardiac hypertrophy[J]. Nat Med, 2016, 22: 1131–1139. DOI: 10.1038/nm.4179 |
| [20] | QUINN J J, ILIK I A, QU K, GEORGIEV P, CHU C, AKHTAR A, et al. Revealing long noncoding RNA architecture and functions using domain-specific chromatin isolation by RNA purification[J]. Nat Biotechnol, 2014, 32: 933–940. DOI: 10.1038/nbt.2943 |
| [21] | ULITSKY I, BARTEL D P. lincRNAs:genomics, evolution, and mechanisms[J]. Cell, 2013, 154: 26–46. DOI: 10.1016/j.cell.2013.06.020 |
| [22] | LATOS P A, PAULER F M, KOERNER M V, ŞENERGIN H B, HUDSON Q J, STOCSITS R R, et al. Airn transcriptional overlap, but not its lncRNA products, induces imprinted Igf2r silencing[J]. Science, 2012, 338: 1469–1472. DOI: 10.1126/science.1228110 |
| [23] | WASHIETL S, KELLIS M, GARBER M. Evolutionary dynamics and tissue specificity of human long noncoding RNAs in six mammals[J]. Genome Res, 2014, 24: 616–628. DOI: 10.1101/gr.165035.113 |
| [24] | TAN J Y, SIREY T, HONTI F, GRAHAM B, PIOVESAN A, MERKENSCHLAGER M, et al. Extensive microRNA-mediated crosstalk between lncRNAs and mRNAs in mouse embryonic stem cells[J]. Genome Res, 2015, 25: 655–666. DOI: 10.1101/gr.181974.114 |
| [25] | CHODROFF R A, GOODSTADT L, SIREY T M, OLIVER P L, DAVIES K E, GREEN E D, et al. Long noncoding RNA genes:conservation of sequence and brain expression among diverse amniotes[J/OL]. Genome Biol, 2010, 11:R72. doi:10.1186/gb-2010-11-7-r72. |
| [26] | ULITSKY I, SHKUMATAVA A, JAN C H, SIVE H, BARTEL D P. Conserved function of lincRNAs in vertebrate embryonic development despite rapid sequence evolution[J]. Cell, 2011, 147: 1537–1550. DOI: 10.1016/j.cell.2011.11.055 |
| [27] | KUTTER C, WATT S, STEFFLOVA K, WILSON M D, GONCALVES A, PONTING C P, et al. Rapid turnover of long noncoding RNAs and the evolution of gene expression[J/OL]. PLoS Genet, 2012, 8:e1002841. doi:10.1371/journal.pgen.1002841. |
| [28] | MUSTAFI D, KEVANY B M, BAI X, MAEDA T, SEARS J E, KHALIL A M, et al. Evolutionarily conserved long intergenic non-coding RNAs in the eye[J]. Hum Mol Genet, 2013, 22: 2992–3002. DOI: 10.1093/hmg/ddt156 |
| [29] | CHEN J, SHISHKIN A A, ZHU X, KADRI S, MAZA I, GUTTMAN M, et al. Evolutionary analysis across mammals reveals distinct classes of long non-coding RNAs[J]. Genome Biol, 2016, 17: 19. DOI: 10.1186/s13059-016-0880-9 |
| [30] | MORÁN I, AKERMAN I, VAN DE BUNT M, XIE R, BENAZRA M, NAMMO T, et al. Human β cell transcriptome analysis uncovers lncRNAs that are tissue-specific, dynamically regulated, and abnormally expressed in type 2 diabetes[J]. Cell Metab, 2012, 16: 435–448. DOI: 10.1016/j.cmet.2012.08.010 |
| [31] | LIZ J, PORTELA A, SOLER M, GÍMEZ A, LING H, MICHLEWSKI G, et al. Regulation of pri-miRNA processing by a long noncoding RNA transcribed from an ultraconserved region[J]. Mol Cell, 2014, 55: 138–147. DOI: 10.1016/j.molcel.2014.05.005 |
| [32] | QUINN J J, ZHANG Q C, GEORGIEV P, ILIK I A, AKHTAR A, CHANG H Y, et al. Rapid evolutionary turnover underlies conserved lncRNA-genome interactions[J]. Genes Dev, 2016, 30: 191–207. DOI: 10.1101/gad.272187.115 |
| [33] | BU D, LUO H, JIAO F, FANG S, TAN C, LIU Z, et al. Evolutionary annotation of conserved long non-coding RNAs in major mammalian species[J]. Sci China Life Sci, 2015, 58: 787–798. DOI: 10.1007/s11427-015-4881-9 |
| [34] | WANG L, PARK H J, DASARI S, WANG S, KOCHER J P, LI W. CPAT:Coding-Potential Assessment Tool using an alignment-free logistic regression model[J/OL]. Nucleic Acids Res, 2013, 41:e74. doi:10.1093/nar/gkt006. |
| [35] | SUN K, CHEN X, JIANG P, SONG X, WANG H, SUN H. iSeeRNA:identification of long intergenic non-coding RNA transcripts from transcriptome sequencing data[J]. BMC Genomics, 2013, 14(Suppl 2): S7. |
| [36] | KONG L, ZHANG Y, YE Z Q, LIU X Q, ZHAO S Q, WEI L, et al. CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine[J/OL]. Nucleic Acids Res, 2007, 35(Web Server issue):W345-W349. doi:10.1093/nar/gkm391. |
| [37] | LIU J, GOUGH J, ROST B. Distinguishing protein-coding from non-coding RNAs through support vector machines[J/OL]. PLoS Genet, 2006, 2:e29. doi:10.1371/journal.pgen.0020029. |
| [38] | FINN R D, BATEMAN A, CLEMENTS J, COGGILL P, EBERHARDT R Y, EDDY S R, et al. Pfam:the protein families database[J]. Nucleic Acids Res, 2014, 42: D222–D230. DOI: 10.1093/nar/gkt1223 |
| [39] | HU W, YUAN B, FLYGARE J, LODISH H F. Long noncoding RNA-mediated anti-apoptotic activity in murine erythroid terminal differentiation[J]. Genes Dev, 2011, 25: 2573–2578. DOI: 10.1101/gad.178780.111 |
| [40] | AUYEUNG V C, ULITSKY I, McGEARY S E, BARTEL D P. Beyond secondary structure:primary-sequence determinants license pri-miRNA hairpins for processing[J]. Cell, 2013, 152: 844–858. DOI: 10.1016/j.cell.2013.01.031 |
| [41] | FINN R D, CLEMENTS J, ARNDT W, MILLER B L, WHEELER T J, SCHREIBER F, et al. HMMER web server:2015 update[J/OL]. Nucleic Acids Res, 2015, 43(W1):W30-W38. doi:10.1093/nar/gkv397. |
| [42] | WASHIETL S, FINDEISS S, MVLLER S A, KALKHOF S, VON BERGEN M, HOFACKER I L, et al. RNAcode:robust discrimination of coding and noncoding regions in comparative sequence data[J]. RNA, 2011, 17: 578–594. DOI: 10.1261/rna.2536111 |
 2017, Vol. 38
2017, Vol. 38


