2. 第二军医大学学员旅学员7队,上海200433
2. The7th Team of Student Brigade,Administration Office for Undergraduate Students,Second Military Medical University, Shanghai 200433, China
寨卡病毒(Zika virus)为单正链RNA病毒,是黄病毒科黄病毒属成员,基因组由10 794个核苷酸组成(MR 766,NC_012532),编码3个结构基因(核衣壳蛋白C、膜蛋白M和包膜蛋白E)和7个非结构基因(NS1、NS2a、NS2b、NS3、NS4a、NS4b和NS5)。寨卡病毒最早于1947年从乌干达维多利亚湖附近的寨卡森林中一只发热的恒河猴身上分离出来[1],然后于1953年在同样地域从非洲伊蚊(Aedes africanus)中再次分离得到,接着在多种蚊子中先后分离出寨卡病毒;与登革热、黄热病、圣路易斯脑炎、西尼罗河脑炎和日本脑炎病毒等黄病毒属病毒一样,寨卡病毒主要通过蚊子传播[2-3]。1964年,乌干达报道了首例寨卡病毒感染人病例,患者出现发热、皮疹、关节和肌肉疼痛、头痛及眶周疼痛[4]。血清学显示寨卡病毒在多个亚洲国家广泛分布[5]。寨卡病毒首次在人群中大规模爆发发生在2007年的西太平洋密克罗尼西亚联邦(Federated States of Micronesia)的雅浦及周围岛屿,约5 000人感染[6],而此前总共只有14例报道;接着在2013—2014年南太平洋的法属波利尼西亚(French Polynesia)发生更大规模的爆发(约3.2万人感染)[7];2015年5月在巴西确诊出首例寨卡病毒感染患者[8],截至2016年2月29日,巴西约有44万至130万感染病例;42个国家有寨卡病毒感染疫情报道,其中23个属美洲国家。自2016年2月9日中国内地确诊首例输入性寨卡病毒感染病例以来,截至3月30日一共确诊了13例,其中江西省1例、广东省8例、浙江省4例。在巴西,研究人员发现寨卡病毒感染可能与小头畸形有关[9-12]。现已在羊水、胎儿脑组织中检测到寨卡病毒,细胞实验也证实寨卡病毒可感染与胎儿脑发育相关的神经干细胞,寨卡病毒和小头症及罕见疾病格林-巴利综合征相关的证据越来越多[13]。但由于寨卡病毒最近几年才发生大规模爆发,之前并未引起人们的重视,目前对寨卡病毒的认识还很缺乏,开发寨卡病毒感染早期诊断技术对我国寨卡病毒防控,尤其对于育龄妇女有效预防新生儿小头畸形具有重要意义。
本研究分析了寨卡病毒基因的序列特点,在此基础上建立了基于反转录-实时荧光定量PCR(RT-qPCR)的寨卡病毒核酸检测方法。RT-qPCR是快速、特异和高敏感性的检测方法,适用于寨卡病毒感染的早期检测,然而到目前为止,只有2篇RT-qPCR文章聚焦于密克罗尼西亚寨卡病毒检测[14-15],没有完全覆盖寨卡病毒的遗传和地域多样性。本研究设计的引物和探针能够检测非洲和亚洲型寨卡病毒毒株。
1 材料和方法 1.1 虫媒传播黄病毒及寨卡病毒系统进化树的构建使用最大似然法构建虫媒传播黄病毒和寨卡病毒系统进化树,病毒株间的距离使用位点替换/总位点评估[16]。使用Neighbor-Join和BioNJ算法计算进化树分支,多次计算统计分支生成概率,评估进化树分支可信度。所有计算使用MEGA7软件完成[17],用于系统进化树构建的虫媒传播黄病毒毒株基因组序列(GenBank ID)见表 1。
|
|
表 1 黄病毒系统发生树构建所用的病毒株 Tab 1 Flavivirus virus strains used for phylogenetic tree |
1.2 引物和探针设计
从NCBI数据库中获得有全基因组序列的寨卡病毒毒株25株,去掉同源性近的14株,选择有地区代表性的11株寨卡病毒毒株筛选保守序列。利用Clustal W程序进行比对获得保守序列[18],利用Primer Express软件进行引物和探针设计[19],探针5'端设计有荧光报告基团FAM,3'端基团为BHQ。
1.3 病毒RNA提取和RT-qPCR细胞培养的寨卡病毒SZ01株(GenBank No.KU866423)由军事医学科学院微生物与流行病研究所秦成峰研究员馈赠。参照Invitrogen病毒核酸提取试剂盒(cat:12280050)说明书提取寨卡病毒、登革病毒和日本脑炎病毒RNA。用ABI公司的One step plus型PCR仪进行RT-qPCR检测,采用TaKaRa PCR扩增试剂,20 μL体系(10 μL Mixture,0.4 μL ROX染料,正、反向引物各0.4 μL,探针0.8 μL,7.2 μL无RNA酶水,0.8 μL模板)。反应条件:第一阶段42℃ 5 min,95℃ 10 s;第二阶段(40个循环)95℃ 5 s,60℃ 34 s。
1.4 敏感性和特异性检测利用细胞培养的寨卡病毒和含检测靶基因的质粒标准品对引物和探针的敏感性进行评估。质粒标准品是通过将合成的寨卡病毒NS2E基因序列(97 bp)插入载体pUC19而获得,用紫外分光光度法检测质粒浓度。为评估引物和探针的检测最低拷贝数,将质粒(浓度2.1×1010拷贝/mL)以10倍梯度稀释为10-1、10-2、10-3、10-4、10-5、10-6、10-7、10-8、10-9、10-10 共10个浓度梯度分别检测。利用细胞培养的登革病毒1~4型和日本脑炎病毒评估引物和探针的特异性。
2 结果 2.1 寨卡病毒与其他虫媒传播黄病毒序列比较图 1A 是81种虫媒传播黄病毒的进化树,最顶层是2个大分支,一个分支以日本脑炎、Ntaya、Aroa、登革病毒等构成的蚊传播(mosquito-borne)组成;另一分支比较杂乱,包括蜱传播(tick-borne)、未知传播媒介和部分未知节肢动物宿主的蚊传播黄病毒。寨卡病毒属于蚊传播黄病毒分支,与Spondweni、Kedougou病毒同源性最近,其次是登革病毒。寨卡病毒与Spondweni病毒的基因C、M、E、NS2a、NS2b、NS3、NS4a、P2k、NS4b和NS5氨基酸序列同源性分别为65.57%、63.69%、71.80%、73.58%、59.73%、76.15%、80.55%、75.82%、79.44%和77.85%,而与登革病毒4型的同源性分别为39.34%、45.24%、55.40%、53.98%、25.22%、38.46%、66.61%、43.14%、49.19%和67.66%。

|
图 1 虫媒传播黄病毒进化树(A)和寨卡病毒、登革病毒4型、日本脑炎病毒核酸(B)及氨基酸(C)序列比较 Fig 1 Phylogenetic tree of the genus Flavivirus (A) and nucleic acid (B),and amino acid (C)overlaps of complete genome of ZIKV, DENV 4 and JEV |
登革病毒和日本脑炎病毒是目前研究较成熟的虫媒病毒,本研究寨卡病毒与登革病毒4型、日本脑炎病毒的全基因组核酸和氨基酸序列进行比较。全基因组核酸序列比较结果显示,寨卡病毒、登革病毒4型和日本脑炎病毒都一致的核酸位点为4 463个,占总数的43.14%。寨卡病毒与登革病毒4型、日本脑炎病毒相同的核酸位点分别为6 139和6 017个,占总数的59.34%和58.16%;寨卡病毒与登革病毒4型的同源性略高于日本脑炎病毒(图 1B)。氨基酸序列比较结果显示,寨卡病毒、登革病毒4型和日本脑炎病毒都一致的氨基酸位点为1 460个,占氨基酸总数的42.44%。寨卡病毒与登革病毒4型、日本脑炎病毒相同的氨基酸位点分别为1 920和1 936个,占氨基酸总数的55.81%和56.28%;寨卡病毒与日本脑炎病毒的同源性略高于登革病毒4型(图 1C)。进一步对单个基因分析,寨卡病毒的C、E、NS1、NS3、NS4a和NS5基因与日本脑炎病毒同源性更高,而M、NS2a、NS2b和NS4b与登革病毒同源性更高。3种病毒公共部分的核酸或氨基酸位点(图 1B、1C d区)可能决定着三者共有的生物学特性,如蚊传播。而寨卡病毒与登革病毒共有部分的核酸或氨基酸位点(b区)可能决定着两者特有的生物学特性,如伊蚊传播、诱发皮疹等;g区的核酸或氨基酸位点可能决定寨卡病毒与日本脑炎病毒特有的生物学特性,如对神经细胞敏感等;a、c和f区则分别是登革病毒、寨卡病毒与日本脑炎病毒三者各自特有的生物学特性。
2.2 寨卡病毒的进化及变异特征寨卡病毒进化树分成明显的2个大分支,分别对应亚洲型和非洲型(图 2A)。寨卡病毒亚洲型与非洲型存在82个氨基酸差异位点,其中PrM基因氨基酸位点突变率最高(6.45%),包膜基因EP(1.00%)和NS4a(0.67%)最保守。变异氨基酸位点见表 2,其中每个位点的左侧为亚洲型氨基酸,右侧为非洲型。

|
图 2 寨卡病毒系统进化树(A)及我国江西赣县株氨基酸序列变异位点(B) Fig 2 Zika virus phylogenetic tree (A) and the variability of VE Ganxian Zika virus (B) |
|
|
表 2 寨卡病毒亚洲型相比非洲型的氨基酸突变位点 Tab 2 The variability of amino acid mutations in Asian and African Zika virus |
NCBI核酸数据库中可获得我国研究人员提交的4株寨卡病毒全基因组序列,都属于亚洲型。广东GD01、GDZ16001毒株序列接近,只存在8个核酸差异位点,与巴西(KU365778)和波多黎各(KU501215)病毒株同源性最高。广东GD01株相比以往亚洲型毒株存在5个氨基酸位点变化(D107E、D445G、I1285V、T2749I和V2787A),而广东GDZ16001株有3个氨基酸位点差异,与广东GD01株的1285、2749和2787位点变异完全相同。浙江ZJ03毒株和2013年在波利尼西亚爆发的毒株同源性最高,与该患者在前往萨摩亚旅行时被感染的事实吻合,该毒株相比以往亚洲型毒株存在6个氨基酸位点变化(S109N、K709R、 R1118Q、 V2634M、N3144S和K3190R)。赣县VE Ganxian株与2014年海地(Haiti)株同源性最近,但已存在较高突变,相比以往亚洲型和非洲型毒株其氨基酸变异位点高达33个,占寨卡病毒总氨基酸数的0.96%。图 2B展示了赣县株与其他亚洲株、非洲株在33个变异位点的氨基酸情况,在这33个位点中,赣县株的位点特异。
2.3 RT-qPCR引物和探针设计比较11株寨卡病毒毒株(8株亚洲型和3株非洲型),选择保守序列,获得1组引物和探针(Zika-3990F: 5'-CCA CGC ACT GAT AAC AT-3',Zika-4068R: 5'-AAG TAG CAA GGC CTG CTC T-3',Zika-4021probe: 5'-FAM-TGG CTG CTC TRA CAC CAC T-BHQ-3'),位于NS2a基因。正、反向引物和探针与Spondweni病毒、登革病毒1~4型序列都存在很高的差异(图 3),表明设计的引物和探针均具有特异性。

|
图 3 寨卡病毒核酸检测引物和探针序列 Fig 3 Primers and probe of Zika virus |
2.4 RT-qPCR检测方法的敏感性和特异性
对各稀释梯度的标准品(最高浓度2.1×109拷贝/mL)进行RT-qPCR检测,前8个浓度检测结果为阳性;后2个梯度质粒样本检测Ct值大于35,认为阴性。对Ct值和质粒拷贝浓度进行拟合,获得标准曲线(图 4),检测限约100拷贝/mL。模拟寨卡病毒血清样本检测为阳性,登革病毒1~4型和日本脑炎病毒检测均为阴性,表明该检测方法具有很高的特异性。
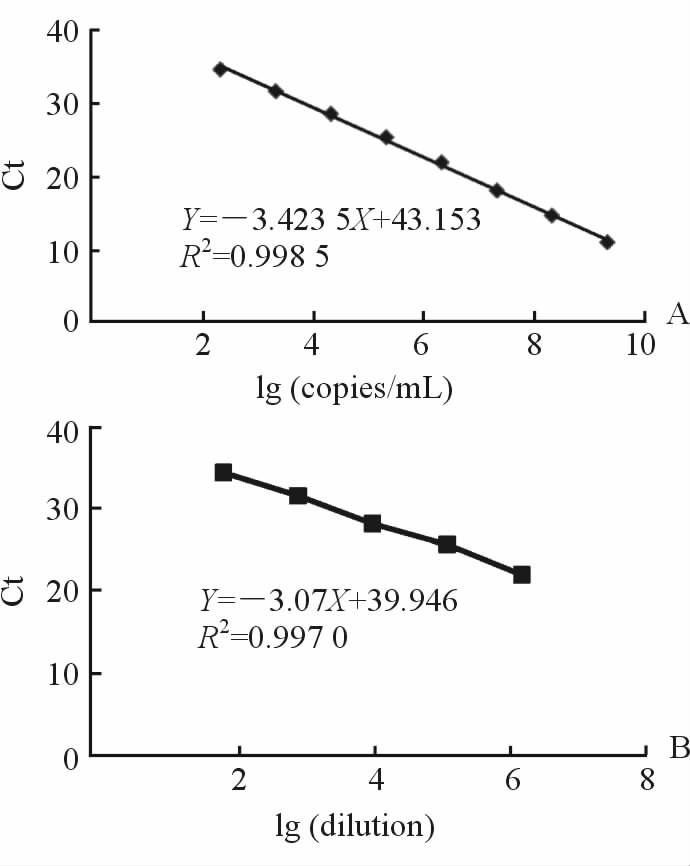
|
图 4 寨卡病毒核酸定量PCR检测标准曲线 Fig 4 Standard curve of RT-qPCR for the nucleic acid detection for Zika virus |
3 讨论
目前有关寨卡病毒病原学研究的报道较少,通过与其他虫媒传播黄病毒的基因序列比较可在一定程度推测寨卡病毒的生物学特性。本研究首先构建了81种虫媒传播黄病毒的系统发生树,明确了寨卡病毒同源性最近的病毒,并重点比较了寨卡病毒与登革病毒4型、日本脑炎病毒的基因组序列差异。Kuno等[20]最早对虫媒传播黄病毒系统进化进行研究,并构建了71株虫媒传播黄病毒的系统进化树,结果显示Kedougou病毒为独立一株,与寨卡病毒遗传距离较远;而寨卡病毒与日本脑炎病毒同源性更近,与登革病毒遗传距离较远,与本研究结果存在一定差别。[JP2]分析产生差异的原因可能是Kuno等[20]构建系统发生树采用的是NS5基因,寨卡病毒NS5基因与日本脑炎病毒氨基酸相同位点的百分比为68.55%,而与登革病毒为67.66%。本研究结果显示由于寨卡病毒与登革病毒、日本脑炎病毒的遗传差异比较接近,以不同的基因、核酸或蛋白质序列构建系统进化树都可能产生不同结果。但可以明确的一点是,日本脑炎病毒和登革病毒之间的遗传距离是寨卡病毒、登革病毒和日本脑炎病毒三者之间最远的。
截至2016年3月30日,中国共检测出13例寨卡病毒感染病例,对最早发现的4株寨卡病毒毒株进行了全基因组测序,结果发现广东GD01和GDZ16001毒株之间存在8个核酸位点差异,与以往亚洲型毒株相比GDZ16001株存在3个氨基酸变异,而GD01株有5个氨基酸位点变异,其中3个与GDZ16001株的氨基酸变异位点相同,表明GD01很可能是由GDZ16001变异产生。浙江ZJ01株有6个氨基酸变异位点,且与广东株的5个位点完全不同,是由2013年波利尼西亚毒株变异而来,与当前南美正流行的毒株也不同,表明萨摩亚寨卡病毒流行跟南美流行是独立发生的。广东毒株和赣县毒株的感染患者都是来自委内瑞拉的工作人员,说明委内瑞拉已存在氨基酸序列差异较大的毒株。相比亚洲和波利尼西亚毒株,江西赣县株与2013年在南美爆发的多个毒株同源性更近,表明赣县株是南美毒株变异的结果。相比2007年亚洲型毒株对非洲型毒株的仅仅82个变异位点,赣县株高达33个氨基酸位点的突变表明最近3年寨卡病毒在南美正经历快速变异。寨卡病毒在人群中的大规模爆发正加速寨卡病毒的变异以更好地适应新宿主,其变异位点的功能值得重点关注。但寨卡病毒本身变异是否为本次寨卡病毒大爆发的原因,或其引发原因是否为拉美地区自然环境,均还未知。
本研究设计的RT-qPCR 引物和探针具有更高的通用性,与登革病毒1~4型和日本脑炎病毒无交叉,能高灵敏地检测出细胞培养的寨卡病毒,为寨卡病毒感染的诊断提供了一种有效方法。
| [1] | DICK G W, KITCHEN S F, HADDOW A J. Zika virus. Ⅰ. Isolations and serological specificity. Trans R Soc Trop Med Hyg[J]. 1952, 45 :509–520 . |
| [2] | MARCHETTE N J, GARCIA R, RUDNICK A. Isolation of Zika virus from Aedes aegypti mosquitoes in Malaysia. Am J Trop Med Hyg[J]. 1969, 18 :411–415 . |
| [3] | BOORMAN J P, PORTERFIELD J S. A simple technique for infection of mosquitoes with viruses; transmission of Zika virus. Trans R Soc Trop Med Hyg[J]. 1956, 50 :238–242 . |
| [4] | SIMPSON D I. Zika virus infection in man. Trans R Soc Trop Med Hyg[J]. 1964, 58 :335–338 . |
| [5] | SALUZZO J F, IVANOFF B, LANGUILLAT G, GEORGES A J. Serological survey for arbovirus antibodies in the human and simian populations of the South-East of Gabon (author's transl. Bull Soc Pathol Exot Filiales[J]. 1982, 75 :262–266 . |
| [6] | DUFFY M R, CHEN T H, HANCOCK W T, POWERS A M, KOOL J L, LANCIOTTI R S, et al. Zika virus outbreak on Yap Island, Federated States of Micronesia. N Engl J Med[J]. 2009, 360 :2536–2543 . |
| [7] | CAO-LORMEAU V M, ROCHE C, TEISSIER A, ROBIN E, BERRY A L, MALLET H P, et al. Zika virus, French Polynesia, South Pacific, 2013. Emerg Infect Dis[J]. 2014, 20 :1085–1086 . |
| [8] | CAMPOS G S, BANDEIRA A C, SARDI S I. Zika virus outbreak, Bahia, Brazil. Emerg Infect Dis[J]. 2015, 21 :1885–1886 . |
| [9] | HEYMANN D L, HODGSON A, SALL A A, FREEDMAN D O, STAPLES J E, ALTHABE F, et al. Zika virus and microcephaly: why is this situation a PHEIC?. Lancet[J]. 2016, 387 :719–721 . |
| [10] | LIUZZI G, PURO V, VAIRO F, NICASTRI E, CAPOBIANCHI M R, DI CARO A, et al. Zika virus and microcephaly: is the correlation, causal or coincidental?. New Microbiol[J]. 2016, 39 :83–85 . |
| [11] | MARRS C, OLSON G, SAADE G, HANKINS G, WEN T, PATEL J, et al. Zika virus and pregnancy: a review of the literature and clinical considerations. Am J Perinatol[J]. 2016, 33 :625–639 . |
| [12] | RUBIN E J, GREENE M F, BADEN L R. Zika virus and microcephaly. N Engl J Med[J]. 2016, 374 :984–985 . |
| [13] | ROZÉ B, NAJIOULLAH F, FERGÉ J L, APETSE K, BROUSTE Y, CESAIRE R, et al. Zika virus detection in urine from patients with Guillain-Barre Syndrome on Martinique, January 2016. Euro Surveill[J]. 2016, 21 . |
| [14] | FAYE O, DIALLO D, DIALLO M, WEIDMANN M, SALL A A. Quantitative real-time PCR detection of Zika virus and evaluation with field-caught mosquitoes. Virol J[J]. 2013, 10 :311. |
| [15] | LANCIOTTI R S, KOSOY O L, LAVEN J J, VELEZ J O, LAMBERT A J, JOHNSON A J, et al. Genetic and serologic properties of Zika virus associated with an epidemic, Yap State, Micronesia, 2007. Emerg Infect Dis[J]. 2008, 14 :1232–1239 . |
| [16] | TAMURA K, NEI M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol[J]. 1993, 10 :512–526 . |
| [17] | TAMURA K, STECHER G, PETERSON D, FILIPSKI A, KUMAR S. MEGA6: molecular evolutionary genetics analysis version 6. Mol Biol Evol[J]. 2013, 30 :2725–2729 . |
| [18] | LARKIN M A, BLACKSHIELDS G, BROWN N P, CHENNA R, MCGETTIGAN P A, MCWILLIAM H, et al. Clustal W and Clustal X version 2. Bioinformatics[J]. 2007, 23 :2947–2948 . |
| [19] | YOON J R, LAIBLE P D, GU M, SCOTT H N, COLLART F R. Express primer tool for high-throughput gene cloning and expression. Biotechniques[J]. 2002, 33 :1328–1333 . |
| [20] | KUNO G, CHANG G J, TSUCHIYA K R, KARABATSOS N, CROPP C B. Phylogeny of the genus Flavivirus. J Virol[J]. 1998, 72 :73–83 . |
 2016, Vol. 37
2016, Vol. 37


