肝细胞癌(hepatocellular carcinoma,HCC)的发病机制复杂,涉及到不同信号通路的基因组变异和表观遗传的改变[1-2]。基因组变异包括基因突变、拷贝数变异和染色体易位。拷贝数变异(copy number variation,CNV)是基因组在结构上发生变异的一种形式,指染色体上≥1 kb DNA片段上的复制、插入、缺失和复杂变异。拷贝数变异主要来源于DNA重组,包括非等位同源重组(non-allelic homologous recombination,NAHR)和非同源末端连接(non-homologous end-joining,NHEJ)[3]。在肿瘤发生过程中,凋亡相关基因的拷贝数通常发生减少,而增殖相关基因的拷贝数则发生扩增[4]。在HCC中,乙型肝炎病毒(hepatitis B virus,HBV)通过将病毒DNA整合进入宿主基因组中,可以导致CNV的发生,造成染色体的不稳定,并最终导致肝癌发生[5]。因而,从CNV角度入手研究肝癌,可以加深对肝癌发病机制的精确认识。同时,由于CNV的发生是一个长期累积效应,也为运用基因靶技术进行及早诊断和治疗肝癌提供了新的思路[6]。
目前,已知在HCC中CNV发生扩增的高频片段位于染色体1q、6p、8q、17q和20p,而发生缺失的高频片段位于染色体4q、6q、8p、9p、13q、14q、16q和17p[7-8]。这些CNV高频片段上是否存在与HCC发生相关的基因、发挥何种作用目前还不清楚。通过前期工作,我们从芯片中筛选得到了在肝癌与癌旁组织中存在差异表达的基因。将这些基因与CNV高频片段进行整合分析,发现其中位于CNV扩增高频区段8q24.3上的蛋白编码基因PPP1R16A在肝癌组织的表达高于癌旁组织(上调倍数1.76,P=0.03)[9],提示其在肝癌发生中具有重要作用,可能发挥促癌的功能。本研究拟通过实时荧光定量PCR技术,验证106对肝癌组织和癌旁组织PPP1R16A的拷贝数和转录表达情况及两者的相关性;并通过siRNA技术体外靶向沉默肝癌细胞株HCC LM3中PPP1R16A的表达,探索对HCC LM3细胞增殖和凋亡的影响。
1 材料和方法 1.1 细胞和组织标本人肝癌细胞株HCC LM3购自中国科学院细胞库,由本实验室保存。细胞用含10%胎牛血清的DMEM培养基,置于37℃、5%CO2、饱和湿度的培养箱中培养。106例肝癌患者的肿瘤组织和癌旁组织、41例正常肝组织、22例肝炎组织以及患者资料由第二军医大学东方肝胆外科医院提供,并取得患者或其家属知情同意。所有组织标本取得后立即放入液氮中保存备用。
1.2 主要材料和试剂DMEM高糖培养基购自Hyclone公司,胎牛血清购自Gibco公司,LipofectamineTM 3000试剂购自Invitrogen公司,基因组DNA抽提试剂盒购自Axygen公司,RNA抽提试剂TRIzol RNAiso购自TaKaRa公司,反转录试剂购自Invitrogen公司,CCK-8试剂盒购自Dojindo公司,Annexin Ⅴ-FITC/PI细胞凋亡检测试剂盒购自美国BD公司,细胞周期检测试剂盒购自碧云天生物技术有限公司,蛋白酶抑制剂(Cocktail)购自Sigma公司,硝酸纤维素(NC)膜购自Millipore公司,Anti-PPP1R16A抗体购自Sigma公司。
1.3 基因组DNA抽提对于组织样本,取20 mg肝癌或肝组织,移入冰水浴预冷的研钵中,快速研磨成匀浆。加入350 μL PBS和0.9 μL RNase A后温和地研磨30 s。收集350 μL研磨好的组织匀浆并转入2 mL离心管。如匀浆体积不足350 μL,补充PBS至350 μL。加入150 μL Buffer C-L和20 μL Proteinase K。立即涡旋振荡1 min 混合均匀。短暂离心后,将离心管置于56℃水浴10 min。加入350 μL Buffer P-D,涡旋振荡30 s混合均匀,12 000×g离心10 min。将DNA制备管置于2 mL离心管中,将上一步的混合液移至制备管中,12 000×g离心1 min。弃滤液,将制备管置回到原来的2 mL离心管中,加入500 μL Buffer W1,12 000×g离心1 min。再用700 μL Buffer W2 洗涤2次。最后用150 μL去离子水洗脱DNA。
1.4 细胞转染PPP1R16A siRNA序列:正义链5′-GCG GCA UCU AUA CUC CAA GTT-3′,反义链5′-CUU GGA GUA UAG AUG CCG CTT-3′,由上海吉玛公司合成。非特异性siRNA由吉玛公司提供。利用LipofectamineTM3000试剂将PPP1R16A siRNA和非特异性siRNA转染HCC LM3细胞,即si-16A组和NC组;未转染siRNA的HCC LM3为空白对照组。在125 μL DEPC水中加入2.5 nmol siRNA,溶解后为20 μmol/L的母液。转染前1 d,细胞传代培养于6孔板中,培养24 h至细胞铺满孔底的60%~70%。将5 μL 20 μmol/L的siRNA与125 μL的opti-MEM混合于无菌的Eppendorf管中。取3.75 μL 的LipofectamineTM 3000试剂与125 μL 的opti-MEM混合于另一无菌的Eppendorf管中。将上述两管液体混合均匀,室温静置至少5 min,形成siRNA-脂质体复合物,随后逐滴加入细胞培养基中。将细胞瞬时转染48 h后,收集细胞用于所需实验。
1.5 总RNA的抽提对于组织样本,每50~100 mg肝癌组织或肝组织标本在液氮中研磨粉碎,待液氮挥发后迅速加入1 mL TRIzol;对于细胞样本,按照6孔板每个培养孔添加1 mL TRIzol的比例加入TRIzol试剂直接裂解细胞。1 mL TRIzol试剂中加入200 μL氯仿,剧烈震荡15 s混匀,室温放置5 min。12 000×g 4℃离心15 min。样本形成上层的无色水相、中间层和下层的红色苯酚-氯仿相的三相分层状态。小心吸取上层的无色水相至新的Eppendorf管中,等体积加入约0.5 mL异丙醇,轻微混匀后将样本在室温下孵育10 min,12 000×g 4℃离心10 min。弃上清,以75%乙醇洗涤RNA沉淀2次,每次7 500×g 4℃离心5 min。冰上短暂干燥RNA(4℃干燥5 min左右)。根据沉淀体积以RNase-free的水溶解RNA,以枪头吹打数次。RNA保存于-80℃。使用Nanodrop仪器和软件,测定并计算RNA的D260/D280的比值,判断总RNA质量并进行定量。
1.6 引物的设计与合成PPP1R16A基因引物序列:上游5′-CGG AAT TCA TGG CCG AGC ACC TGG-3′,下游5′-CGG GAT CCT CAC ATG AGC AGG CAG CAC-3′;内参基因GAPDH引物序列:上游5′-CGG ATT TGG TCG TAT TGG G-3′,下游5′-CTG GAA GAT GGT GAT GGG ATT-3′。所有引物均由生工生物工程(上海)股份有限公司合成。
1.7 实时荧光定量PCR(qRT-PCR)与相对定量分析实时荧光定量PCR实验使用Applied Biosystems 公司的StepOneTM Real-Time PCR System 系统。模板为反转录所得的cDNA和稀释后的gDNA。每个反应重复3次,每次反应均设3个复孔,并均包含了相应的无模板对照。定量结果采用相对定量的方法(2-ΔΔCt法)进行比较、分析。
1.8 蛋白质印迹法检测LM3细胞中PPP1R16A的表达细胞转染48 h后提取各组细胞总蛋白,BCA法测定蛋白浓度。根据蛋白定量结果,调整蛋白上样量,每个样品取20 μg加入上样孔,进行SDS-PAGE(5%浓缩胶:50 V,40 min;12%分离胶:100 V,100 min)。电泳结束后根据蛋白大小进行切胶,并切取同样大小的NC膜,按照阴极板-滤纸-SDS-PAGE胶-NC膜-滤纸-阳极板的顺序,以100 V 进行电转。电转完成后,用5%脱脂奶粉进行封闭,置于室温,放置2 h。加入一抗4℃孵育过夜,加入二抗室温孵育2 h。蛋白条带使用Li-Cor公司的Odyssey infrared scanner 进行扫描、定量。
1.9 CCK-8细胞增殖实验将干扰PPP1R16A的HCC LM3细胞及其对照组细胞消化后等密度重新铺于96孔板中,每种细胞铺5个复孔,每孔3 000个细胞,100 μL完全培养基培养。待生长6 h细胞贴壁后,按10 μL/孔加入CCK-8试剂,分别在试剂加入后不同的时间点(0、12、24、36、48、60 h)使用酶标仪测定D450,绘制细胞的增殖曲线,根据增殖曲线比较不同组的细胞增殖情况。
1.10 平板克隆形成实验取对数生长期的细胞,用0.25% 胰蛋白酶消化吹打成单个细胞,把细胞悬浮在含10%胎牛血清的DMEM培养液中备用。按每孔3 000个细胞接种于6孔板中,并轻轻转动,使细胞分散均匀。置于37℃、5%CO2及饱和湿度的环境下,静置培养2周。当培养皿中出现肉眼可见的克隆时,终止培养。弃上清液,用PBS浸洗2次,加入4%多聚甲醛,固定20 min。然后去固定液,按每孔1 mL加入0.5%结晶紫染色液染色1.5 h,然后用PBS洗涤多次,去除染色液,空气干燥。
1.11 流式细胞术检测细胞周期细胞转染48 h后,用胰酶消化细胞,离心收集,1 000×g离心5 min。吸除上清,加入1 mL冰浴预冷的PBS,重悬细胞。再次离心,吸除上清后,加入1 mL冰浴预冷的70%乙醇,轻轻吹打混匀,4℃固定12~24 h。1 000×g离心5 min,沉淀细胞。小心吸除上清后,加入1 mL冰浴预冷的PBS,重悬细胞。每管样品加入0.5 mL PI染色液[0.5 mL染色缓冲液,25 μL PI染色液(20×),10 μL RNase A (50×)],缓慢并充分重悬细胞沉淀,37℃温浴30 min后,经流式细胞仪检测并分析细胞周期。
1.12 统计学处理采用SPSS 18.0统计软件进行数据分析。定量资料用x±s表示。肝癌与配对癌旁组织的比较采用Wilcoxon符号秩检验,多组间均值的比较采用单因素方差分析,两因素的相关性采用Pearson相关分析。检验水准(α)为0.05。
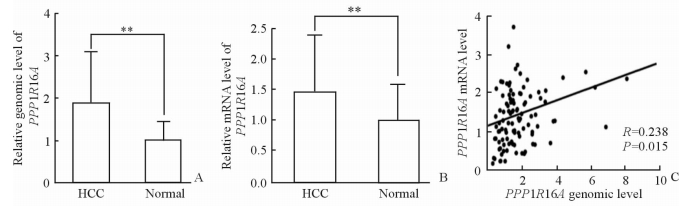
2 结 果 2.1 临床样本中PPP1R16A 基因组和转录水平的变化在基因组DNA水平,PPP1R16A在106对肝癌组织和癌旁组织中的相对量分别为1.87±1.32和1.00±0.45,差异有统计学意义(P<0.01),证实肝癌组织中PPP1R16A的拷贝数发生了扩增。在转录水平,PPP1R16A在肝癌和癌旁组织的相对表达量分别为1.44±0.92和1.00±0.64,差异有统计学意义(P<0.01),证实PPP1R16A的转录表达也发生了增加。对PPP1R16A基因组DNA和转录水平进行相关性分析,两者的Pearson相关系数R=0.238(P=0.015),说明PPP1R16A 转录水平的上调与拷贝数的扩增存在正相关(图 1A~1C)。
|
图 1 肝癌组织中PPP1R16A的拷贝数发生扩增且转录水平增加 Fig 1 The genomic and transcription level of PPP1R16A gene were increased in HCC tissues A: The genomic level of PPP1R16A gene in HCC tissues and paired adjacent normal liver tissues (**P<0.01. n=106, x±s ); B: The transcription level of PPP1R16A gene in HCC tissues and paired adjacent normal liver tissues (**P<0.01. n=106, x±s ); C: The correlation between genomic level and transcription level of PPP1R16A gene (n=106). HCC: Hepatocellular carcinoma |
2.2 PPP1R16A 基因组水平与肝癌进程的关系
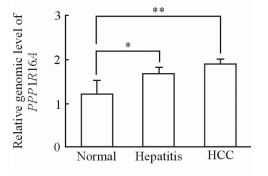
对41例正常肝组织、22例肝炎组织以及106例肝癌组织中PPP1R16A基因组水平进行比较,结果发现,随着肝癌的发病过程,PPP1R16A的基因组水平也在逐渐增加。PPP1R16A在肝癌组织中的基因组水平高于正常肝组织(P<0.01),肝炎组织中的基因组水平也高于正常肝组织(P<0.05)。肝癌组织与肝炎组织中的基因组水平差异无统计学意义(P>0.05,图 2)。说明随着肝癌的发展,PPP1R16A基因的拷贝数也逐渐递增。
|
图 2 PPP1R16A的拷贝数水平随着肝癌的进展逐渐增加 Fig 2 The genomic level of PPP1R16A gene increased with HCC progression HCC: Hepatocellular carcinoma. Normal liver tissues, n=41; Hepatitis tissues, n=22; HCC tissues, n=106. * P<0.05, ** P<0.01. x±s |
2.3 siRNA转染HCC LM3细胞对PPP1R16A基因转录和翻译的影响
实时荧光定量PCR结果(图 3A)显示,转染PPP1R16A siRNA后,si-16A组PPP1R16A基因的相对表达量低于NC组和空白组,差异均有统计学意义(P<0.001);NC组和空白组相比基因表达差异无统计学意义(P>0.05)。蛋白质印迹实验结果(图 3B)显示,si-16A组PPP1R16A蛋白表达量(0.06±0.05)低于NC组(2.81±0.71)和空白组(2.98±0.51),差异均有统计学意义(P<0.01);NC组与空白组蛋白表达差异没有统计学意义(P>0.05)。说明PPP1R16A siRNA能够有效抑制其基因的转录和翻译。

|
图 3 靶向沉默PPP1R16A基因抑制HCC LM3细胞PPP1R16A基因转录和翻译 Fig 3 Inhibition on PPP1R16A transcription and translation in HCC LM3 cells transfected with PPP1R16A siRNA A: The transcription level of PPP1R16A gene in HCC LM3 cells (** P<0.01. n=3, x±s ); B: The expression level of PPP1R16A protein in HCC LM3 cells. si-16A: HCC LM3 cells transfected with PPP1R16A siRNA; NC: HCC LM3 cells transfected with non-specificity siRNA; Blank: HCC LM3 cells untreated with siRNA |
2.4 靶向沉默PPP1R16A 对HCC LM3细胞增殖能力和细胞周期的影响
克隆形成实验结果(图 4A、4B)显示,si-16A组克隆形成率低于NC组和空白组,差异有统计学意义(P<0.01)。CCK-8细胞增殖实验结果(图 4C)显示,随着时间的延长,si-16A组细胞增殖明显受到了抑制,在60 h时si-16A组D450(1.43±0.05)较NC组(3.17±0.15)和空白组(2.61±0.17)低(P<0.01),说明转染siRNA后,HCC LM3细胞增殖被抑制。细胞周期检测结果(图 4D)显示,si-16A组较NC组和空白组处于G0/G1期的细胞比例增加,处于S期和G2/M期的细胞比例减少。说明转染PPP1R16A siRNA后HCC LM3细胞周期发生了阻滞。

|
图 4 靶向沉默PPP1R16A基因抑制HCC LM3细胞的增殖并阻滞细胞周期 Fig 4 Transfection of PPP1R16A siRNA inhibited HCC LM3 cell proliferation and induced G0/G1 arrest in HCC LM3 cells A: Clone formation assay of HCC LM3 cells transfected with PPP1R16A siRNA; B: Analysis of clone formation assay of HCC LM3 cells (** P<0.01. n=3 , x±s ); C: CCK-8 analysis of HCC LM3 cells transfected with PPP1R16A siRNA (**P<0.01 vs NC and blank control. n=3, x±s ); D: Cell cycle of HCC LM3 cells analyzed by flow cytometry |
3 讨 论
本课题前期通过芯片分析发现,PPP1R16A肝癌组织中的表达异常增高[9]。同时,该基因位于拷贝数扩增的高频区段——8号染色体长臂2区4带3亚带内。基于上述结果,我们推测,在肝癌组织中,由于拷贝数发生了扩增,导致区段内基因PPP1R16A的表达增加。故我们首先在106例临床样本中对其基因组水平和转录水平进行验证,我们证实PPP1R16A基因在肝癌组织中的拷贝数发生了扩增,PPP1R16A的转录表达增加,并且拷贝数的扩增与基因转录水平的上调呈正相关。
在对肝癌发病各阶段肝脏组织(正常肝、肝炎和肝癌组织)中PPP1R16A的拷贝数水平进行比较后,我们观察到,随着疾病的进展,PPP1R16A的拷贝数水平也随之增加。说明PPP1R16A拷贝数的扩增与肝癌的发生密切相关,并提示其可以作为评判肝癌发病风险的指标。进一步通过检索Oncomine数据库中的芯片数据发现,在基因组DNA水平上PPP1R16A在肝癌组织(n=26)中的表达高于正常肝组织(n=26),差异有统计学意义(P<0.001)(GSE32649)[10];在转录水平,PPP1R16A在肝癌组织(35例)中的表达高于肝癌前体组织(肝硬化组织13例,不典型增生组织17例),差异有统计学意义(P<0.001)(GSE6764)[11]。
PPP1R16A是蛋白磷酸酶(protein phosphatase 1,PP1)的调节亚基[12]。目前已知,PP1参与了糖原代谢、基因转录、蛋白合成、细胞分裂和细胞凋亡等诸多生理过程[13-14]。据报道,PP1的表达失调与肿瘤细胞的增殖和凋亡密切相关[15]。在靶向沉默PPP1R16A后,CCK-8细胞增殖实验和克隆形成实验显示,HCC LM3细胞的增殖受到了明显的抑制(P<0.01);细胞周期和凋亡检测结果显示,si-16A组细胞发生了G0/G1阻滞,而细胞凋亡没有受到影响。这一结果提示,PPP1R16A对细胞增殖的抑制作用主要体现在对细胞周期的阻滞。同时,我们还在肝癌细胞Huh7中观察了干扰PPP1R16A对细胞增殖的影响,结果显示,靶向沉默PPP1R16A并不能抑制Huh7细胞的增殖(资料未发表)。针对这一现象,我们对涉及HCC LM3和Huh7细胞研究的相关文献进行检索,发现这两种细胞的差异之一,体现在其p53突变状态的不同。其中,HCC LM3中的p53为野生型(wild-type),Huh7中p53为突变型(mutant)[16]。当癌基因激活或DNA发生损伤时,p53可以通过抑制细胞周期和促进细胞凋亡而发挥抑癌作用[17]。p53参与多种基因的转录激活或抑制[18],其自身也受到磷酸化[19]、乙酰化[20]和泛素化[21]等多种翻译后的修饰调节[22]。野生型p53的磷酸化可以增强其自身的稳定性,并在核内聚集,有利于其发挥抑癌作用,而突变型p53通过磷酸化作用,则会增强其促癌的功能[23]。根据以上结论,我们推测p53的突变状态以及磷酸化修饰可能是造成不同细胞增殖的差异表现的原因。
综上所述,本研究初步发现,在肝癌组织中,PPP1R16A基因拷贝数发生扩增,转录水平上调,且与肝癌发病密切相关。通过siRNA干扰基因的表达,可以抑制肝癌HCC LM3细胞的增殖。提示PPP1R16A基因在肝癌中发挥癌基因的作用。后续还将探讨HCC中影响PPP1R16A拷贝数发生扩增的因素有哪些、能否通过人为干预降低CNV的频率并降低肝癌的发病风险等,为临床预防、诊断和治疗HCC提供参考。
| [1] | Zucman-Rossi J, Villanueva A, Nault J C, Llovet J M. Genetic landscape and biomarkers of hepatocellular carcinoma. Gastroenterology[J]. 2015, 149 :1226–1239 . |
| [2] | Jhunjhunwala S, Jiang Z, Stawiski E W, Gnad F, Liu J, Mayba O, et al. Diverse modes of genomic alteration in hepatocellular carcinoma. Genome Biol[J]. 2014, 15 :436. |
| [3] | Zhang F, Khajavi M, Connolly A M, Towne C F, Batish S D, Lupski J R. The DNA replication FoSTeS/MMBIR mechanism can generate genomic, genic and exonic complex rearrangements in humans. Nat Genet[J]. 2009, 41 :849–853 . |
| [4] | Mauro J A, Butler S N, Ramsamooj M, Blanck G. Copy number loss or silencing of apoptosis-effector genes in cancer. Gene[J]. 2015, 554 :50–57 . |
| [5] | Neuveut C, Wei Y, Buendia M A. Mechanisms of HBV-related hepatocarcinogenesis. J Hepatol[J]. 2010, 52 :594–604 . |
| [6] | Xu H, Zhu X, Xu Z, Hu Y, Bo S, Xing T, et al. Non-invasive analysis of genomic copy number variation in patients with hepatocellular carcinoma by next generation DNA sequencing. J Cancer[J]. 2015, 6 :247–253 . |
| [7] | Guo X, Ba Y, Ma X, An J, Shang Y, Huang Q, et al. A meta-analysis of array-CGH studies implicates antiviral immunity pathways in the development of hepatocellular carcinoma. PLoS One[J]. 2011, 6 :e28404. |
| [8] | Wang K, Lim H Y, Shi S, Lee J, Deng S, Xie T, et al. Genomic landscape of copy number aberrations enables the identification of oncogenic drivers in hepatocellular carcinoma. Hepatology[J]. 2013, 58 :706–717 . |
| [9] | Yang F, Zhang L, Huo X S, Yuan J H, Xu D, Yuan S X, et al. Long noncoding RNA high expression in hepatocellular carcinoma facilitates tumor growth through enhancer of zeste homolog 2 in humans. Hepatology[J]. 2011, 54 :1679–1689 . |
| [10] | Guichard C, Amaddeo G, Imbeaud S, Ladeiro Y, Pelletier L, Maad I B, et al. Integrated analysis of somatic mutations and focal copy-number changes identifies key genes and pathways in hepatocellular carcinoma. Nat Genet[J]. 2012, 44 :694–698 . |
| [11] | Wurmbach E, Chen Y B, Khitrov G, Zhang W, Roayaie S, Schwartz M, et al. Genome-wide molecular profiles of HCV-induced dysplasia and hepatocellular carcinoma. Hepatology[J]. 2007, 45 :938–947 . |
| [12] | Yong J, Tan I, Lim L, Leung T. Phosphorylation of myosin phosphatase targeting subunit 3(MYPT3) and regulation of protein phosphatase 1 by protein kinase A. J Biol Chem[J]. 2006, 281 :31202–31211 . |
| [13] | Rebelo S, Santos M, Martins F, da Cruz E, Silva E F, da Cruz E, Silva O A. Protein phosphatase 1 is a key player in nuclear events. Cell Signal[J]. 2015, 27 :2589–2598 . |
| [14] | Figueiredo J, da Cruz E , Silva O A, Fardilha M. Protein phosphatase 1 and its complexes in carcinogenesis. Curr Cancer Drug Targets[J]. 2014, 14 :2–29 . |
| [15] | Winkler C, De Munter S, Van Dessel N, Lesage B, Heroes E, Boens S, et al. The selective inhibition of protein phosphatase-1 results in mitotic catastrophe and impaired tumor growth. J Cell Sci[J]. 2015, 128 :4526–4537 . |
| [16] | Zhao J, Wu G, Bu F, Lu B, Liang A, Cao L, et al. Epigenetic silence of ankyrin-repeat-containing, SH3-domain-containing, and proline-rich-region-containing protein 1(ASPP1) and ASPP2 genes promotes tumor growth in hepatitis B virus-positive hepatocellular carcinoma. Hepatology[J]. 2010, 51 :142–153 . |
| [17] | Vousden K H, Prives C. Blinded by the light:the growing complexity of p53. Cell[J]. 2009, 137 :413–431 . |
| [18] | Muller P A, Vousden K H. p53 mutations in cancer. Nat Cell Biol[J]. 2013, 15 :2–8 . |
| [19] | Jenkins L M, Durell S R, Mazur S J, Appella E. p53 N-terminal phosphorylation:a defining layer of complex regulation. Carcinogenesis[J]. 2012, 33 :1441–1449 . |
| [20] | Reed S M, Quelle D E. p53 acetylation:regulation and consequences. Cancers (Basel)[J]. 2014, 7 :30–69 . |
| [21] | Brooks C L, Gu W. p53 regulation by ubiquitin. FEBS Lett[J]. 2011, 585 :2803–2809 . |
| [22] | Gu B, Zhu W G. Surf the post-translational modification network of p53 regulation. Int J Biol Sci[J]. 2012, 8 :672–684 . |
| [23] | Meek D W. Regulation of the p53 response and its relationship to cancer. Biochem J[J]. 2015, 469 :325–346 . |
 2016, Vol. 37
2016, Vol. 37


