2. 中国科学院上海巴斯德研究所, 上海 200031
2. Institute Pasteur of Shanghai, Chinese Academy of Sciences, Shanghai 200031, China
丙型肝炎病毒(hepatitis C virus,HCV)感染是引发慢性肝炎、肝硬化和肝癌的原因之一[1]。目前,全世界有大约1.7亿人感染了HCV[1]。尽管现在已经研究出了针对HCV的有效的抗病毒药物,但是由于其昂贵的费用以及病毒的耐药性突变,其他新的HCV抗病毒药物及HCV疫苗的研发仍然受到关注[2]。HCV是正链的单链RNA病毒,属于黄病毒科。其基因组长度大约为9 600 nt,编码一条多肽链,经宿主本身蛋白酶和病毒编码的丝氨酸蛋白酶及信号肽酶裂解,产生结构蛋白core、包膜蛋白1(envelop protein 1,E1)、E2和离子通道蛋白p7,非结构蛋白NS2、NS3、NS4A/4B和NS5A/5B[3]。
感染性HCV颗粒的组装与宿主细胞因子紧密相关,主要是载脂蛋白A1(apolipoprotein A1,apoA1)、C1、B和E。尽管有报道称apoB是HCV颗粒的组分[4],但是更多的实验证据证实apoE和apoC1是感染性HCV颗粒的组分,尤其是apoE[2]。利用特异性小干扰RNA(small interfering RNA,siRNA)下调Huh7.5.1细胞中apoE的表达,可显著减少细胞内和分泌到细胞外的HCV水平[5~6],并且apoE和apoC1的抗体也可以中和HCV的感染性[6~7]。此外,通过电子显微镜也发现apoE为HCV颗粒的组分[8~9]。
增强型绿色荧光蛋白(enhanced green fluorescence protein,EGFP)是一种跟踪活体细胞中蛋白质行为的标记物[10]。由于apoE是感染性HCV的组分,因此构建apoE-EGFP HCV用以研究HCV的生活史具有一定的可能性。然而,目前仍不清楚apoE-EGFP细胞系是否支持感染性HCV的组装。本研究通过比较apoE-EGFP和Huh7.5.1细胞来源的HCV的感染性,证实了apoE-EGFP细胞系支持感染性HCV的组装,而且apoE-EGFP也能成功组装到感染性HCV颗粒中。
1 材料和方法 1.1 细胞培养和试剂Huh7.5.1 细胞由中国科学院上海巴斯德研究所钟劲研究员馈赠,HEK293T细胞由中国科学院上海巴斯德研究所钱志康研究员馈赠,Huh7.5.1细胞来源的对照细胞sh-NT细胞(转导非靶向野生型细胞基因的shRNA质粒的细胞)、apoE稳定敲低的Huh7.5.1细胞(sh-apoE细胞)、表达apoE-EGFP的sh-apoE细胞(apoE-EGFP细胞)均由本实验室构建。以上细胞均在含有10%胎牛血清(FBS,Gibco)、1%青霉素-链霉素(penicillin-streptomycin,Gibco)、2 mmol/L非必需氨基酸(non-essential amino acid,Gibco)的DMEM(Gibco)培养液中培养。pFK_Jc1_FlagE2、pAPM_sh-apoE_Puro、psPAX2和pMD2.G质粒为德国海德堡大学Ralf Bartenschlager教授馈赠。Goat anti-human apoE (ab947,Merck Millipore),rabbit anti-GFP (sc8334,Santa-cruz)和mouse anti-tubulin (66031,Proteintech),mouse anti-FLAG (M20008L,Abmart),mouse anti-NS5A(实验室自制);PCR引物由上海铂尚生物技术有限公司合成、纯化。T7 polymerase由本实验室纯化。
1.2 质粒及细胞系的构建pWPI_apoE-eGFP由本实验室构建;将人源性apoE与EGFP基因分别通过PCR扩增,使用TIANgel Midi Purification Kit[天根生化科技(北京)有限公司]纯化得到apoE和EGFP基因的PCR扩增产物。然后,将这两个基因片段通过重叠PCR连接,得到融合的apoE-EGFP基因。然后此基因通过BamH Ⅰ和SpeⅠ限制酶切位点插入pWPI_BLR载体中,获得pWPI_apoE_EGFP_BLR质粒。经上海铂尚生物技术有限公司测序鉴定,确认无误。扩增apoE-EGFP融合基因所用的引物:apoE上游引物为5′-AGA GGA TCC ATG AAG GTT CTG TGG-3′; apoE和EGFP共下游引物为5′-TCC TCC TCT TCC TCC TGA GTG ATT GTC GCT GGG-3′;apoE和EGFP共上游引物为5′-CAG GAG GAA GAG GAG GAG TGA GCA AGG GCG AG-3′;EGFP下游引物为5′-TCT ACT AGT TTA CTT GAC AGC TCG-3′。将psPAX2、pMD2.G、pWPI_apoE-EGFP_BLR、pAPM_sh-apoE_Puro和pAPM_sh-NT_Puro(对照组)质粒转染进入HEK293T细胞中,制备apoE-EGFP、sh-apoE和sh-NT慢病毒(sh-apoE RNA 靶向apoE mRNA的3′非编码区)。然后将sh-NT和sh-apoE慢病毒分别转导进入目的细胞Huh7.5.1 中,48 h后加入相应的抗生素进行筛选。获得sh-apoE细胞系后,再将apoE-EGFP慢病毒转导进入sh-apoE细胞系中,加抗生素筛选。最后,采用蛋白质印迹法鉴定apoE siRNA对靶细胞内apoE蛋白表达的下调作用,荧光显微镜观察apoE-EGFP的表达。
1.3 体外转录和HCV滴度的检测将pFK_HCV-Jc1_FLAGE2 DNA使用TIANquick Midi Purification Kit[天根生化科技(北京)有限公司]纯化后,按照Bartenschlager实验室的体外转录方法[3]将纯化后的HCV DNA转录、抽提RNA。通过电穿孔法将HCV Jc1-E2FLAG RNA分别转染进入Huh7.5.1、sh-NT、sh-apoE和apoE-EGFP细胞中,转染96 h后收取病毒液,使用半数组织培养感染剂量(TCID50)方法检测HCV的滴度[8]。
1.4 HCV的纯化研究所用的HCV RNA均为Jc1-E2FLAG RNA,即在HCV结构蛋白E2的N-末端加入了FLAG标签[6]。将HCV病毒液浓缩60倍后,按照FLAG-specific affinity gel(Sigma)操作指南纯化HCV颗粒,最后用100 μg/mL FLAG短肽溶液洗脱,得到纯化后的病毒液。
1.5 免疫荧光分析将靶细胞Huh7.5.1 铺入放有盖玻片的24孔板中,24 h后弃细胞培养液,加入HCV病毒液,37℃孵育4 h。弃病毒液,用磷酸盐缓冲液(PBS)洗3次,加入新鲜培养液,37℃孵育72 h。弃细胞培养液,用多聚甲醛固定20 min。移除多聚甲醛,PBS洗3次,用0.1% Triton X-100处理15 min;PBS洗3次,加入5%FCS封闭20 min。PBS洗3次,加入一抗,室温孵育1 h;PBS洗3次,加入二抗,室温孵育1 h;PBS洗3次,ddH2O洗1次。将长有细胞的盖玻片倒扣在载玻片上,用指甲油固定盖玻片,共聚焦荧光显微镜拍摄图片。
1.6 蛋白质印迹法分析收集的细胞或病毒用loading buffer处理,水浴30 min。SDS-PAGE分离蛋白后,将蛋白转移至PVDF膜上。用5%的脱脂牛奶溶液封闭1 h,加入一抗(5%脱脂牛奶溶液稀释),室温孵育1 h;PBST洗3次,加入二抗(1%脱脂牛奶溶液稀释),室温孵育1 h。PBST洗3次后用ECL发光液显影。
1.7 统计学处理采用GraghPad Prism 5.0进行数据处理与分析,组间比较采用Student’s t检验。检验水准(α)为0.05。
2 结 果 2.1 apoE-EGFP细胞系的构建利用shRNA基因沉默技术构建sh-apoE细胞系,并使外源基因apoE-EGFP在sh-apoE细胞系中稳定表达。蛋白质印迹法分析结果(图 1A)显示,sh-apoE细胞中apoE的蛋白表达水平下调,说明sh-apoE细胞系构建成功;此外,apoE-EGFP蛋白在apoE-EGFP细胞中大量表达。荧光倒置显微镜分析结果(图 1B)显示,在apoE-EGFP细胞的细胞质能观察到EGFP荧光,而在sh-NT细胞和sh-apoE细胞中观察不到绿色荧光,表明apoE-EGFP细胞系中apoE-EGFP可以高效表达。

|
图 1 表达apoE-EGFP的细胞系的构建 Fig 1 Establishment of apoE-EGFP-expressing cell line apoE-EGFP in apoE-EGFP cell lines was analyzed by Western blotting analysis (A) and fluorescence microscope (B). ▲: Non-specific band. apoE: Apolipoprotein E; EGFP: Enhanced green fluorescence protein. Original magnification: ×40 (B) |
2.2 apoE-EGFP融合蛋白表达对HCV感染性的影响
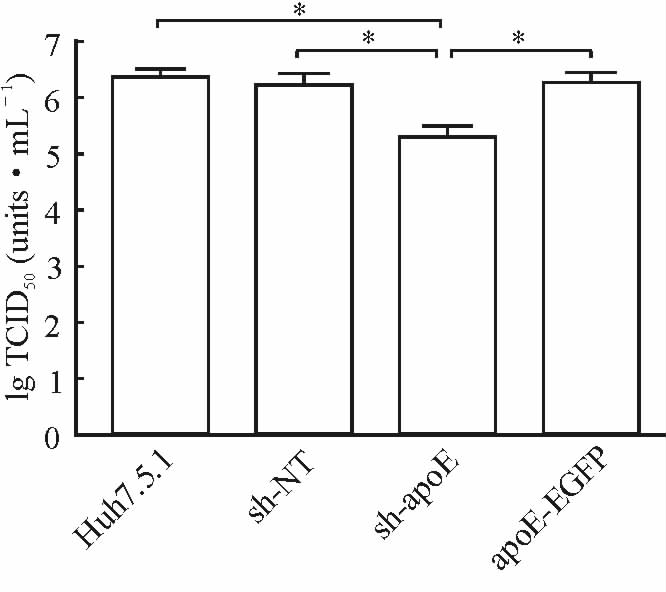
HCV的滴度检测结果(图 2)显示,与野生型Huh7.5.1 细胞相比,下调apoE的表达后,细胞产生的HCV的感染性下降约10倍;而在sh-apoE细胞中表达apoE-EGFP后,其产生的HCV的感染性达到了sh-NT细胞的水平。
|
图 2 ApoE-EGFP细胞系支持感染性HCV颗粒组装 Fig 2 ApoE-EGFP cell line supports assembly of infectious HCV particles HCV Jc1-E2FLAG RNA was electroporated into Huh7.5.1 ,sh-NT,sh-apoE and apoE-EGFP cell lines. The titers of these viruses were determined by TCID50 after culturing with free-serum media for 96 h. apoE: Apolipoprotein E; EGFP: Enhanced green fluorescence protein; HCV: Hepatitis C virus. *P<0.05. n=3,x±s |
2.3 apoE-EGFP融合蛋白与感染性HCV的关系
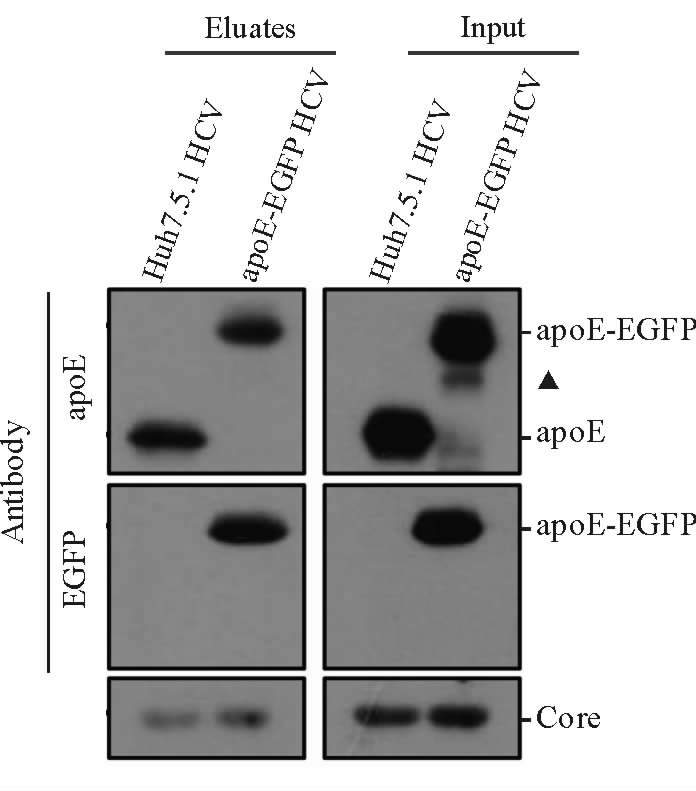
HCV分别感染Huh细胞和apoE-EGFP细胞后,采用蛋白质印迹法检测各病毒液,结果(图 3)显示,在所有细胞的病毒液中均检测到了病毒的结构蛋白core,并且在apoE-EGFP细胞来源的病毒液中纯化的HCV颗粒上,可检测到apoE-EGFP融合蛋白,而对照组中没有检测到,表明apoE-EGFP融合蛋白是感染性HCV的组分。
|
图 3 蛋白质印迹分析示apoE-EGFP是感染性HCV的组分 Fig 3 apoE-EGFP is a component of HCV particles by Western blotting analysis HCV Jc1-E2FLAG RNA was electroporated into Huh7.5.1and apoE-EGFP cell lines. apoE-EGFP and core proteins on the surface of HCV particles purified by FLAG-specific affinity gel were determined by Western blotting analysis. Eluates: Purified HCV; Input: Unpurified HCV. apoE: Apolipoprotein E; EGFP: Enhanced green fluorescence protein; HCV: Hepatitis C virus |
2.4 apoE-EGFP蛋白与HCV结构蛋白E2的相互作用
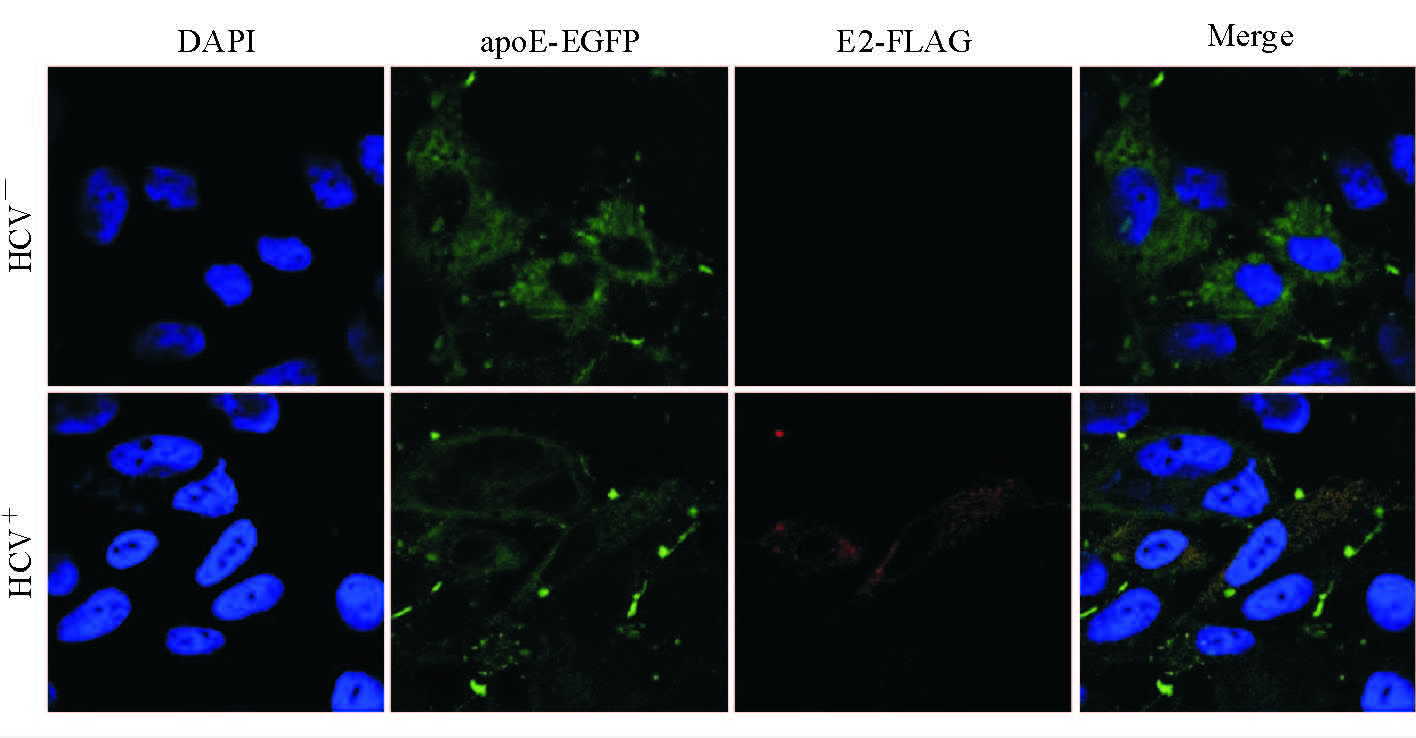
免疫荧光分析结果(图 4)显示,apoE-EGFP细胞内的apoE-EGFP与HCV结构蛋白E2共定位。
|
图 4 apoE-EGFP与HCV结构蛋白E2相互作用 Fig 4 apoE-EGFP interacts with HCV structural protein E2 apoE-EGFP cells were infected with HCV Jc1-E2FLAG viruses. Co-localization of apoE-EGFP and E2 was analyzed by immunofluorescence assay and confocal microscope. E2-FLAG was tested by anti-FLAG antibody. apoE: Apolipoprotein E; EGFP: Enhanced green fluorescence protein; HCV: Hepatitis C virus. Original magnification: ×200 |
3 讨 论
人源apoE作为一类重要的载脂蛋白,对脂类的运输具有重要作用。研究发现apoE与心血管疾病、阿尔茨海默病密切相关[11]。此外,科学家还发现它能影响HCV的感染性[5]。首先,HCV表面的apoE与宿主细胞表面的硫酸乙酰肝素的结合介导了HCV的吸附[12]。其次,细胞内的apoE与HCV的非结构蛋白NS5A相互作用,协助感染性HCV颗粒的组装[13]。最后,感染性HCV的释放依赖于细胞内apoE的分泌[14]。
本研究在shRNA介导apoE下调的Huh7.5.1细胞系中高效表达了apoE-EGFP融合蛋白,结果发现apoE-EGFP细胞系中产生的HCV颗粒的感染性与野生型细胞中产生的HCV颗粒的感染性相当。此外,将HCV RNA基因组转染进入apoE-EGFP细胞系后,在收集的病毒液中纯化出来的HCV颗粒上能检测到大量的apoE-EGFP融合蛋白。Lee等[15]研究证实apoE与包膜糖蛋白E2在细胞中相互作用,帮助HCV组装出成熟的病毒颗粒。本研究用HCV感染apoE-EGFP细胞系,通过荧光共聚焦实验也观测到了apoE-EGFP和E2的相互作用。这些结果表明,apoE-EGFP细胞系支持感染性HCV颗粒的组装。此外,基于EGFP具有跟踪活细胞中蛋白质行为的特性,apoE-EGFP细胞系也可以用于示踪HCV颗粒组装的动态过程。
利用外源的荧光蛋白及标签标记活体细胞内的蛋白质是一种研究蛋白质功能的有效手段。Moradpour等[16]将GFP插入HCV非结构蛋白NS5A中,实现了对HCV RNA复制复合体的可视化。有研究报道,用四半胱氨酸标签标记HCV结构蛋白core,可展示core在细胞中的转运及其与宿主细胞因子之间的相互关系[17]。然而,迄今为止还没有对感染性HCV颗粒进行标记的研究报道。本研究构建的apoE-EGFP HCV颗粒的感染性与野生型HCV相当,有望用于更加深入地研究HCV的生活史,为HCV患者找到新的治疗策略。
| [1] | GOOSSENS N, HOSHIDA Y. Hepatitis C virus-induced hepatocellular carcinoma[J]. Clin Mol Hepatol, 2015, 21: 105–114. DOI: 10.3350/cmh.2015.21.2.105 |
| [2] | PERALES C, QUER J, GREGORI J, ESTEBAN J I, DOMINGO E. Resistance of hepatitis C virus to inhibitors: complexity and clinical implications[J]. Viruses, 2015, 7: 5746–5766. DOI: 10.3390/v7112902 |
| [3] | BARTENSCHLAGER R, PENIN F, LOHMANN V, ANDRÉ P. Assembly of infectious hepatitis C virus particles[J]. Trends Microbiol, 2011, 9: 95–103. |
| [4] | THOMSSEN R, BONK S, PROPFE C, HEERMANN K H, KÖCHEL H G, UY A. Association of hepatitis C virus in human sera with beta-lipoprotein[J]. Med Microbiol Immunol, 1992, 181: 293–300. DOI: 10.1007/BF00198849 |
| [5] | CHANG K S, JIANG J, CAI Z, LUO G. Human apolipoprotein E is required for infectivity and production of hepatitis C virus in cell culture[J]. J Virol, 2007, 81: 13783–13793. DOI: 10.1128/JVI.01091-07 |
| [6] | JIANG J, LUO G. Apolipoprotein E but not B is required for the formation of infectious hepatitis C virus particles[J]. J Virol, 2009, 83: 12680–12691. DOI: 10.1128/JVI.01476-09 |
| [7] | MEUNIER J C, RUSSELL R S, ENGLE R E, FAULK K N, PURCELL R H, EMERSON S U. Apolipoprotein c1 association with hepatitis C virus[J]. J Virol, 2008, 82: 9647–9656. DOI: 10.1128/JVI.00914-08 |
| [8] | MERZ A, LONG G, HIET M S, BRÜGGER B, CHLANDA P, ANDRE P, et al. Biochemical and morphological properties of hepatitis C virus particles and determination of their lipidome[J]. J Biol Chem, 2011, 286: 3018–3032. DOI: 10.1074/jbc.M110.175018 |
| [9] | CATANESE M T, URYU K, KOPP M, EDWARDS T J, ANDRUS L, RICE W J, et al. Ultrastructural analysis of hepatitis C virus particles[J]. Proc Natl Acad Sci USA, 2013, 110: 9505–9510. DOI: 10.1073/pnas.1307527110 |
| [10] | 艾伯茨. 细胞生物学精要[M]. 北京: 科学出版社, 2012. |
| [11] | MAHLEY R W, RALL S C Jr. Apolipoprotein E: far more than a lipid transport protein[J]. Annu Rev Genomics Hum Genet, 2000, 1: 507–537. DOI: 10.1146/annurev.genom.1.1.507 |
| [12] | JIANG J, CUN W, WU X, SHI Q, TANG H, LUO G. Hepatitis C virus attachment mediated by apolipoprotein E binding to cell surface heparan sulfate[J]. J Virol, 2012, 86: 7256–7267. DOI: 10.1128/JVI.07222-11 |
| [13] | BENGA W J, KRIEGER S E, DIMITROVA M, ZEISEL M B, PARNOT M, LUPBERGER J, et al. Apolipoprotein E interacts with hepatitis C virus nonstructural protein 5A and determines assembly of infectious particles[J]. Hepatology, 2010, 51: 43–53. DOI: 10.1002/hep.23278 |
| [14] | HISHIKI T, SHIMIZU Y, TOBITA R, SUGIYAMA K, OGAWA K, FUNAMI K, et al. Infectivity of hepatitis C virus is influenced by association with apolipoprotein E isoforms[J]. J Virol, 2010, 84: 12048–12057. DOI: 10.1128/JVI.01063-10 |
| [15] | LEE J Y, ACOSTA E G, STOECK I K, LONG G, HIET M S, MUELLER B, et al. Apolipoprotein E likely contributes to a maturation step of infectious hepatitis C virus particles and interacts with viral envelope glycoproteins[J]. J Virol, 2014, 88: 12422–12437. DOI: 10.1128/JVI.01660-14 |
| [16] | MORADPOUR D, EVANS M J, GOSERT R, YUAN Z, BLUM H E, GOFF S P, et al. Insertion of green fluorescent protein into nonstructural protein 5A allows direct visualization of functional hepatitis C virus replication complexes[J]. J Virol, 2004, 78: 7400–7409. DOI: 10.1128/JVI.78.14.7400-7409.2004 |
| [17] | COLLER K E, HEATON N S, BERGER K L, COOPER J D, SAUNDERS J L, RANDALL G. Molecular determinants and dynamics of hepatitis C virus secretion[J/OL]. PLoS Pathog, 2012, 8: e1002466. doi: 10.1371/journal.ppat.1002466. |
 2016, Vol. 37
2016, Vol. 37


