2. 第二军医大学长海医院肛肠外科, 上海 200433
△共同第一作者
2. Department of Colorectal Surgery, Changhai Hospital, Second Military Medical University, Shanghai 200433, China
△Co-first authors.
结直肠癌(colorectal carcinoma,CRC)是全球最常见的恶性肿瘤之一,全球每年新发病例100多万人,且有上升趋势[1]。CRC的发病与饮食、环境、遗传等多种因素相关,其分子机制复杂,目前尚不明确。近年来,随着微阵列和高通量测序等组学技术的发展,组学检测逐渐进入广大生物医学研究者的工作平台。但是,一次组学检测常发现数百甚至上千CRC相关基因[2, 3, 4],使研究者难以确定哪些是重要的,哪些是次要的。同时,组学检测费用昂贵,致使许多CRC研究的样本量较小,再加上CRC个体之间存在较大异质性[5, 6, 7],使得不同组学研究所得到的差异基因表达谱有很大不同[8]。因此,开发新的数据分析和整合策略对组学数据挖掘、发现重要的甚至新的CRC发生相关关键分子十分重要。
系统生物学观点认为: 在大批疾病相关基因和通路中,并非所有都有重要意义,其中许多都是被动发生变化(“乘客”基因),只有部分基因的变化是驱动性的(“司机”基因),研究者应该发现那些发挥“司机”作用的基因和通路[9, 10]。在本研究中,我们首先利用meta分析技术从既往5项研究中寻找CRC发生相关差异表达基因,然后构建差异表达基因在癌组织中的共表达网络,进而发现共表达网络中的核心亚网络并分析其生物功能和基因节点的剂量-效应关系[10],最后确定CRC发生中的核心通路和驱动分子,从而为今后CRC发生机制的研究提供候选分子。
1 资料和方法 1.1 基因表达谱芯片数据下载及预处理从GEO数据库中搜索并下载5项与CRC发生相关的表达谱芯片研究(GSE8671[11]、GSE22598[12]、GSE23878[13]、GSE9348[14]和GSE37364[15])的原始数据。所有研究的检测平台均为Affymetrix Plus 2.0,研究所包含的癌旁和癌组织样本均超过10例。具体情况为:GSE8671包含配对的癌和癌旁样本各32例;GSE22598包含配对的癌和癌旁样本各17例;GSE23878包含非配对的癌旁样本24例和癌样本35例;GSE9348包含癌旁样本12例和癌样本70例;GSE37364包含癌旁样本38例和癌样本27例。在R.3.0.1环境下,原始数据(.cel格式)首先用fRMA[16]提取表达谱数据,基于barcode文件[16]剔除那些在5项研究中均不表达的探针,再进一步基于Jetset文件[17]选择那些标记为“Perfect”的探针,最终留存10 717个探针进行深入分析。
1.2 差异表达基因分析5项CRC相关研究的表达谱数据在格式调整后,用MetaDE软件[18]继续在R统计平台下进行单独及合并的差异表达基因分析。具体过程为:首先根据MetaDE软件建议过滤掉30%低表达的基因和30%样本间变异小的基因,然后计算单独研究中癌和癌旁组织的差异表达基因(其中GSE8671和GSE22598为配对设计),最后用maxP法[18]合并各单独研究的结果并选择假阳性率(false discovery rate,FDR)≤0.01的差异表达基因。本研究剔除5项研究中差异表达方向不一致的差异表达基因进行后续分析。
1.3 共表达网络的构建及可视化为了构建差异表达基因在CRC中的共表达网络,首先利用sva软件包[19]中的“combat”命令将5项CRC相关研究的表达谱数据合并,手工提取差异表达基因所对应的表达谱数据,之后用PCIT软件[20]对癌组织样本进行共表达网络构建(PCIT可利用偏回归系数法去掉那些间接相关的共表达基因对)。为了获得稳定的共表达网络,我们将181个癌组织样本随机分成A组(n=90)和B组(n=91),计算A、B组以及(A+B)组的共表达网络,选择各组共表达分析有统计学意义且相关系数绝对值>0.6(且P<0.05)的基因对,并在Cytoscape 3.1.0软件[21]平台下获得3组研究一致存在的共表达网络。
1.4 核心网络的搜索及功能分析对于形成的共表达网络,我们利用CFinder软件[22]进行核心亚网络的搜索,其中参数K为4。 对于CFinder所确定的核心亚网络,进一步用Gather[23] (http://gather.genome.duke.edu/)对其所富集的GO(Gene Ontology)和KEGG相关生物学功能进行分析,并选择Bayes因子≥6的功能为有统计学意义。
1.5 核心亚网络相关驱动基因的分析那些mRNA水平受到DNA拷贝数影响的基因在癌症形成中更可能发挥“驱动”作用[9, 10, 24]。为了确定核心亚网络中的驱动基因,我们从美国明尼苏达癌症中心cBioPortal网站(http://www.cbioportal.org/)下载所有核心亚网络基因在CRC样本[25]中的基因拷贝数变异信息(即下载GISTIC文件);其中,“-2和-1”代表基因拷贝数缺失,“2和1”代表基因拷贝数扩增,“0”代表基因拷贝数正常。接下来,首先计算核心亚网络基因在CRC样本中的扩增频率和缺失频率,然后联合这些基因在CRC中的表达变化方向,确定那些在癌组织中扩增频率高且表达升高和那些在癌组织中缺失频率高且表达降低的基因为亚网络功能形成中的候选驱动基因。
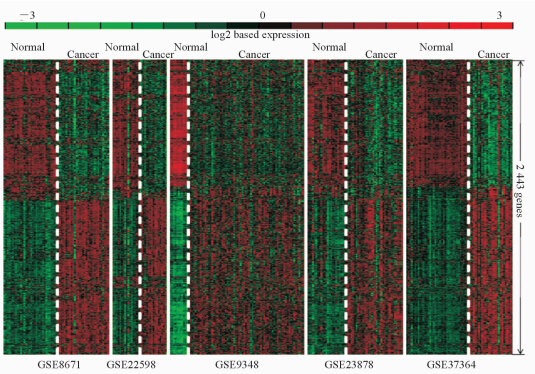
2 结 果 2.1 CRC及其癌旁组织之间的差异表达基因当FDR≤0.01时,MetaDE分析发现GSE8671、GSE22598、GSE23878、GSE9348和GSE37364等5项研究分别获得3 173、2 130、2 726、1 770和2 961个差异表达基因。利用maxP法合并5项研究的P值后共得到2 443个差异表达基因,其中在4项及以上研究的癌组织中一致上调的有1 304个基因,一致下调的有1 018个基因;在5项研究的癌组织中一致上调的有1 174个基因,一致下调的有899个基因。图 1为2 443个基因在5项研究中表达的整体情况。
|
图 1 CRC及其癌旁组织差异表达基因在5项研究中的表达模式 Fig 1 Expression pattern of differentially expressed genes identified from 5 studies between CRC and adjacent normal mucosa CRC:Colorectal carcinoma |
在5项研究中一致上调或一致下调的2 073个基因被用于共表达网络构建。2 073个差异基因在A组、B组和(A+B)组中的两两相关系数的频数分布如图 2,发现有统计学意义的相关系数依次为272 986、232 649和253 506个(其中一个系数对应一个共表达基因对);进一步将3组中相关系数绝对值>0.6的挑出,依次获得3 897、3 721和3 926个基因对,分别对应996、987和1 021个差异表达基因。利用Cytoscape 3.1.0对3个共表达网络进行合并,发现在3者中一致存在的网络是由798个基因和1 462条边组成。

|
图 2 A组、B组和(A+B)组样本中有意义共表达相关系数的分布 Fig 2 Distribution of meaningful correlation coefficients from samples of A,B,and (A+B) groups Meaningful coefficients marked red. n=90 for A group,n=91 for B group,and n=181 for all |
CFinder从CRC共表达网络中发现22个核心亚网络,其中最大的核心亚网络有77个节点基因,其余核心亚网络从大到小依次为29个节点基因(1个)、10个节点基因(1个)、9个节点基因(1个)、8个节点基因(1个)、7个节点基因(3个)、6个节点基因(4个)、5个节点基因(2个)和4个节点基因(8个)。其中,最大的5个核心亚网络(SubNet 1~5)如图 3所示。

|
图 3 CRC发生相关差异基因所形成的5个核心亚网络(SubNet 1~5) Fig 3 Five core sub-networks (SubNet 1-5) constructed based on differently expressed genes in CRC |
亚网络功能富集度分析发现(表 1):SubNet 1的功能主要涉及蛋白质等大分子的生物合成;SubNet 3的功能主要涉及细胞周期调控和增殖,该通路已被广泛报道与多种癌症发生密切相关[9, 26];SubNet 2、SubNet 4和SubNet 5未发现富集某种生物学功能,但其亚网络节点基因分别富集于不同的染色体(表 1);尤其SubNet 2,其29个节点基因中分别有16个和11个位于染色体20q13和20q11,提示CRC组织DNA拷贝数变异影响SubNet 2中基因的表达。20q13和20q11染色体区域拷贝数增加,以及20q13染色体区域多态性位点已被报道和CRC的发生显著相关[24, 27]。
|
|
表 1 SubNet 1~5的功能富集度分析情况 Tab 1 Functional enrichment analysis of gene nodes in SubNet 1-5 |
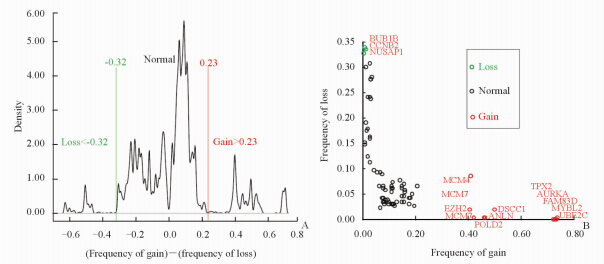
对于功能富集明确的SubNet 3,进一步考察其所包含基因在CRC中上调、下调方向和对应染色体区扩增或缺失之间的对应关系。为此,我们首先计算cBioPortal网站中下载的257例CRC样本中20 876个基因的扩增频率、缺失频率及扩增频率减去缺失频率的差值,并对所有的差值绘制概率密度图(图 4A)。根据概率密度图界定:(扩增频率-缺失频率)> 0.23为扩增,(扩增频率-缺失频率)<-0.32为缺失,其余为正常。随后SubNet 3中77个基因在CRC中的拷贝数变异相应分为扩增、缺失和正常,由于这些基因在CRC中均呈高表达(图 1),故UBE2C、MYBL2、FAM83D、AURKA和TPX2等11个基因的高表达与染色体扩增正相关(图 4B),可能为细胞周期和增殖调控的驱动基因;虽然BUB1B、CCNB2和NUSAP1等基因在CRC中高表达,但其染色体区域却高频率缺失,提示这3个基因的表达变化可能由其他机制(如miRNA等)调控。
|
图 4 核心亚网络SubNet 3中关键驱动基因的确定 Fig 4 Identification of the driver genes of SubNet 3 in CRC A:Development of cut-off values for chromosomal aberrations in CRC;B:The relationship of mRNA expression and corresponding copy number variation of genes in SubNet 3. CRC:Colorectal carcinoma |
CRC的发生具有很大异质性[5, 6, 7],导致患者的临床表现和预后具有很大不同,要研究CRC发生相关的基因表达谱必须具有较大的样本量。然而,由于基因表达谱芯片价格昂贵,很少有单个实验室能够使用大量的基因芯片进行CRC及其癌旁差异表达基因的筛选。在本研究中,我们基于既往5项有关CRC发生的转录组学研究,用MetaDE软件包中的maxP方法[18]整合发现CRC及其癌旁组织中存在2 443个差异表达基因,其中一致性上调的有1 174个基因,一致性下调的有899个基因,5项研究不一致的有370个基因(图 1)。这样的分析不仅扩增了研究的样本量,也找到了不同实验室之间可以重复的研究结果,显著提高了CRC发生相关基因的筛选效率。
表达谱芯片技术发现的数百甚至数千疾病相关基因往往组成生物学通路信号执行相关功能[28],故对于CRC差异表达基因的理解不能仅停留在基因倍数的差异,而应该对其形成的信号网络进行探索。共表达网络是一种探索疾病相关基因间正向或负向相关变化的方法,许多有共表达关系的基因更倾向于形成生物学通路信号[20]。为了得到稳定的共表达网络,我们首先将5项CRC研究中的癌组织表达谱数据合并,随机分为2组,并与所有样本组进行3次共表达网络构建,取其共有且相关系数绝对值>0.6的共表达基因对构成1个由798个基因和1 462条共表达关系组成的稳健的网络,该网络清楚地展示了CRC相关差异表达基因在癌症表型维持中的组织模式。根据共表达网络的拓扑结构,CFinder发现了5个较大的核心亚网络SubNet 1~5(图 3),提示这些亚网络有可能通过不同的功能促进CRC的发生和发展。尽管SubNet 2、4、5所富集的功能并不能用目前KEGG和Gene Ontology数据库所搜集的通路信号功能解释,但是,我们发现SubNet 3的功能主要涉及细胞周期和增殖调控,SubNet 1的功能主要涉及蛋白质等大分子合成(表 1)。事实上,细胞周期和增殖调控等生物学功能均报道与癌症的发展密切相关[9, 26],提示我们的共表达网络分析在揭示CRC发生机制上是有效的。
在癌症相关亚网络中的众多基因中,许多基因的表达变化是被动的或伴随的,只有少数基因才发挥“始动”作用(亦即“司机”基因),发现这些具有始动作用的基因才是研究的关键[9, 10, 24]。许多致癌因素首先通过破坏基因组DNA稳定性,然后导致DNA拷贝数变异和基因组突变,并通过这些异常的结构变化来促进癌相关基因在mRNA和(或)蛋白层面量和质的变化[28, 29]。一些学者认为:癌症相关差异表达基因中,同时具有相应基因组结构变化(突变、扩增、缺失等)者可能是癌症形成的“司机”基因[9, 10, 24]。围绕该假设,我们进一步获得SubNet 3中所有基因在CRC组织中对应染色体区域扩增和缺失的频率,并将其与这些基因在CRC中的表达变化方向相结合,以发现SubNet 3中的“司机”基因。我们发现UBE2C、MYBL2、FAM83D、AURKA和TPX2等11个基因的高表达与染色体扩增正相关,提示这些基因是SubNet 3能形成的始动因素,是本研究中的关键基因(图 4)。事实上,UBE2C和AURKA已被报道在细胞周期调控中发挥关键作用[30, 31]。
归纳而言,本研究利用meta分析的方法从既往5项CRC发生相关基因组研究中获得了2 443个癌和癌旁组织差异表达基因;并进一步经共表达网络分析发现细胞周期和增殖调控等生物学功能是CRC发生中的核心通路;最后,联合基因拷贝数变异数据发现UBE2C、MYBL2、FAM83D、AURKA和TPX2等11个基因的高表达与染色体扩增正相关,是CRC细胞周期和增殖调控通路的关键驱动基因,为今后课题组深入研究CRC发生的分子机制提供大量研究基础。当然,本研究也存在不足,例如在探讨基因表达变化和基因组拷贝数变异关系时,所用的信息来自不同的样本群,当两个样本群存在较大变异时,研究推论将会受到影响。
| [1] | Siegel R, Naishadham D, Jemal A. Cancer statistics, 2013[J]. CA Cancer J Clin,2013, 63: 11-30. |
| [2] | Xu Y, Xu Q, Yang L, Liu F, Ye X, Wu F, et al. The effect of colonoscopy on whole blood gene expression profile: an experimental investigation for colorectal cancer biomarker discovery[J]. J Cancer Res Clin Oncol, 2015,14: 591-599. |
| [3] | Solé X, Crous-Bou M, Cordero D, Olivares D, Guinó E, Sanz-Pamplona R, et al. Discovery and validation of new potential biomarkers for early detection of colon cancer[J]. PLoS One, 2014, 9: e106748. |
| [4] | Wang Y, Zheng T. Screening of hub genes and pathways in colorectal cancer with microarray technology[J]. Pathol Oncol Res, 2014,20: 611-618. |
| [5] | Marisa L, de Reyniès A, Duval A, Selves J, Gaub M P, Vescovo L, et al. Gene expression classification of colon cancer into molecular subtypes: characterization, validation, and prognostic value[J]. PLoS Med, 2013, 10: e1001453. |
| [6] | De Sousa E Melo F, Wang X, Jansen M, Fessler E, Trinh A, de Rooij L P, et al. Poor-prognosis colon cancer is defined by a molecularly distinct subtype and develops from serrated precursor lesions[J]. Nat Med, 2013, 19: 614-618. |
| [7] | Hinoue T, Weisenberger D J, Lange C P, Shen H, Byun H M, Van Den Berg D, et al. Genome-scale analysis of aberrant DNA methylation in colorectal cancer[J]. Genome Res, 2012, 22: 271-282. |
| [8] | Chang W, Gao X, Han Y, Du Y, Liu Q, Wang L, et al. Gene expression profiling-derived immunohistochemistry signature with high prognostic value in colorectal carcinoma[J]. Gut, 2014, 63: 1457-1467. |
| [9] | Mine K L, Shulzhenko N, Yambartsev A, Rochman M, Sanson G F, Lando M, et al. Gene network reconstruction reveals cell cycle and antiviral genes as major drivers of cervical cancer[J]. Nat Commun, 2013, 4: 1806. |
| [10] | Akavia U D, Litvin O, Kim J, Sanchez-Garcia F, Kotliar D, Causton H C, et al. An integrated approach to uncover drivers of cancer[J]. Cell, 2010, 143: 1005-1017. |
| [11] | Sabates-Bellver J, van der Flier L G, de Palo M, Cattaneo E, Maake C, Rehrauer H, et al. Transcriptome profile of human colorectal adenomas[J]. Mol Cancer Res, 2007, 5: 1263-1275. |
| [12] | Okazaki S, Ishikawa T, Iida S, Ishiguro M, Kobayashi H, Higuchi T, et al. Clinical significance of UNC5B expression in colorectal cancer[J]. Int J Oncol, 2012, 40: 209-216. |
| [13] | Uddin S, Ahmed M, Hussain A, Abubaker J, Al-Sanea N, AbdulJabbar A, et al. Genome-wide expression analysis of Middle Eastern colorectal cancer reveals FOXM1 as a novel target for cancer therapy[J]. Am J Pathol, 2011, 178: 537-547. |
| [14] | Hong Y, Downey T, Eu K W, Koh P K, Cheah P Y. A‘metastasis-prone’signature for early-stage mismatch-repair proficient sporadic colorectal cancer patients and its implications for possible therapeutics[J]. Clin Exp Metastasis, 2010, 27: 83-90. |
| [15] | Galamb O, Sipos F, Solymosi N, Spisák, S, Krenács T, Tóth K, et al. Diagnostic mRNA expression patterns of inflamed, benign, and malignant colorectal biopsy specimen and their correlation with peripheral blood results[J]. Cancer Epidemiol Biomarkers Prev, 2008, 17: 2835-2845. |
| [16] | McCall M N, Bolstad B M, Irizarry R A. Frozen robust multiarray analysis (fRMA)[J]. Biostatistics, 2010, 11: 242-253. |
| [17] | Li Q, Birkbak N J, Gyorffy B, Szallasi Z, Eklund A C. Jetset: selecting the optimal microarray probe set to represent a gene[J]. BMC Bioinformatics, 2011, 12: 474. |
| [18] | Wang X, Kang D D, Shen K, Song C, Lu S, Chang L C, et al. An R package suite for microarray meta-analysis in quality control, differentially expressed gene analysis and pathway enrichment detection[J]. Bioinformatics, 2012, 28: 2534-2536. |
| [19] | Leek J T, Johnson W E, Parker H S, Jaffe A E, Storey J D. The sva package for removing batch effects and other unwanted variation in high-throughput experiments[J]. Bioinformatics, 2012, 28: 882-883. |
| [20] | Watson-Haigh N S, Kadarmideen H N, Reverter A. PCIT: an R package for weighted gene co-expression networks based on partial correlation and information theory approaches[J]. Bioinformatics, 2010, 26: 411-413. |
| [21] | Shannon P, Markiel A, Ozier O, Baliga N S, Wang J T, Ramage D, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13: 2498-2504. |
| [22] | Adamcsek B, Palla G, Farkas I J, Derényi I, Vicsek T. CFinder: locating cliques and overlapping modules in biological networks[J]. Bioinformatics, 2006, 22: 1021-1023. |
| [23] | Chang J T, Nevins J R. GATHER: a systems approach to interpreting genomic signatures[J]. Bioinformatics, 2006, 22: 2926-2933. |
| [24] | Mekenkamp L J, Haan J C, Koopman M, Vink-Börger M E, Israeli D, Teerenstra S, et al. Chromosome 20p11 gains are associated with liver-specific metastasis in patients with colorectal cancer[J]. Gut, 2013, 62: 94-101. |
| [25] | Cancer Genome Atlas Network. Comprehensive molecular characterization of human colon and rectal cancer[J]. Nature, 2012, 487: 330-337. |
| [26] | Cuzick J, Swanson G P, Fisher G, Brothman A R, Berney D M,Reid J E,et al. Prognostic value of an RNA expression signature derived from cell cycle proliferation genes in patients with prostate cancer: a retrospective study[J]. Lancet Oncol, 2011, 12: 245-255. |
| [27] | Sillars-Hardebol A H, Carvalho B, Tijssen M, Belin J A, de Wit M, Delis-van Diemen P M, et al. TPX2 and AURKA promote 20q amplicon-driven colorectal adenoma to carcinoma progression[J]. Gut, 2012, 61: 1568-1575. |
| [28] | Campbell P J,Yachida S,Mudie L J,Stephens P J,Pleasance E D,Stebbings L A,et al. The patterns and dynamics of genomic instability in metastatic pancreatic cancer[J]. Nature, 2010, 467: 1109-1113. |
| [29] | Alhopuro P,Sammalkorpi H,Niittymäki I,Biström M,Raitila A,Saharinen J, et al. Candidate driver genes in microsatellite-unstable colorectal cancer[J]. Int J Cancer, 2012, 130: 1558-1566. |
| [30] | Xie C,Powell C,Yao M,Wu J,Dong Q. Ubiquitin-conjugating enzyme E2C: A potential cancer biomarker[J]. Int J Biochem Cell Biol, 2014, 47: 113-117. |
| [31] | Mehra R, Serebriiskii I G, Burtness B, Astsaturov I, Golemis E A. Aurora kinases in head and neck cancer[J]. Lancet Oncol, 2013, 14: e425-e435. |
 2015, Vol. 36
2015, Vol. 36


