2. 复旦大学生命科学学院, 上海 200433;
3. 第二军医大学热带医学和公共卫生学系生物防御(微生物)教研室, 上海 200433
2. School of Life Sciences, Fudan University, Shanghai 200433, China;
3. Department of Biological Prevention (Microbiology), Faculty of Tropical Medicine and Public Health, Second Military Medical University, Shanghai 200433, China
埃博拉病毒(Ebola virus,EBOV)是能够在人类和灵长类动物中引起高死亡率的病毒,被WHO列为对人类危害最严重的病毒之一,位居第四级致病生物[1]。EBOV属于丝状病毒科(Filoviridae)、丝状病毒属(Filovirus),其基因组为不分节段的单股负链RNA,全长约19 kb,共有7个基因、9个开放阅读框,分别编码NP (nucleoprotein)、VP35(virion protein 35)、VP40(virion protein 40)、GP(glycoprotein)、sGP(secreted glycoprotein)、ssGP(small secreted glycoprotein)、VP30(virion protein 30)、VP24(virion protein 24)和L(RNA-dependent RNA polymerase)蛋白。
EBOV共分为5个亚型,分别是扎伊尔型、苏丹型、科特迪瓦型(塔伊森林型)、莱斯顿型及本迪布焦型。自1976年首次暴发以来,乌干达、苏丹和刚果民主共和国等中非国家先后发生过20多起EBOV疫情,绝大数的疫情都是由扎伊尔型或苏丹型EBOV引发的。在既往发生的历次EBOV疫情中,一般仅有几十或上百例感染者,最严重的疫情也只有300多例感染者(1976年和1995年刚果民主共和国暴发的两次EBOV疫情中,分别有318例和315例感染者)。然而,2014年在西非国家暴发的EBOV疫情,自2月以来在西非的几内亚、塞拉利昂和利比里亚等国家不断蔓延,根据世界卫生组织(WHO)的统计,截至2014年10月7日,上述3个国家的EBOV感染病例共计8 376人,其中4 024人死亡,病死率为48%(http://www.who.int/csr/disease/ebola/situation-reports/en/)。此次疫情中流行的毒株是扎伊尔型EBOV,与既往该型病毒引发的绝大多数疫情相比,此次疫情感染者的病死率显著降低(http://www.who.int/)。这提示此次疫情中的EBOV毒株基因组序列很可能发生了变异,从而导致流行病学特征的变化,即病毒的致死能力减弱(表现为病死率显著低于既往疫情),人际传播能力增强(表现为感染病例远远多于历次疫情感染病例的总和)。本研究对NCBI公共数据库中所有的EBOV 2014扎伊尔型毒株基因组序列(102株)进行了分析,通过研究其变异特征,探讨EBOV基因组变异与其流行病学特征改变之间的关系。
1 材料和方法 1.1 EBOV基因组序列来源选取NCBI公共数据库中现有的扎伊尔型EBOV全基因组序列共计130条,其中102条序列来自2014年毒株,序列编号为AF272001.1,AF499101.1,AY142960.1,AY354458.1,EU224440.2,HQ613402.1,HQ613403.1,JQ352763.1,KC242784.1-KC242801.1,KF827427.1,NC_002549.1,KJ660346.1-KJ660348.1,KM034549.1-KM034563.1,KM233035.1-KM233118.1。
1.2 分析方法以1976年EBOV扎伊尔型毒株的全基因组序列(NCBI序列号:NC_002549.1)作为参考序列,通过Mummer3.0软件[2](http://mummer.sourceforge.net/)分析其余129株EBOV的基因组变异特征。使用MEGA5软件[3](http://www.megasoftware.net/),利用近邻结合法(neighbor-joining)进行蛋白进化分析。应用CPHmodels(http://www.cbs.dtu.dk/services/CPHmodels/)软件,根据蛋白的同源性获得GP蛋白的pdb文件,利用PyMOL软件(http://www.pymol.org/)模拟GP蛋白的三维构型。
2 结 果 2.1 EBOV 2014扎伊尔型毒株基因组变异特征选取EBOV 1976毒株的基因组序列作为参考序列,我们发现在102条EBOV 2014扎伊尔型毒株基因组序列中,共存在606个变异位点(single nucleotide variants,SNVs),其中228个SNVs分布在基因组的非编码区,378个SNVs分布在NP、VP35、VP40、GP/ sGP/ ssGP、VP30、VP24和L基因的编码区。位于基因编码区的378个SNVs中,共有84个位点的变异属于非同义突变,其中35个SNVs均曾经一次或多次出现于以往的EBOV毒株序列中,而其余49个SNVs是EBOV 2014扎伊尔型毒株所特有的(表 1)。从表 1中可以看出,有37个EBOV 2014毒株特有的SNVs(表 1中以粗体标示)在102条EBOV 2014扎伊尔型毒株的基因组序列中是多次重现的(recurrent)。而另外12个SNVs位点,即第572、2 124、2 364、3 388、4 759、6 175、6 909、10 801、12 878、15 599、16 054和16 750位碱基的变异,仅在个别(≤3个)EBOV 2014毒株中重现。这些位点不能够很好地代表EBOV 2014扎伊尔型毒株的基因组变异特征,因此在本文中不予探讨。
|
|
表 1 EBOV 2014扎伊尔型毒株特有的49个非同义SNV位点 Tab 1 The 49 unique nonsynonymous SNVs in Zaire EBOV 2014 |
在上述的37个EBOV 2014扎伊尔型毒株特有的SNVs中,第852、6 283和17 431位碱基的突变,将分别导致NP蛋白第128位氨基酸由赖氨酸突变为精氨酸、GP蛋白第82位氨基酸由丙氨酸突变为缬氨酸、L蛋白第1 951位氨基酸由异亮氨酸突变为缬氨酸。通过序列保守性分析,我们发现在2014年之前的所有EBOV亚型中(包括扎伊尔型、苏丹型、塔伊森林型、莱斯顿型及本迪布焦型),NP蛋白第128位氨基酸、GP蛋白第82位氨基酸和L蛋白第1 951位氨基酸是高度保守的,仅在EBOV 2014扎伊尔型毒株发生了突变(图 1)。其余34个位点所编码的氨基酸,在埃博拉病毒各亚型之间是可变的、非保守的。

|
图 1 埃博拉病毒NP(A)、GP(B)和L蛋白(C)保守性分析 Fig 1 Multiple alignments of NP (A),GP (B) and L(C) protein sequence The amino acids which were highly conserved among all EBOV species except for 2014 strains are indicated by red color,and amino acids which are not highly conserved among all EBOV species are blue. The numbers at the top of sequences refer to the position of amino-acid residues. The GenBank accession numbers of the sequences are as follows: Zaire-2014(KM233117.1),Zaire-1976(NC_002549.1),Zaire-1977(KC242791.1),Zaire-1994(KC242792.1),Zaire-1995(JQ352763.1),Zaire-1996(KC242793.1),Zaire-2002(KC242800.1),Zaire-2007(HQ613403.1),Zaire-2008(HQ613402.1),Sudan(NC_006432) ,Reston(NC_004161.1),Cote d’Ivoire(NC_014372.1),and Bundibugyo(NC_014373.1) |
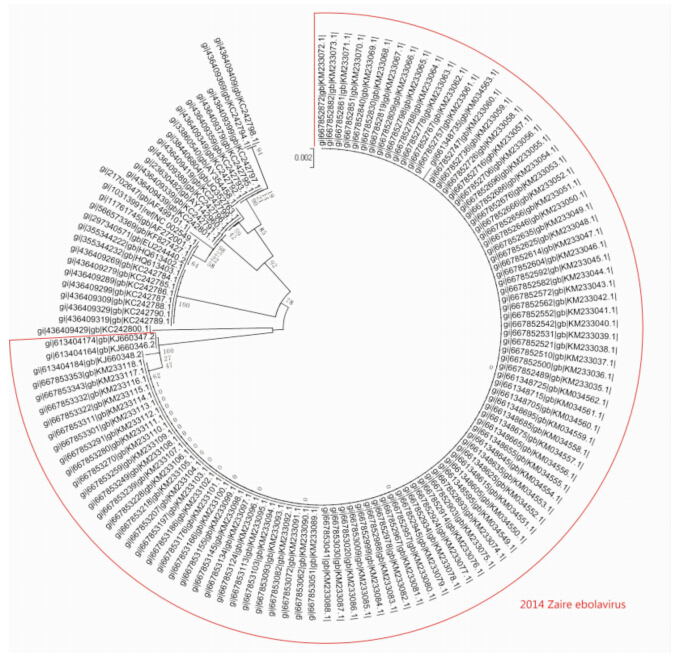
我们采用近邻结合法(neighbor-joining),根据序列相似性绘制了扎伊尔型EBOV毒株GP蛋白的进化树(图 2)。总体来说,扎伊尔型EBOV不同毒株之间GP蛋白同源性较高,但2014毒株的GP蛋白与既往扎伊尔型EBOV毒株(除2002年毒株KC242800.1以外)在种系发生上还是分属于不同分支的,这与每一次埃博拉疫情暴发都是一次独立的动物疫源性事件的假说[4]相符。我们的结果提示,扎伊尔型2014毒株与2002毒株的GP蛋白在进化上的亲缘关系较近,这与Dudas 等[5]学者的研究结果一致。
|
图 2 扎伊尔型EBOV毒株GP蛋白的进化树 Fig 2 Phylogenetic analysis of Zaire EBOV protein GP All the sequences of EBOV 2014 (marked in red),together with EBOV 2002 (KC242800.1) belong to a divergent lineage |
我们通过对EBOV的GP蛋白受体结合区(receptor binding region,RBR,位于GP蛋白第57~第149位氨基酸)结构的分析发现,GP蛋白第82位氨基酸位于RBR的α螺旋处(图 3)。EBOV 2014扎伊尔型毒株的GP蛋白第82位氨基酸由丙氨酸突变为缬氨酸,由于缬氨酸带有较大的疏水侧链,我们推测这可能将影响α螺旋的稳定性,进而影响GP蛋白与受体的结合。
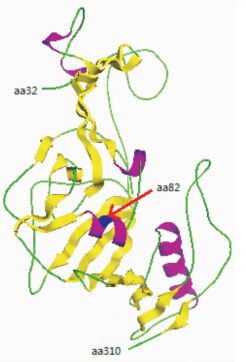
|
图 3 GP蛋白的三维构型模拟 Fig 3 3D modeling of EBOV protein GP (amino acid 32-310) The alpha helixes are colored by purple and the aa82 is marked in blue (red arrow) |
EBOV属于RNA病毒,由于其编码的RNA依赖的RNA聚合酶缺乏校正功能,在病毒基因组复制过程中容易产生碱基的错配。Gire等[4]的研究表明,2014年暴发的疫情中,EBOV基因组突变的速率加快,大约为无疫情暴发时的2倍。因此,我们有必要对EBOV 2014扎伊尔型毒株的基因组变异特征进行深入分析,实时跟踪基因组序列的变异,为此次疫情的分子流行病学研究提供一定的理论支持。
本研究对NCBI公共数据库中现有的扎伊尔型EBOV全基因组序列,包括102条2014年EBOV毒株和此前的28条全基因组序列进行了分析。我们发现,EBOV 2014扎伊尔型毒株基因组中共有606个SNVs,其中378个SNVs分布在基因组的编码区,228个分布在非编码区,而编码区中有49个SNVs是EBOV 2014扎伊尔型毒株所特有的、并导致所编码的氨基酸发生非同义突变。这49个SNVs中,尤其值得注意的是基因组第852、6 283和17 431位碱基的变异,它们分别导致NP蛋白第128位氨基酸由赖氨酸突变为精氨酸、GP蛋白第82位氨基酸由丙氨酸突变为缬氨酸和L蛋白第1 951位氨基酸由异亮氨酸突变为缬氨酸。这3个位点的氨基酸,不仅在2014年之前所有的扎伊尔型EBOV毒株中是保守不变的,而且在EBOV各亚型之间也是高度保守的,但在EBOV 2014扎伊尔型毒株中却发生了高度重现的变异。
EBOV的GP基因,通过不同的RNA编辑方式,形成3种不同的转录本,分别编码GP、sGP和ssGP 3种蛋白。GP蛋白是其中最大的一种,其前体被切割为GP1和GP2两个亚基,GP2通过跨膜区锚定于病毒包膜上,而GP1则位于膜外,两者通过二硫键相连,并形成同源三聚体的成熟GP蛋白。GP蛋白为已知的、唯一位于病毒颗粒表面的结构蛋白,是最有可能诱导产生中和抗体的病毒蛋白,也是病毒入侵宿主细胞所必需的一种蛋白质[6, 7, 8]。sGP是一种非结构性的可溶蛋白,作为GP的早期产物被大量分泌,可能通过抑制中性粒细胞的早期活化而干扰宿主免疫系统对病毒的杀伤作用[9, 10]。ssGP也是一种分泌型蛋白,功能未知。GP蛋白的第57~第149位氨基酸形成了一个受体结合区,负责与被感染细胞表面相应的受体如TIM-1结合,介导病毒进入宿主的T或B淋巴细胞、自然杀伤细胞及中性粒细胞等[6, 7, 8, 11, 12, 13, 14]。本研究结果表明,EBOV 2014扎伊尔型毒株中,位于GP蛋白受体结合区内的第82位氨基酸由丙氨酸突变为缬氨酸。我们推测,由于缬氨酸带有较大的疏水侧链,可能将影响其所在的α螺旋的稳定性,进而导致GP蛋白与受体的结合能力下降,感染宿主细胞的能力随之降低,这可能是EBOV 2014毒株致死能力减弱的原因之一。此外,由于EBOV的GP、sGP和ssGP蛋白的N端第1~第295位氨基酸是完全相同的(即共用相同的一段开放阅读框),第82位氨基酸变异也有可能影响sGP和ssGP蛋白功能。EBOV感染试验性治疗药物ZMapp可给实验方法感染EBOV的非人灵长类动物带来生存获益,并且在紧急的原则下已被用于一些EBOV感染患者的治疗[15]。该药物由3种单克隆抗体混合制成,作用的靶点是GP蛋白上的3个抗原表位,其中一种单克隆抗体13C6针对的抗原表位是GP蛋白第1~第501位氨基酸。GP蛋白第82位氨基酸位于与13C6抗体结合的抗原表位区域内,其变异是否会影响抗体药物的疗效还有待于进一步的研究[16]。
EBOV的NP蛋白在所有不分节段的单股负链RNA病毒的NP蛋白中是最大的,由739个氨基酸组成,可分为N端疏水区(大约350个氨基酸)和C端亲水区两部分[17]。研究表明,NP蛋白的第1~第450位氨基酸对于NP-NP相互作用(自组装)、病毒核衣壳样结构的形成和病毒基因组的复制是至关重要的[18]。EBOV 2014毒株的NP蛋白第128位氨基酸由赖氨酸突变为精氨酸,侧链由氨基变为胍基,可能对NP蛋白的上述功能产生一定的影响。
EBOV的RNA合成需要NP、VP35、VP30和L蛋白的共同作用。已知L蛋白的第1~第380位氨基酸是其与VP35蛋白相互作用所必需,第1~第450位氨基酸是其自身形成同源寡聚结构域所必需[19]。EBOV 2014毒株的L蛋白第1951位氨基酸由异亮氨酸突变为缬氨酸后,其疏水性有所减弱,但是否对L蛋白的功能产生影响,还有待于后续的实验证明。
本研究结果提示,EBOV 2014扎伊尔型毒株基因组具有独特的变异特征,使得NP、GP和L蛋白发生变异,特别是GP蛋白第82位氨基酸由丙氨酸突变为缬氨酸,将影响其所在的α螺旋的稳定性,还有可能影响GP蛋白与试验性抗体类治疗药物的结合。这些变异特征是否与此次疫情中病毒的流行病学特征改变(如病毒的致死能力减弱、人际传播能力增强)相关,还需要进一步的研究证实。
| [1] | Nakayama E,Saijo M. Animal models for Ebola and Marburg virus infections[J].Front Microbiol,2013,4:267. |
| [2] | Kurtz S,Phillippy A,Delcher A L,Smoot M,Shumway M,Antonescu C,et al.Versatile and open software for comparing large genomes[J].Genome Biol,2004,5:R12. |
| [3] | Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28:2731-2739. |
| [4] | Gire S K,Goba A,Andersen K G,Sealfon R S,Park D J,Kanneh L,et al.Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak[J].Science,2014,345:1369-1372. |
| [5] | Dudas G,Rambaut A.Phylogenetic analysis of Guinea 2014 EBOV Ebolavirus outbreak[J].PLoS Curr,2014:6. |
| [6] | Wool-Lewis R J,Bates P.Characterization of Ebola virus entry by using pseudotyped viruses:identification of receptor-deficient cell lines[J].J Virol,1998,72:3155-3160. |
| [7] | Lee J E,Saphire E O.Ebolavirus glycoprotein structure and mechanism of entry[J].Future Virol,2009,4:621-635. |
| [8] | Takada A,Robison C,Goto H,Sanchez A,Murti K G,Whitt M A,et al.A system for functional analysis of Ebola virus glycoprotein[J].Proc Natl Acad Sci USA,1997,94:14764-14769. |
| [9] | Yang Z,Delgado R,Xu L,Todd R F,Nabel E G,Sanchez A,et al.Distinct cellular interactions of secreted and transmembrane Ebola virus glycoproteins[J].Science,1998,279:1034-1037. |
| [10] | Kindzelskii A L,Yang Z,Nabel G J,Todd R F 3rd,Petty H R.Ebola virus secretory glycoprotein (sGP) diminishes Fc gamma RIIIB-to-CR3 proximity on neutrophils[J].J Immunol,2000,164:953-958. |
| [11] | Geisbert T W,Hensley L E,Gibb T R,Steele K E,Jaax N K,Jahrling P B.Apoptosis induced in vitro and in vivo during infection by Ebola and Marburg viruses[J].Lab Invest,2000,80:171-186. |
| [12] | Kuhn J H,Radoshitzky S R,Guth A C,Warfield K L,Li W,Vincent M J,et al.Conserved receptor-binding domains of Lake Victoria marburgvirus and Zaire ebolavirus bind a common receptor[J].J Biol Chem,2006,281:15951-15958. |
| [13] | Dube D,Brecher M B,Delos S E,Rose S C,Park E W,Schornberg K L,et al.The primed ebolavirus glycoprotein (19-kilodalton GP1,2):sequence and residues critical for host cell binding[J].J Virol,2009,83:2883-2891. |
| [14] | Kondratowicz A S,Lennemann N J,Sinn P L,Davey R A,Hunt C L,Moller-Tank S,et al.T-cell immunoglobulin and mucin domain 1 (TIM-1) is a receptor for Zaire Ebolavirus and Lake Victoria Marburgvirus[J].Proc Natl Acad Sci USA,2011,108:8426-8431. |
| [15] | Qiu X,Wong G,Audet J,Bello A,Fernando L,Alimonti J B,et al.Reversion of advanced Ebola virus disease in nonhuman primates with ZMapp[J].Nature,2014,514:47-53. |
| [16] | Kugelman J R,Sanchez-Lockhart M,Andersen K G,Gire S,Park D J,Sealfon R,et al.Evaluation of the potential impact of Ebola virus genomic drift on the efficacy of sequence-based candidate therapeutics[J].MBio,2015,6(1).pii:e02227-14. |
| [17] | Sanchez A,Kiley M P,Holloway B P,McCormick J B,Auperin D D.The nucleoprotein gene of Ebola virus:cloning,sequencing,and in vitro expression[J].Virology,1989,170:81-91. |
| [18] | Watanabe S,Noda T,Kawaoka Y.Functional mapping of the nucleoprotein of Ebola virus[J].J Virol,2006,80:3743-3751. |
| [19] | Trunschke M,Conrad D,Enterlein S,Olejnik J,Brauburger K,Muhlberger E.The L-VP35 and L-L interaction domains reside in the amino terminus of the Ebola virus L protein and are potential targets for antivirals[J].Virology,2013,441:135-145. |
 2015, Vol. 36
2015, Vol. 36


