2. 国家肝癌科学中心, 上海 201805
2. National Center for Liver Cancer, Shanghai 201805, China
Co-first authors.
MicroRNAs(miRNAs)是存在于真核生物体内的一类长约22 nt的非编码RNA,其通过与靶基因mRNA的3′非翻译区(3′UTR)结合,靶向降解mRNA或抑制其翻译,从而调节生理、病理功能。研究发现,miRNAs在肿瘤中既可起到促癌作用,又可发挥抑癌功能[1, 2]。miR-429是miRNA-200s家族中的一员,同该家族其余4个成员miR-200c、miR-141、miR-200b和miR-200a一样广泛表达于不同组织的上皮细胞中[3]。miR-429在不同上皮来源的肿瘤组织中的表达与作用不尽相同,而关于miR-429与肝癌及其肿瘤干细胞的报道较少[4]。
肝细胞癌(hepatocellular carcinoma,HCC)是常见的恶性肿瘤,其发病率在男性中居第5位,在女性中居第9位[5, 6]。HCC恶性程度高,目前除甲胎蛋白(AFP)以外,尚缺乏较有效的早期筛查和诊断方法。外科切除是目前常用的治疗性方法。在我国,接受手术治疗后的HCC患者5年生存率为32.64%,而非手术治疗患者的5年生存率仅为9.01%[7]。因此,寻找与肝癌相关的癌基因,明确其作用机制,进而寻找以其为靶点的化学、基因治疗药物,显得尤为重要。EpCAM作为黏附分子家族中的一员,具有调节细胞信号转导、迁移、增殖和分化等功能[8],其通过参与β连环蛋白依赖的Wnt级联反应,激活c-myc、cyclinA/E等基因的表达,从而具有致瘤作用[9]。 在肝癌中,EpCAM被认为是肿瘤干细胞的标记物之一,EpCAM阳性的肝癌干细胞能够诱导免疫缺陷的小鼠发生肿瘤[10]。本研究靶向特定的HCC细胞群体,将HCC细胞分为EpCAM阳性和阴性两群,探索在HCC的发生发展过程中,miR-429对HCC细胞增殖活性、分化潜能以及炎性浸润的影响。
1 材料和方法1.1 材料
SPF级NOD-SCID小鼠购自上海斯莱克实验动物有限责任公司[许可证号SCXK(沪)2012-0002];人肝癌细胞系HCCLM3购自中国科学院上海生命科学研究院生物化学与细胞生物学研究所。Taqman microRNA反转录试剂盒(人类)为美国应用生物公司(Applied Biosystems)产品;小RNA过表达拟态物(miR-mimics)及其对照(negative control miR-mimics)、小RNA拮抗剂(antago-miR)及其对照(negative control antago-miR)均购自广州锐博生物科技公司(miR-10000);INTERFERin in vitro siRNA转染试剂、JetPEI转染试剂购自Poly plus公司(409-50); PCR引物使用相关软件BioXM 2.6自行设计,并由生工生物工程(上海)股份有限公司合成;总RNA抽提液TRIzol Reagent购自Invitrogen公司;过氧化氢(H2O2)购自Sigma-Aldrich公司; DMEM培养基、小牛或胎牛血清 (FCS/FBS)购自 Gibco BRL公司;EpCAM流式检测、磁珠分选抗体购自美天旎公司(Miltenyi Biotech);辣根过氧化物酶标记的羊抗兔以及羊抗小鼠二抗购自华舜公司;荧光二抗购自Invitrogen公司。超净工作台购自苏州净化设备公司;免疫组化石蜡包埋机、制片仪、冰冻切片仪均购自Leica公司;荧光实时定量PCR仪(7900HT)购自Applied Biosystems公司;荧光显微镜(Olympus IX70)购自Olympus公司;细胞培养孵箱购自Thermo公司;多功能酶标仪购自Bio Tek公司;流式细胞仪(型号:Moflo XDP)购自Beckman公司。 1.2 免疫磁珠方法分选HCCLM3细胞系
取生长状态良好的HCCLM3细胞,用胰酶消化法制备单细胞悬液,136×g离心3 min;弃上清,PBS清洗,136×g 离心3 min,弃上清;用300 μL PBS重悬,加入6 μL EpCAM抗体(1∶50),混匀,4℃ 孵育15 min,每5 min混匀1次;136×g离心3 min;弃上清,PBS清洗,136×g离心3 min,弃上清;用200 μL磁珠缓冲液重悬,加入30 μL免疫磁珠(抗兔),混匀,4℃ 孵育15 min,每5 min混匀1次;136×g离心3 min;弃上清,缓冲液清洗,136×g离心3 min,弃上清;准备好分离柱,并用500 μL缓冲液清洗3次,500 μL缓冲液重悬细胞,缓慢加入到分离柱中(避免产生气泡),收集流出液体,得到EpCAM阴性细胞;将分离柱移出磁场,加500 μL分选缓冲液至分离柱,用力推出,得到EpCAM阳性细胞。 1.3 Real-time PCR检测转染后细胞中miR-429表达
磁珠分选出EpCAM阳性HCCLM3细胞和EpCAM阴性HCCLM3细胞,种于6孔板中; EpCAM阳性HCCLM3细胞用40 μmol/L的AntagomiR-429处理1 d,EpCAM阴性HCCLM3细胞以100 nmol/L的miR-429mimic处理1 d;收集细胞,TRIzol裂解法提取总RNA,取1 μg RNA反转录制备cDNA,使用miR-429特异性的反转录引物进行反转录,然后取1 μL反转录产物进行PCR检测,实验使用U6作为PCR内参。PCR反应总体系:反转录产物0.5 μL,上、下游引物(10 μmol/L)各1 μL,2×Taqman Unibersal PCR Master Mix 12.5 μL,双蒸水补足反应体系至20 μL。反应条件:95℃ 4 min;95℃ 15 s、58℃ 30 s、72℃ 35 s,共40个循环。应用Applied Biosystems 7300 Fast Real-Time PCR System 软件分析结果,用2-ΔΔCt法分析基因相对表达量。 1.4 流式细胞术检测转染后EpCAM阳性细胞的比例
将转染24 h后的细胞,用胰酶消化法制备单细胞悬液,136×g离心3 min;弃上清,PBS清洗,136×g离心3 min,弃上清;用100 μL PBS重悬,加入10 μL EpCAM直标抗体(1∶10,APC通道),并设置空白对照,混匀,4℃ 孵育15 min,每5 min混匀1次;136×g离心3 min;弃上清,PBS清洗,136×g离心3 min;500 μL PBS重悬,利用Beckman流式细胞仪FL/8通道检测不同处理组细胞表面EpCAM比例。 1.5 CCK8法检测miR-429对肝癌细胞增殖活性的影响
将磁珠分选的EpCAM阳性和阴性HCCLM3细胞种于6孔板中;分别用40 μmol/L的Antagomir-429和100 nmol/L miR-429mimic处理1 d;取对数生长期细胞,0.5%胰酶消化3 min,136×g室温离心3 min,加入适量培养基制成单细胞悬液;计数细胞,按每孔5 000个细胞接种于96孔板中,每组设置0、24、48、72、84 h 5个时间点,每个时间点设3个重复处理孔;使细胞分布均匀,37℃、5% CO2培养;到待测时间点时,按说明书每孔加入CCK-8试剂 10 μL,继续培养1 h,用分光光度计测其在450 nm处的光密度值。 1.6 NOD-SCID小鼠荷瘤实验验证miR-429对肿瘤细胞成瘤能力的影响
磁珠分选出EpCAM阳性和EpCAM阴性HCCLM3细胞,种于6孔板中;EpCAM阳性HCCLM3细胞用40 μmol/L的AntagomiR-429处理1 d,EpCAM阴性HCCLM3细胞以miR-429mimic处理1 d;用0.5%的胰酶消化细胞,136×g离心3 min,PBS重悬细胞;计数细胞并调整细胞密度,使每100 μL PBS 中含2×104或4×104个细胞;将含2×104的EpCAM阳性HCCLM3细胞或含4×104的EpCAM阴性HCCLM3细胞的100 μL PBS 分别种植到NOD-SCID小鼠躯干右侧皮下。记录肿瘤发生情况。肿瘤细胞种植6周成瘤后,进行活体呈像,然后断颈处死动物,收集肿瘤样本及血浆样本。肿瘤体积的计算公式如下:肿瘤体积 (cm3)=宽2 (cm2)×长 (cm)/2。 1.7 免疫组织化学法检测miR-429对肿瘤细胞分化潜能及炎性浸润的影响
取荷瘤NOD-SCID小鼠体内的瘤体组织,石蜡包埋,切片;将石蜡切片60℃烤箱过夜后脱蜡至水,置于3% H2O2甲醇液去除内源性过氧化物酶;微波修复抗原;1%BSA封闭。甩去封闭液,滴加Ki67、AFP、F4/80一抗,置于湿盒中,并放置于4℃冰箱过夜;取出后室温下复温,0.01 mol/L PBS洗4次;滴加DAKO二抗,放于湿盒中,置于37℃孵育45 min;0.01 mol/L PBS洗4次,DAB显色,甲基绿复染,无水乙醇脱色;二甲苯脱水、透明,滴加中性树脂,盖玻片覆盖并封片;显微镜下观察Ki67、AFP、F4/80的阳性染色。 1.8 统计学处理 应用SPSS 13.0 统计软件进行分析。计量资料以x±s 表示,采用非配对t检验比较两组间的差异,用方差分析比较多组间的差异。检验水准(α)为0.05。 2 结 果 2.1 miR-429有助于维持肝癌细胞EpCAM阳性表型
将EpCAM阳性细胞分组并分别转染miR-429拮抗剂及其对照,将EpCAM阴性细胞分组并分别转染miR-429过表达拟态物及其对照。24 h后,转染了AntagomiR-429的EpCAM阳性细胞中miR-429表达受到抑制(降低约90%,P=0.016),同时转染了miR-429拟态物的EpCAM阴性细胞中miR-429的表达升高(升高约100倍,P=0.001 7),证实转染效率良好(图 1A)。同时通过流式细胞技术检测不同处理组EpCAM阳性细胞比例,结果发现:抑制miR-429表达后,EpCAM阳性细胞的比例由84.65%降至49.32%;反之,经过miR-429过表达,EpCAM阳性细胞的比例由2.72%上升至37.46%(图 1B)。结果提示miR-429对于EpCAM阳性表型的维持很重要。
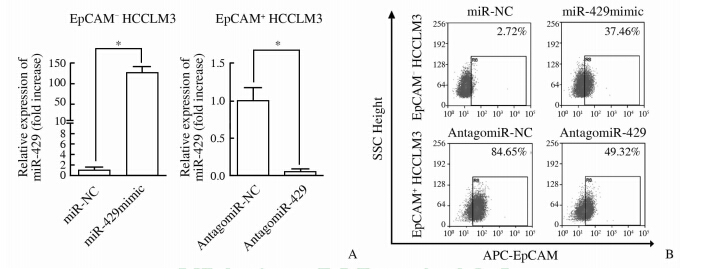 | 图 1 细胞瞬时转染效果鉴定及对EpCAM表型的影响 Fig 1 Identification of tansfection efficiency and influence on EpCAM phenotype A: Identification of transfection by RT-PCR (P<0.05. n=3,x±s ); B:EpCAM-positive cell population with different treatments by flow cytometry |
小鼠皮下荷瘤实验证实,EpCAM阳性HCCLM3形成肿瘤所需的癌细胞数(Antagomir-NC处理,20 000个细胞)远少于EpCAM阴性细胞(miR-NC处理,40 000个细胞),并且肿瘤发生的时间更早,发生肿瘤的比例更高。然而,抑制miR-429表达后,EpCAM阳性细胞的肿瘤发生率降低,出现肿瘤的时间延迟,瘤体减小,肿瘤生长缓慢;而过表达miR-429后,EpCAM阴性细胞的肿瘤发生率升高,出现肿瘤的时间由原来的6周变为3周,瘤体明显增大,肿瘤生长速度增快(表 1,图 2)。结果说明miR-429能增强肝癌细胞的体内成瘤能力。
|
|
表 1 产生合成记录所用参数 Tab 1 Single-variable logistic regression analysis of factors influencing HBV infection of newborns |
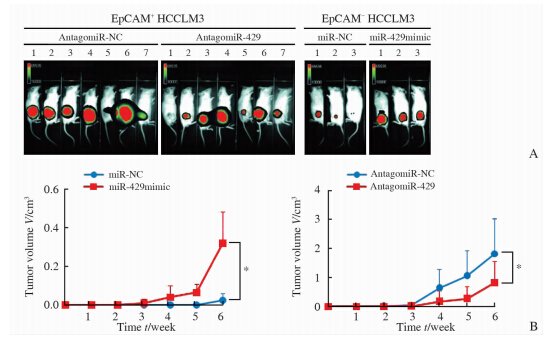 | 图 2 miR-429对肝癌细胞体内成瘤能力的影响 Fig 2 Influence of miR-429 on tumorigenesis of hepatocellular carcinoma cell lines in vivo A: Live-imaging of tumors in different groups; B: Volume variation of tumors in image A at different weeks (P<0.05. n=3 for miR-NC and miR-429mimic groups; n=7 for AntagomiR-NC and AntagomiR-429 groups. x±s ) |
将瘤体组织从小鼠皮下分离,连续切片行H-E染色及巨噬细胞F4/80免疫组化染色,结果显示:相比对照,抑制miR-429表达后的EpCAM阳性HCCLM3细胞成瘤组F4/80表达的巨噬细胞浸润减少;而过表达miR-429的EpCAM阴性HCCLM3细胞成瘤组与对照相比,F4/80表达的巨噬细胞浸润增加(图 3)。结果提示在miR-429参与的肝癌形成过程中,其微环境中的巨噬细胞等炎性细胞浸润增多。
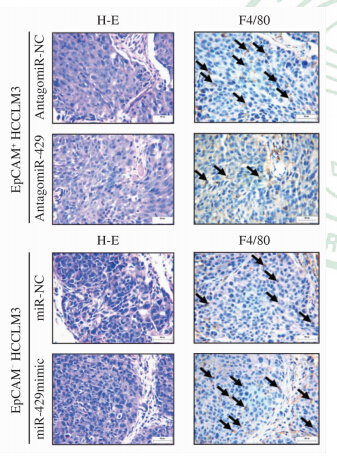 | 图 3 免疫组化分析不同瘤体组织中F4/80阳性细胞 Fig 3 Immunohistochemical analysis of F4/80-positive cells in tumors of different treatment groups Black arrows showing macrophages stained with F4/80. Original magnification:×200 |
在体外水平,EpCAM阳性HCCLM3的增殖速度明显高于EpCAM阴性HCCLM3细胞。抑制miR-429表达后,EpCAM阳性细胞增殖明显变慢;而过表达miR-429后,EpCAM阴性细胞的增殖变快(图 4)。结果说明miR-429不仅有助于维持EpCAM阳性肝癌细胞的高增殖能力,还能部分逆转EpCAM阴性HCCLM3细胞增殖缓慢的现象。在体内成瘤实验中,对皮下肿瘤同一部位连续切片进行免疫组化染色,结果显示:抑制miR-429表达的EpCAM阳性HCCLM3细胞成瘤组Ki67(反映增殖活性)、AFP(反映分化潜能)阳性染色与对照相比均降低,而过表达miR-429的EpCAM阴性HCCLM3细胞成瘤组Ki67、AFP阳性染色与对照相比均升高(图 5)。结果提示miR-429的过表达与高增殖、低分化相关。
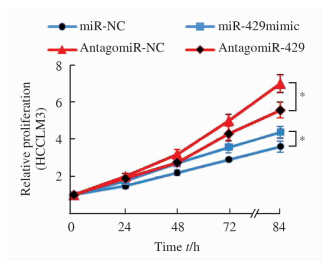 |
图 4 miR-429对肝癌细胞体外增殖的影响
Fig 4 Influence of miR-429 on proliferation of HCC cell lines in vitro
cell lines in vitro P<0.05. n=3,x±s |
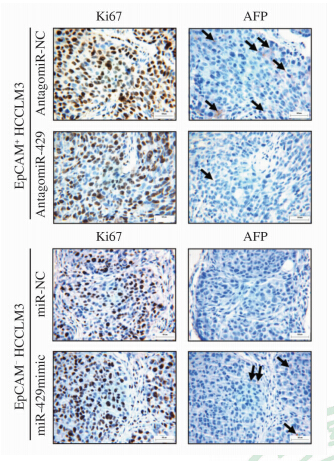 | 图 5 免疫组化显示不同瘤体组织中Ki67、AFP的表达 Fig 5 Immunohistochemical analysis of Ki67 and AFP expression in tumors from different groups Black arrows showing Ki67 or AFP positive cells. AFP: Alpha fetoprotein. Original magnification:×200 |
肝癌是消化系统常见肿瘤,恶性程度较高,是病死率最高的恶性肿瘤之一。我国是肝癌的高发国家,又是病死率最高的国家,以上海市社区人群为基础的研究显示,我国每年男性和女性人群中的发病率分别达到37.9/10万和14.2/10万[7],严峻的形势给我国的社会和医疗带来了沉重的负担。对于肝癌发病机制的研究目前主要集中在遗传改变与表观修饰,它们通过调节不同的信号转导通路而致癌[11, 12]。近来,二代测序等技术的应用进一步证实了miRNAs调控的信号通路在肝癌发病中发挥重要作用,miRNAs 可以作为肝癌治疗的潜在靶点[13, 14, 15]。研究证实,相比肝脏癌旁组织,某些特定miRNAs在癌组织中特异性低表达或高表达[16],细胞学和分子学实验证实 miRNAs在肝脏组织中的异常表达影响了肿瘤相关信号通路的活性,从而进一步影响了癌症的发生与发展[17, 18]。
多项肿瘤相关研究证实,患者体内的1号染色体长臂3区6带的稳定性较差,而miR-200b/miR-200a/miR-429家族正位于此区域。本课题组之前的研究发现miR-429在EpCAM阳性的肝癌细胞及具有成球能力的肝癌起始细胞中高表达,可作为独立的预后影响因素促进肝脏肿瘤的发生与生长,并且miR-429启动子区的4个甲基化位点的异常会直接导致miR-429在肝癌细胞系及临床样本中呈高表达状态[4]。EpCAM在肝癌中可以作为肝癌干细胞的标记物,并且和肝癌细胞的高度异质性相关。EpCAM阳性和EpCAM阴性的肝癌细胞在形态学和功能学方面有着显著差异,例如,高表达EpCAM的肝癌细胞呈现较强的增殖能力和干细胞特性[10]。本实验通过磁珠分选人肝癌细胞系HCCLM3得到EpCAM阳性及阴性的细胞,将分选后的EpCAM阳性、EpCAM阴性细胞分别转染AntagomiR-429及miR-429mimic,进行miR-429的干扰和过表达。以此为研究工具,我们发现抑制miR-429表达会导致EpCAM阳性细胞比例、增殖能力、去分化水平及体内成瘤能力下降,而过表达miR-429则使EpCAM阴性肝癌细胞中出现EpCAM阳性细胞,并伴随着增殖能力、去分化水平及体内成瘤能力的升高,说明miR-429可能通过调节肝癌细胞中EpCAM阳性细胞的比例来促进肝脏肿瘤的发生、发展。
近年来,肿瘤细胞与肿瘤微环境中的炎性细胞、成纤维细胞之间的交互对话逐渐得到人们的关注。肿瘤微环境中的巨噬细胞可通过分泌趋化因子18、骨桥蛋白等与乳腺癌、结直肠癌中的肿瘤细胞相互作用,形成正反馈环路,促进肿瘤的生长、转移[19, 20]。在肝脏中,乙肝病毒介导的肝病组织中往往出现大量的M2型巨噬细胞浸润[21],然而,却缺少足够的证据说明肝癌细胞与其相关巨噬细胞之间的相互作用可以直接影响肝癌的发生发展。本实验发现,在miR-429过表达的瘤体组织内,巨噬细胞的浸润显著增多;而抑制miR-429表达的瘤体内,巨噬细胞的浸润明显减少,提示miR-429在肝脏肿瘤细胞表达可能有助于为其招募相关的巨噬细胞,从而进一步促进肿瘤的发生、发展。我们之前的实验研究发现miR-429可被分泌到胞外[4],因此,我们推测:EpCAM 阳性的肝癌细胞通过分泌miR-429,促进巨噬细胞的迁徙能力而诱导其浸润,巨噬细胞通过分泌炎性细胞因子促进肿瘤细胞的增殖。但该推测有待更进一步实验证实。
在前期研究中,我们对miR-429调控EpCAM阳性肝脏肿瘤起始细胞的作用靶基因及相关信号转导通路进行了深入研究。结果发现,通过抑制其靶基因RB结合蛋白4(RBBP4)的表达,miR-429能够释放出E2F1的转录活性,进一步促进OCT4的表达,从而维持EpCAM阳性肝脏肿瘤起始细胞的特性[4]。因此,我们认为,miR-429很可能是通过RBBP4/E2F1/OCT4轴调节肝脏肿瘤的炎性微环境、分化以及增殖。
近年来,以miRNA或抗miRNA作为肿瘤治疗的靶向分子研究正逐渐得到人们的关注和重视。miR-429在肝癌细胞和组织中高表达,且选择性地表达于EpCAM阳性细胞中,为寻找肝癌治疗的新靶点提供了思路。
| [1] | DeSano J T, Xu L. MicroRNA regulation of cancer stem cells and therapeutic implications[J]. AAPS J, 2009,11:682-692. |
| [2] | Esquela-Kerscher A, Slack F J. Oncomirs-microRNAs with a role in cancer[J]. Nat Rev Cancer, 2006,6:259-269. |
| [3] | Wienholds E, Kloosterman W P, Miska E, Alvarez-Saavedra E, Berezikov E, de Bruijn E, et al. MicroRNA expression in zebrafish embryonic development[J]. Science,2005,309:310-311. |
| [4] | Li L, Tang J, Zhang B, Yang W, LiuGao M, Wang R, et al. Epigenetic modification of MiR-429 promotes liver tumor-initiating cell properties by targeting Rb binding protein 4[J]. Gut,2015,64:156-167. |
| [5] | Jemal A, Bray F, Center M M, Ferlay J, Ward E, Forman D. Global cancer statistics[J]. CA Cancer J Clin,2011,61:69-90. |
| [6] | El-Serag H B. Hepatocellular carcinoma[J]. N Engl J Med, 2011, 365:1118-1127. |
| [7] | 韩 雪,黄辰曦,张宏伟,乔 鹏,谢 梦,张 蓉,等. 2002-2010年上海市杨浦区居民原发性肝癌发病及生存情况[J].中华预防医学杂志,2012,46:119-124. |
| [8] | Kurtz J E, Dufour P. Adecatumumab: an anti-EpCAM monoclonal antibody, from the bench to the bedside[J].Expert Opin Biol Ther,2010,10:951-958. |
| [9] | Maetzel D, Denzel S, Mack B, Canis M, Went P, Benk M, et al. Nuclear signalling by tumour-associated antigen EpCAM[J].Nat Cell Biol, 2009,11:162-171. |
| [10] | Kimura O, Takahashi T, Ishii N, Inoue Y, Ueno Y, Kogure T, et al. Characterization of the epithelial cell adhesion molecule(EpCAM)+cell population in hepatocellular carcinoma cell lines[J]. Cancer Sci, 2010,101:2145-2155. |
| [11] | Gramantieri L, Fornari F, Callegari E, Sabbioni S, Lanza G, Croce C M, et al. MicroRNA involvement in hepatocellular carcinoma[J]. J Cell Mol Med, 2008, 12: 2189-2204. |
| [12] | Aravalli R N, Cressman E N, Steer C J. Cellular and molecular mechanisms of hepatocellular carcinoma: an update[J]. Arch Toxicol, 2013, 87: 227-247. |
| [13] | Guichard C, Amaddeo G, Imbeaud S, Ladeiro Y, Pelletier L, Maad I B, et al. Integrated analysis of somatic mutations and focal copy-number changes identifies key genes and pathways in hepatocellular carcinoma[J]. Nat Genet, 2012, 44: 694-698. |
| [14] | Cleary S P, Jeck W R, Zhao X, Chen K, Selitsky S R, Savich G L, et al. Identification of driver genes in hepatocellular carcinoma by exome sequencing[J]. Hepatology, 2013, 58: 1693-1702. |
| [15] | Kan Z, Zheng H, Liu X, Li S, Barber T D, Gong Z, et al. Whole-genome sequencing identifies recurrent mutations in hepatocellular carcinoma[J]. Genome Res, 2013, 23: 1422-1433. |
| [16] | Negrini M, Gramantieri L, Sabbioni S, Croce C M. microRNA involvement in hepatocellular carcinoma[J]. Anticancer Agents Med Chem, 2011, 11: 500-521. |
| [17] | Negrini M, Ferracin M, Sabbioni S, Croce C M. MicroRNAs in human cancer: from research to therapy[J]. J Cell Sci, 2007, 120(Pt 11): 1833-1840. |
| [18] | Gailhouste L, Ochiya T. Cancer-related microRNAs and their role as tumor suppressors and oncogenes in hepatocellular carcinoma[J]. Histol Histopathol, 2013, 28: 437-451. |
| [19] | Rao G, Wang H, Li B, Huang L, Xue D, Wang X,etal. Reciprocal interactions between tumor-associated macrophages and CD44-positive cancer cells via osteopontin/CD44 promote tumorigenicity in colorectal cancer[J]. Clin Cancer Res, 2013,19:785-797. |
| [20] | Su S, Liu Q, Chen J, Chen J, Chen F, He C, etal. A positive feedback loop between mesenchymal-like cancer cells and macrophages is essential to breast cancer metastasis[J]. Cancer Cell, 2014,25:605-620. |
| [21] | Yang P, Markowitz G J, Wang X F. The hepatitis B virus-associated tumor microenvironment in hepatocellular carcinoma[J]. Natl Sci Rev, 2014: 396-412. |
 2015, Vol. 36
2015, Vol. 36


