2. 上海中医药大学附属上海市中医医院肿瘤科, 上海 200071
2. Department of Oncology, Shanghai Municipal Hospital of Traditional Chinese Medicine, Shanghai University of Traditional Chinese Medicine, Shanghai 200071, China
胃癌作为世界范围内最常见的恶性肿瘤之一,每年导致数以百万计的患者死亡[1, 2, 3],其中在中国,胃癌死亡率一直居恶性肿瘤的首位[4, 5]。胃癌是多因素交叉作用后引起的病理性疾病,与许多癌基因和抑癌基因都有着密切的联系[6, 7, 8]。随着对肿瘤相关基因研究的不断深入,基因敲除动物模型在肿瘤研究中的应用也越发受到重视[9, 10]。基于基因敲除动物模型在胃癌发病机制研究中有独特的优势,本课题旨在利用国际上最为先进,并且方便、快捷、高效的CRISPR/Cas9基因编辑技术[11, 12, 13, 14, 15, 16],以miR-301a为例,建立miRNA基因敲除小鼠模型,为后续研究miRNA在胃癌发病过程中的作用提供重要的研究工具。 1 材料和方法 1.1 动物及试剂
C57BL/6小鼠,4~6周龄,SPF级,购自上海斯莱克实验动物有限责任公司,许可证号SCXK(沪)2012-0002。小鼠饲养于上海中医药大学实验动物室12 h∶12 h照明节律、食物与饮水充足的SPF环境。NcoⅠ限制性内切酶、T4 DNA连接酶、Q5 PCR聚合酶、T7 endonuclease Ⅰ、dNTPs均购自NEB公司;mMESSAGE mMACHINE® SP6体外转录试剂盒购自Life Technologies公司;体外转录T7试剂盒购自TaKaRa公司; DH5α感受态细胞购自天根公司;pEASY-Blunt克隆载体购自北京全式金公司;蛋白酶K购自Calbiochem公司;氨苄青霉素、琼脂A粉末、Tris、SDS、EDTA购自上海生工生物工程有限公司;蛋白胨、酵母提取物购自Sigma公司;pX260载体由华东师范大学生命医学研究所惠赠;PCR仪购自Applied Biosystems公司;MX3005型real-time PCR仪购自Stratagene公司;DNA电泳系统购自Bio-Rad公司;凝胶成像分析系统购自天能公司。 1.2 引导RNA(guideRNA,gRNA)体外转录模板DNA的获得针对miR-301a靶向敲除靶点,设计并合成2条引物,其中1条引物序列含有用于体外转录的T7启动子序列(蓝色)、靶点特异性序列(绿色)以及gRNA骨架序列的前一部分(青色),另一条引物含有gRNA骨架序列的其余部分(图 1)。两条引物的3′端存在互补配对序列。利用这对引物进行PCR扩增即可得到用来体外转录的模板DNA。
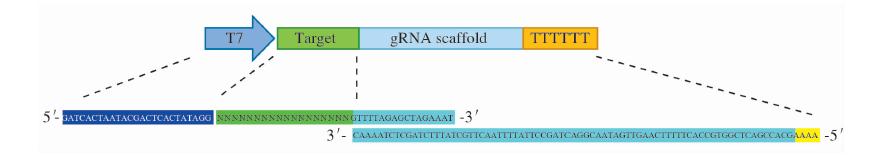 | 图 1 guideRNA体外转录模板的构建示意图 Fig 1 Construction diagram of in vitro transcription template of guideRNA |
由于质粒pX260中Cas9起始密码子ATG恰好是位于此处的NcoⅠ(CCATGG)酶切位点的一部分,因此可以通过该酶切位点引入用于体外转录Cas9的SP6启动 子序列。通过合成带有5′磷酸基团的如下两段引物:NcoⅠ-SP6-Sense: CATGG ATTTAGGTGACACTATAGAAGAG C ,NcoⅠ-SP6-Antisense: CATGG CTCTTCTATAGTGTCACCTAAAT C 。 下划线部分代表NcoⅠ(CCATGG)酶切位点。经过等比例混合,退火后即可形成两端各带有NcoⅠ黏性末端,中间序列为SP6启动子的双链DNA。同时用NcoⅠ处理pX260质粒,并去磷酸化,而后与上述退火产物进行连接、转化,经测序鉴定方向,即可得到可被SP6体外转录试剂盒作为模板的Cas9质粒。 1.4 Cas9及gRNA的体外转录
PCR产物形式的gRNA模板通过酚-氯仿抽提和乙醇沉淀进行纯化,使用体外转录T7试剂盒进行转录,并通过酚-氯仿抽提和异丙醇沉淀进行回收。使用NotⅠ处理带有SP6启动子序列的pX260质粒(该质粒只有1个NotⅠ酶切位点,位于Cas9下游的polyA序列之后),mMESSAGE mMACHINE® SP6体外转录试剂盒进行体外转录,氯化锂沉淀进行纯化回收。 1.5 gRNA/Cas9 mRNA的显微注射
显微注射所使用的受精卵来自与同品系雄鼠交配后超排的母鼠。注射前将受精卵培养在KSOM胚胎培养基当中。利用显微注射用TE缓冲液(无核酸酶)将针对miR-301a的gRNA与Cas9 mRNA混合并稀释至12.5 ng/μL的gRNA以及25 ng/μL的Cas9。使用Eppendorf TransferMan NK2显微操作装置将上述溶液通过显微注射针头注入单细胞期的受精卵胞质中。注射后的受精卵被立即或经KSOM过夜培养后移植进假孕C57BL/6母鼠。 1.6 小鼠基因组DNA的提取及PCR扩增
新生小鼠出生约1周后,剪下脚趾,55℃水浴消化过夜后,苯酚-氯仿法提取基因组DNA。PCR反应条件:94℃ 3 min; 94℃ 30 s; 56℃ 30 s; 72℃ 45 s; 72℃ 8 min,30个循环。 1.7 利用T7E1酶切对突变进行检测
PCR产物吸5 μL点样,琼脂糖凝胶(1.5%)电泳观察有无目的条带。另外吸10 μL,T7E1酶切(酶切体系15 μL:DNA 10 μL,T7E1 0.1 μL,10×buffer 1.5 μL,H2O 3.4 μL),37℃ 60 min,点样,挑选突变测序。 2 结 果 2.1 设计针对miR-301a的gRNA序列
PAM(protospacer adjacent motif)是Cas9蛋白识别序列的必要条件,其能够聚集到PAM区域进行识别切割。利用CRISPR/Cas9 gene targeting技术,构建针对miR-301a基因的gRNA,指导Cas9蛋白在特定位点剪切DNA双链;由于miR-301a为非编码区,且序列较短,故选择设计合适靶点即可,但考虑到靶点效率及后期鉴定,选择设计双靶点(图 2)。
 | 图 2 针对miR-301a的gRNA序列 (双靶点) Fig 2 Sequence of gRNA targeting miR-301a (two targets)PAM: Protospacer adjacent motif |
PCR扩增和凝胶电泳检测结果表明:获得预期大小(732 bp)的DNA片段(图 3),并经测序确认。
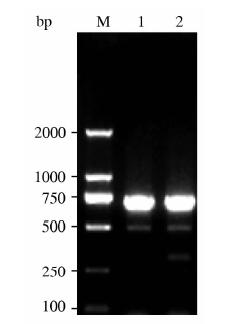 | 图 3 凝胶电泳检测PCR扩增条带 Fig 3 Gel electrophoresis assay for PCR amplification products M: Marker; 1,2: Amplified DNA fragment targeting miR-301a |
基于PCR扩增获得体外转录的模板DNA序列和实验室构建好的Cas9体外转录模板(图 4),体外转录并通过氯化锂沉淀进行纯化回收,顺利获得用于显微注射的mRNA。
 | 图 4 用于体外转录的Cas9/RNA表达载体系统 Fig 4 Cas 9/RNA expression vector system for in vitro transcriptionT7,SP6 refers to promoter sequence for in vitro transcription,respectively; Target+gRNA scaffold refers to the artificial fusion crRNA-tracrRNA complex under native conditions,in which the Target is the target-specific sequence,and the six consecutive T bases in 3′ end is the transcription termination signals; hCas9 refers to the coding sequence of Cas9 by codon optimization; NLS: Nuclear localization signal; pA: Transcription termination signal |
通过T7E1鉴定发现8只新生小鼠中有7只(87.5%)被鉴定在miR-301a位 点携带突变,除#5为WT外,其余都 携带突变(图 5)。初步证明通过向小鼠受精卵显微注 射经体外转录的Cas9 mRNA以及靶点特异性的guideRNA能够对小鼠基因进行高效突变。
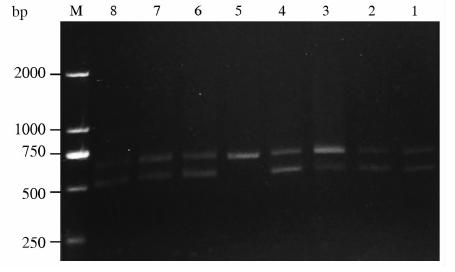 | 图 5 T7E1酶切检测miR-301a基因突变情况 Fig 5 T7E1 enzyme digestion for detecting mutations of miR-301a M:Marker; 1-4,6-8:miR-301a carrying the mutated sites; 5: miR-301a carrying no mutated sites |
结果显示,大约以PAM区域为中心,各小鼠均有不同程度的碱基插入或缺失突变,其中缺失碱基数目最多的#4 F0代小鼠中产生了31个碱基缺失,表明miR-301a在F0代小鼠体内已经实现了高效敲除。 2.6 建立miR-301a敲除小鼠
将上述经鉴定过的F0代小鼠#4与野生型小鼠进行交配,产生8只F1代小鼠。对F1代产生的小鼠进行PCR扩增和基因型鉴定,结果表明,#3、#4、#5、#7为miR-301a 杂合子小鼠(图 6A)。将#4与#5小鼠进行交配,繁殖出6只F2代小鼠,鉴定结果表明,#1和#5为miR-301a基因敲除小鼠(图 6B)。
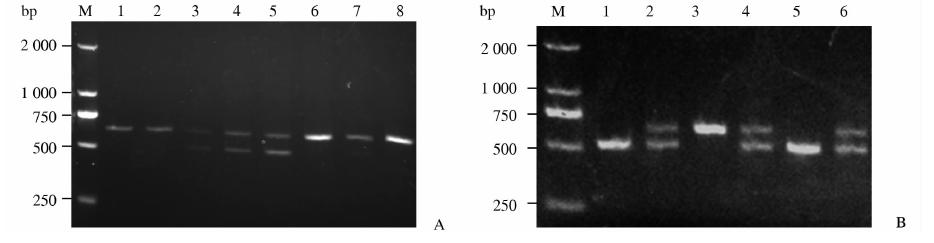 | 图 6 miR-301a F1代(A)及F2代(B)小鼠鉴定 Fig 6 Identification of miR-301a F1 pup mice (A) and F2 pup mice (B)In Fig A,M: Marker; 1,2,6,8: Wildtype mice; 3-5,7: miR-301a heterogenous mice. In Fig B,M: Marker; 3: Wildtype mice;2,4,6: miR-301a heterogenous mice; 1,5: miR-301a knockout mice |
胃癌是多因素、多步骤作用后引起的病理性疾病,其发病机制与许多癌基因或抑癌基因都有着紧密联系[6, 7, 8]。随着对肿瘤发病机制研究的不断深入,基因敲除动物模型在肿瘤基础研究中发挥的作用也越来越大[9, 10]。基因敲除动物可以有很多应用,包括探讨化学药物与基因或蛋白表达产物的关系,幽门螺杆菌等在胃癌形成中的作用机制[17, 18]。
CRISPR/Cas系统是细菌和古细菌中用来抵御噬菌体和质粒等外源DNA片段入侵而建立起来的防御机制[11, 12],由Clustered regularly interspaced short palindromic repeats (CRISPR) 和CRISPR-associated (CAS)基因组成。CRISPR系统的免疫干扰过程主要包括3个阶段:适应、表达和干扰。在适应阶段,CRISPR系统会将来自噬菌体或质粒的DNA短片段整合到前导序列和第一段重复序列之间,每次整合都伴随着重复序列的复制,进而形成新的重复-间隔序列单元。在表达阶段,CRISPR基因座会被转录成一段CRISPR RNA前体pre-crRNA,该前体在Cas蛋白和tracrRNA的存在下会在重复序列处被进一步加工成小的crRNA,成熟的crRNA与Cas蛋白形成Cas/crRNA复合体。在干扰阶段,crRNA通过其与靶序列互补的区域引导Cas/crRNA复合体寻找靶点,并在靶点位置通过Cas蛋白的核酸酶活性造成靶点位置双链DNA断裂,从而使靶标DNA失去功能。
通过人工构建模拟crRNA-tracrRNA复合体的单链嵌合体引导RNA(guide RNA),可有效介导Cas9蛋白对靶点的识别和切割,从而为在目标物种中利用CRISPR系统对目标DNA进行修饰提供了广阔前景[13, 14, 15, 16]。总之,CRISPR/Cas9具有许多技术优越性:克服现有技术在构建基因敲除小鼠技术中存在的周期长、工作量大、难度高、效率低等不足;免去了构建以及优化筛选ZFN和TALEN串联碱基识别模块组合的过程;可供靶向序列相对于ZFN或者TALEN有着更高的出现频率及更大的灵活性,从而为精确的基因组编辑提供了更多选择。
本课题利用CRISPR/Cas9基因编辑技术,以miR-301a为靶点,通过向小鼠受精卵内显微注射CRISPR/Cas9系统有关的RNA来实现对目的基因miRNA的靶向切割与破坏。该模型的建立,为后续研究miR-301a及其他miRNAs在胃癌发病过程中的作用机制提供重要的工具,也为其他重要基因体内功能研究提供了技术借鉴。 4 利益冲突
所有作者声明本文不涉及任何利益冲突。
| [1] | Venerito M,Nardone G,Selgrad M,Rokkas T,Malfertheiner P. Gastric cancer-epidemiologic and clinical aspects [J]. Helicobacter, 2014, 19: 32-37. |
| [2] | Al Dulaimi D. Recent advances in oesophageal diseases [J]. Gastroenterol Hepatol Bed Bench, 2014, 7: 186-189. |
| [3] | Pinheiro D D, Ferreira W A, Barros M B, Araújo M D, Rodrigues-Antunes S, Borges B D. Perspectives on new biomarkers in gastric cancer: Diagnostic and prognostic applications [J]. World J Gastroenterol, 2014, 20: 11574-11585. |
| [4] | 徐 飚,王建明. 胃癌流行病学研究[J]. 中华肿瘤防治杂志,2006,13: 1-7. |
| [5] | 王 妍, 王晶桐. 429例胃癌流行病学资料分析[J]. 胃肠病学和肝病学杂志,2007, 6: 525-527. |
| [6] | Shen X B, Wang J, Li P F, Ren X F, Yan X L, Wang F. Screening of susceptibility genes and multi-gene risk analysis in gastric cancer[J]. Med Oncol,2014,31:196. |
| [7] | D'Angelo G, Di Rienzo T, Ojetti V. Microarray analysis in gastric cancer: A review [J]. World J Gastroenterol,2014,20: 11972-11976. |
| [8] | Liu H S, Xiao H S. MicroRNAs as potential biomarkers for gastric cancer [J]. World J Gastroenterol, 2014, 20: 12007-12017. |
| [9] | Gerlai R. Gene-targeting studies of mammalian behavior: is it the mutation or the background genotype?[J]. Trends Neurosci, 1996,19: 177-181. |
| [10] | Wolfer D P, Crusio W E, Lipp H P. Knockout mice: simple solutions to the problems of genetic background and flanking genes [J]. Trends Neurosci,2002, 25: 336-340. |
| [11] | Makarova K S,Grishin N V,Shabalina S A,Wolf Y I,Koonin E V. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action [J]. Biol Direct,2006,1:7. |
| [12] | Wiedenheft B,Sternberg S H,Doudna J A. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature,2012,482: 331-338. |
| [13] | Mali P, Yang L, Esvelt K M, Aach J, Guell M, DiCarlo J E, et al.RNA-guided human genome engineering via Cas9 [J]. Science, 2013,339: 823-826. |
| [14] | Cong L,Ran F A,Cox D,Lin S,Barretto R,Habib N,et al.Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339: 819-823. |
| [15] | Jinek M,Chylinski K,Fonfara I,Hauer M,Doudna J A,Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science,2012,337: 816-821. |
| [16] | Qiu Z,Liu M,Chen Z,Shao Y,Pan H,Wei G,et al. High-efficiency and heritable gene targeting in mouse by transcription activator-like effector nucleases [J]. Nucleic Acids Res, 2013, 41: e120. |
| [17] | Takaishi S, Wang T C.Gene expression profiling in a mouse model of Helicobacter-induced gastric cancer[J]. Cancer Sci,2007, 98: 284-293. |
| [18] | Taketo M M. Mouse models of gastrointestinal tumors [J]. Cancer Sci,2006, 97: 355-361. |
 2015, Vol. 36
2015, Vol. 36


