肝癌是人类最常见、预后最差的肿瘤之一[1],也是中国癌症致死最主要的原因之一[2]。研究显示肝癌的发生过程模拟了肝脏的发育过程,肝癌细胞与胚胎肝细胞存在许多共同的特点[3],许多在正常肝脏中不表达的分子在肝癌细胞和胚胎肝细胞中上调表达,可用于肝癌的诊断和治疗,如诊断肝癌最常用的分子标志物甲胎蛋白(AFP)以及肝癌患者预后的分子标志物癌胚蛋白SALL4[4],后者还是肝癌治疗的潜在靶点[5]。本课题组在前期研究中发现 长链非编码RNA(lncRNA)-LALR1可以通过调控Wnt/β-catenin信号通路促进肝脏再生[6],而Wnt/β-catenin信号通路对肝癌的发生、发展具有十分重要的意义[7],因此在肝再生过程中差异表达的 lncRNA也可能在肝癌的发生、发展中发挥重要作用。
lncRNA是一类转录本超过200 nt的非编码RNA。研究表明lncRNA与肝脏的病理生理过程有着十分密切的关系,其参与了胚胎肝脏的发育[8]、肝脏损伤后再生[6]、酒精性肝病[9]及肝脏肿瘤的发生、发展[10, 11]。因此,本研究拟通过对已有的肝发育及肝再生lncRNA芯片进行分析及初步的功能研究,以期寻找肝癌发生、发展过程中的关键lncRNA,并探讨其对肝癌细胞增殖、侵袭的影响,为肝癌的治疗提供潜在靶点。 1 材料和方法 1.1 主要试剂及实验动物
Hepa1-6(中国科学院上海生命科学院细胞资源中心),C57BL/6J小鼠 [上海斯莱克实验动物有限公司,许可证号:SCXK(沪)2012-0002],胎牛血清(Gibco),DMEM培养基(HyClon),二乙基亚硝胺(DEN,Sigma),四氯化碳、橄榄油(国药集团化学试剂有限公司)、SYBR Green、TRIzol试剂(TaKaRa),MMLV、LipofectamineTM2000(Invitrogen),siRNA(上海吉玛制药技术有限公司),EdU标记试剂盒(广州市锐博生物科技有限公司)。 1.2 细胞培养
以DMEM培养基培养Hepa1-6细胞,内含10%胎牛血清,于37℃、饱和湿度、5%CO2的培养箱中培养,取对数生长期细胞用于实验。 1.3 细胞转染
培养Hepa1-6 至对数生长期,细胞先用PBS洗2 次,加入无血清DMEM培养基。将siRNA和LipofectamineTM2000分别加入Opti-MEM中,轻轻震摇5 min,混匀,制成转染液。室温静置20 min后,将转染液加入细胞中,轻轻混合,置37℃孵育 6 h。弃去转染液,加含10%胎牛血清的DMEM继续培养。 1.4 小鼠动物模型的建立
肝癌模型建立:选取12只15 d雄性C57BL/6J小鼠,腹腔注射二乙基亚硝胺(DEN),注射剂量为25 mg/kg。10个月后处死小鼠,分离肝癌组织及正常肝组织,H-E染色确认是否为肝癌组织和正常肝组织。
肝部分切除模型建立:选取20 g左右雄性C57BL/6J小鼠12只,在麻醉状态下,切除小鼠左叶和中叶肝脏,以手术结束为0 h,分别在0、1.5、12 h及24 h处死小鼠,分离肝脏。
四氯化碳诱导的肝损伤修复模型建立:选取20 g左右雄性C57BL/6J小鼠12只,将四氯化碳以橄榄油稀释成浓度为0.3%的混合液,以10 mL/kg的剂量对小鼠进行灌胃。以灌胃完成为0 h,在不同时间点(0、6、12 h及24 h)处死小鼠,分离肝脏。
DEN诱导的肝损伤修复模型建立:选取20 g左右雄性C57BL/6J小鼠9只,以200 mg/kg的剂量腹腔注射DEN,以完成腹腔注射时为0 h,在不同时间点(0、6 h及12 h)处死小鼠,分离肝脏。
对于肝损伤修复模型,各个时间点的肝脏组织一部分用于H-E染色,除0 h外的其他时间点肝组织出现肝细胞的肿胀、空泡、核皱缩或破裂时,认为肝损伤修复模型建立成功,可用于后续实验。 1.5 RNA抽提、反转录及实时定量PCR
取1×106 Hepa1-6细胞或研磨均匀的组织,用TRIzol-氯仿萃取法抽提RNA,测得D260/D280为1.9~2.1。取2 μg RNA经MMLV反转录为cDNA。取1 μLcDNA为模板,以GAPDH为内参,用SYBR Green方法检测目的基因表达(引物序列见表 1)。
| 表 1 引物序列信息 Tab 1 Oligonucleotide sequences of primer |
12孔板中转染细胞,培养48 h后,吸去细胞培养液,每孔加入500 μL含EdU (25 μmol/L)的培养基孵育2 h。 经固定(4%多聚甲醛,30 min)和通透 (0.5% Triton X-100,10 min)处理后,加入500 μL的Apollo染色反应液(避光),DAPI染核,最后获取图像并进行定量分析。 1.7 Transwell实验检测细胞侵袭能力
常规消化Hepa1-6细胞后用无血清培养基调整细胞密度为2.0×105个细胞/mL,将200 μL细胞悬液移入小室内,小室植入24孔板内,在小室下层加入500 μL含10% FBS的完全培养基,培养箱孵育24 h,取出Transwell小室, 用无菌棉签擦去上室面细胞及Matrigel,甲醛固定,结晶紫染色,在显微镜下计数5个视野穿膜细胞数,求出每个视野平均穿膜细胞数。 1.8 芯片数据及分析
肝发育芯片数据[8]及肝再生芯片数据(GSE43687)均来自Gene Expression Omnibus数据库(www.ncbi.nlm.nih.gov/geo)。根据各lncRNA的SeqID寻找2个芯片数据中同时表达上调的lncRNA。 1.9 统计学处理
采用SPSS 18.0 软件进行统计分析,实时定量PCR数据用2-ΔΔCt法分析,独立样本t检验用于分析两组之间差异,数据以 x±s 表示;检验水准(α)为0.05。 2 结 果 2.1 小鼠原发肝癌模型的建立
结果(图 1)表明:实验组小鼠肝脏可见多发结节,对照组小鼠未见异常。H-E染色结果(图 1)表明:实验组结节组织细胞呈现肝细胞癌表现,细胞排列紊乱,核大深染,细胞异型性明显,而对照组未见异常。结果证实,小鼠原发肝癌模型成功建立,可用于后续研究。
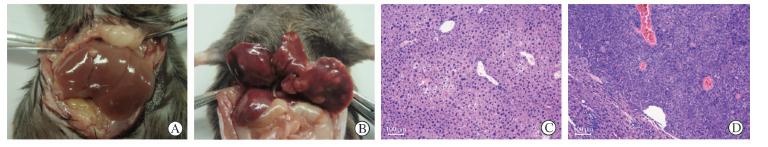 | 图 1 小鼠原发肝癌模型的建立 Fig 1 Establishment of mouse HCC modelA,B: Mouse liver of negative control group (A) or DEN-treated group (B); C,D: Hematoxylin and eosin staining of mouse liver tissue from negative control group (C) or nodule from DEN-treated group (D). Scale bars represent 100 μm |
2.2 肝部分切除模型及胎肝中共同高表达的lncRNA的筛选肝再生芯片数据包含小鼠肝脏部分切除后的时间点为:0、1.5、6、24 h。将除0 h外的3个时间点的数据与0 h比较从而得到在肝部分切除模型中特异高表达的lncRNA共783条;小鼠肝发育芯片包含的数据为:12.5、14.5、17.5 d小鼠胚胎肝脏及新生小鼠和成年小鼠肝脏,通过将小鼠胚胎肝脏与成年小鼠肝脏芯片数据进行比较,进而得到在小鼠胚胎肝脏中特异高表达的lncRNA共443条。将两者综合分析从而得到在小鼠胚胎肝脏及小鼠肝部分切除中同时高表达的候选lncRNA共7条(表 2)。
| 表 2 候选lncRNA详细信息 Tab 2 Information of candidate lncRNAs |
结果(图 2A)表明:lncRNA-AK042594、AK007113和AK003710在DEN诱导的小鼠肝癌组织中较正常肝组织表达上调,上调表达倍数分别为:2.57倍(P=0.023)、3.97倍(P=0.0007)、5.54倍(P=0.0005),差异均有统计学意义;lncRNA-UC.239+、AK035610和AK086441在肝癌及正常组织中表达差异无统计学意义(P>0.05);利用实时定量PCR无法在肝癌组织中检测到lncRNA-AK166273的表达。
lncRNA-AK042594、AK007113和AK003710在肝癌组织中高表达,芯片数据显示lncRNA-AK003710在胚胎肝脏及肝部分切除模型中表达倍数均较lncRNA-AK042594、AK007113高(表 2),而real-time PCR结果也显示,lncRNA-AK003710在癌组织和癌旁组织差异倍数最大。因此本研究首先选择lncRNA-AK003710作为进一步研究的对象。通过生物信息学分析,lncRNA-BC036412可能是lncRNA-AK003710在人类中的同源物,利用real-time PCR检测lncRNA-BC036412在人类肝癌组织中的表达量,结果如图 2B:lncRNA-BC036412在人类肝癌组织中的表达量高于癌旁组织,差异有统计学意义(P=0.02)。
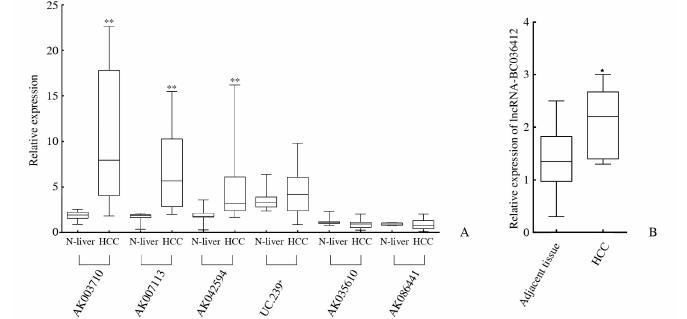 | 图 2 实时定量PCR检测lncRNA在肝癌组织中的表达 Fig 2 Expression of lncRNAs in HCC tissue by real-time PCRA: Expression of candidate lncRNAs in mouse normal liver tissue (N-liver) and HCC tissue by real-time quantitative PCR,n=12; B: The expression of lncRNA-BC036412 in human HCC tissue,n=10. Horizontal lines in the box plots represent the medians,the boxes represent the interquartile range,and the whiskers represent the 2.5th and 97.5th percentiles. *P<0.05,**P<0.01 vs N-liver (A) or adjacent tissue (B) |
结果(图 3)表明: lncRNA-AK003710在胚胎肝脏的各个时间段均较成年肝脏高表达(P<0.01),其在12.5 d胚胎肝脏中表达最高,为成年肝脏的140倍。lncRNA-AK003710在肝损伤修复模型中的表达情况如图 4:lncRNA-AK003710在肝部分切除后12、24 h的肝脏组织均较0 h表达上调,差异有统计学意义(P=0.021 9,P=0.000 5);在四氯化碳肝损伤修复模型中,lncRNA-AK003710在四氯化碳处理后6、12、24 h的肝脏组织均较0 h表达上调,差异有统计学意义(P=0.011 5,P=0.000 1,P=0.001 5);同时lncRNA-AK003710也在DEN诱导的肝损伤修复模型中表达上调,lncRNA-AK003710在DEN处理后6、12 h肝脏组织中的表达均较0 h高,差异有统计学意义(P=0.029 8,P=0.032 1)。
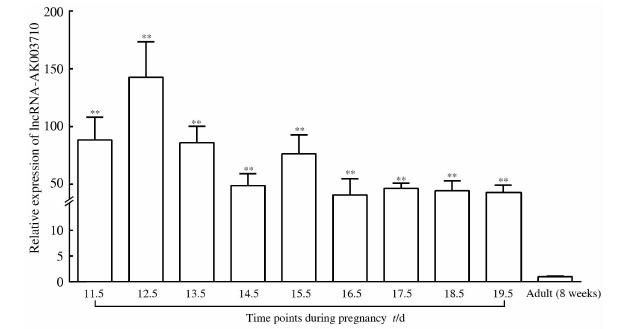 | 图 3 实时定量PCR检测小鼠胚胎肝脏及成年肝脏中lncRNA-AK003710表达 Fig 3 Expression of lncRNA-AK003710 in fetal liver and adult liver by real-time quantitative PCR |
 | 图 4 实时定量PCR检测3种小鼠肝损伤修复模型不同时间点lncRNA-AK003710的表达 Fig 4 Expression of lncRNA-AK003710 in three kinds of injured liver tissues at different time points by real-time quantitative PCR A: PH(partial hepatectomy)induced liver injury tissue; B: CCl4(carbon tetrachloride) induced liver injury tissue; C: DEN (diethylinitrosamine) induced liver injury tissue. *P<0.05,**P<0.01 vs 0 h; n=3, x±s |
用针对lncRNA-AK003710的siRNA(siAK003710)及阴性对照siRNA(siNC)转染小鼠肝癌细胞系Hepa1-6,然后利用EdU标记技术检测转染siRNA后Hepa1-6细胞的增殖能力,结果如图 5A、5B,转染siAK003710组细胞EdU阳性细胞为(10.53±0.85)%,低于对照组的(20.56±1.46)%(P=0.006),提示干扰lncRNA-AK003710后能降低细胞的增殖能力。 Hepa1-6转染siRNA后利用Transwell实验检测细胞的侵袭能力,结果如图 5C、5D,转染siAK003710组细胞侵袭效率为(28.33±3.76)个细胞/视野,低于对照组(56.00±2.08)个细胞/视野(P=0.003),提示干扰lncRNA-AK003710后细胞侵袭能力减弱。
 | 图 5 转染siRNA后Hepa1-6细胞增殖(A、B)及侵袭能力(C、D)的测定 Fig 5 Proliferation (A,B) and invasion ability (C,D) of Hepa1-6 cells transfected with siRNAA,C: siNC; B,D: siAK007310. Original magnification: ×200 (A,B),×100 (C,D) |
研究表明哺乳动物基因组的转录产物大部分为非编码RNA,而且这些RNA似乎有着许多未知的功能[12],lncRNA的功能可能会更多样,对生命过程的调控会更精细。近年来,随着芯片及二代测序技术的广泛使用,lncRNA 逐渐成为研究的热点,lncRNA在不同生命过程中各种各样的功能逐渐被揭示,如参与X染色体失活[13]、调控免疫细胞分化[14]、抑制心肌肥大[15]及调控肿瘤的发生、发展[16]等。尽管有许多具有重要功能的lncRNA被发现,可是仍然有许多lncRNA的功能不清楚,还有待进一步的研究。在以往的lncRNA研究中,筛选lncRNA时往往只考虑了某一个生理或病理过程,但是本研究充分考虑了肝发育、肝损伤修复和肝癌这3个重要的生命过程,找到了在3个过程中同时上调表达的lncRNA,增强了筛选结果的可靠性,同时也提示其在肝癌发生、发展过程中的重要性。而干扰lncRNA-AK003710后肝细胞增殖减慢、侵袭能力减弱,说明lncRNA-AK003710对肝细胞增殖和侵袭具有调控作用,是肝癌发生、发展过程中具有重要功能的lncRNA,可以成为肝癌治疗的潜在靶点。
为进一步研究lncRNA-AK003710,我们首先通过RACE技术获得其全长,确定转录本在基因组的具体起始位点。然后对lncRNA-AK003710的启动子区进行转录因子预测,揭示其表达上调的原因。本研究对lncRNA-AK003710的功能研究主要在细胞水平上进行。本研究利用实时定量PCR的2对引物均无法检测到lncRNA-AK166273在肝癌组织中的表达,同时在其他肝脏组织和细胞系中也无法检测到lncRNA-AK166273的表达。其原因可能有:(1)在肝脏中lncRNA-AK166273不表达;(2)lncRNA-AK166273在肝脏中表达量太低,而无法用实时定量PCR检测到;(3)数据库中lncRNA-AK166273的序列与实际序列不一致。本研究中除了lncRNA-AK003710外,lncRNA-AK042594和lncRNA-AK007113也同时在肝发育、肝损伤修复和肝癌中上调表达,后两者在3个过程中同时上调表达的意义及发挥作用分子机制也将是下一步研究的重点。
癌胚基因是目前研究的热点,癌胚基因是指那些在正常组织中低表达,而在胚胎期组织和肿瘤组织中开放表达的基因。这些基因的编码产物往往在肿瘤的发生发展过程中发挥重要的作用,抑制这些癌胚蛋白的活性能够抑制肿瘤细胞的生长或转移。这些基因既包含了像c-myc这样经典的癌基因,也有一些新近发现的癌胚基因,如ROR1(receptor tyrosine kinase-like orphan receptor 1)[17]、IGF2BP(IGF2 mRNA-binding protein)[18]等。但是前人关于癌胚基因产物的研究多限于蛋白编码基因,关于癌胚lncRNA的研究却非常少,毫无疑问癌胚lncRNA拓宽了人们对癌胚基因的认识,为肿瘤的研究提供了新策略与思路。
综上所述,lncRNA-AK003710在肝发育、肝损伤修复和肝癌中上调表达,是一个癌胚RNA;lncRNA-AK003710可以调控肝细胞增殖和侵袭,有望成为肝癌治疗的潜在靶点。 4 利益冲突
所有作者声明本文不涉及任何利益冲突。
| [1] | Jemal A, Bray F, Center M M, Ferlay J, Ward E, Forman D. Global cancer statistic[J]. CA Cancer J Clin, 2011,61:69-90. |
| [2] | He J, Gu D, Wu X, Reynolds K, Duan X, Yao C, et al. Major causes of death among men and women in China[J]. N Engl J Med,2005,353:1124-1134. |
| [3] | Becker D, Sfakianakis I, Krupp M, Staib F, Gerhold-Ay A, Victor A, et al. Genetic signatures shared in embryonic liver development and liver cancer define prognostically relevant subgroups in HCC[J]. Mol Cancer, 2012,11:55. |
| [4] | Oikawa T, Kamiya A, Zeniya M, Chikada H, Hyuck A D, Yamazaki Y, et al. Sal-like protein 4 (SALL4), a stem cell biomarker in liver cancers[J]. Hepatology,2013,57:1469-1483. |
| [5] | Masuda S, Suzuki K, Izpisua Belmonte J C. Oncofetal gene SALL4 in aggressive hepatocellular carcinoma[J]. N Engl J Med,2013,369:1171. |
| [6] | Xu D, Yang F, Yuan J H, Zhang L, Bi H S, Zhou C C, et al.Long noncoding RNAs associated with liver regeneration 1 accelerates hepatocyte proliferation during liver regeneration by activating Wnt/β-Catenin signaling[J]. Hepatology,2013,58:739-751. |
| [7] | Thompson M D, Monga S P. WNT/beta-catenin signaling in liver health and disease[J]. Hepatology, 2007,45:1298-1305. |
| [8] | Wang F, Yuan J H, Wang S B, Yang F, Yuan S X, Ye C, et al. Oncofetal long noncoding RNA PVT1 promotes proliferation and stem cell-like property of hepatocellular carcinoma cells by stabilizing NOP2[J]. Hepatology, 2014, 60:1278-1290. |
| [9] | Oliva J, Bardag-Gorce F, French B A, Li J, French S W. The regulation of non-coding RNA expression in the liver of mice fed DDC[J]. Exp Mol Pathol, 2009,87:12-19. |
| [10] | Yang F, Huo X S, Yuan S X, Zhang L, Zhou W P, Wang F, et al. Repression of the long noncoding RNA-LET by histone deacetylase 3 contributes to hypoxia-mediated metastasis[J]. Mol Cell,2013,49:1083-1096. |
| [11] | Yuan J H, Yang F, Wang F, Ma J Z, Guo Y J, Tao Q F, et al. A long noncoding RNA activated by TGF-β promotes the invasion-metastasis cascade in hepatocellular carcinoma. [J]. Cancer Cell,2014,25:666-681. |
| [12] | Wilusz J E, Sunwoo H, Spector D L. Long noncoding RNAs: functional surprises from the RNA world[J].Genes Dev,2009,23:1494-1504. |
| [13] | Engreitz J M, Pandya-Jones A, McDonel P, Shishkin A, Sirokman K, Surka C, et al. The Xist lncRNA exploits three-dimensional genome architecture to spread across the X chromosome[J].Science, 2013,341:1237973. |
| [14] | Wang P, Xue Y, Han Y, Lin L, Wu C, Xu S, et al. The STAT3-Binding long noncoding RNA lnc-DC controls human dendritic cell differentiation[J]. Science, 2014,344:310-313. |
| [15] | Han P, Li W, Lin C H, Yang J, Shang C, Nuernberg S T, et al. A long noncoding RNA protects the heart from pathological hypertrophy[J]. Nature, 2014, 514:102-106. |
| [16] | Hu X, Feng Y, Zhang D, Zhao S D, Hu Z, Greshock J, et al A functional genomic approach identifies FAL1 as an oncogenic long noncoding RNA that associates with BMI1 and represses p21 expression in cancer[J]. Cancer Cell, 2014,26:344-357. |
| [17] | Widhopf G F, Cui B, Ghia E M, Chen L, Messer K, Shen Z, et al. ROR1 can interact with TCL1 and enhance leukemogenesis in Eμ-TCL1 transgenic mice[J]. Proc Natl Acad Sci USA, 2014,111:793-798. |
| [18] | Hämmerle M, Gutschner T, Uckelmann H, Ozgur S, Fiskin E, Gross M, et al. Posttranscriptional destabilization of the liver-specific long noncoding RNA HULC by the IGF2 mRNA-binding protein 1 (IGF2BP1) [J].Hepatology, 2013,58:1703-1712. |
 2015, Vol. 36
2015, Vol. 36


