后纵韧带骨化症(ossification of posterior longitudinal ligament,OPLL)是由于颈椎或胸腰椎韧带发生异位骨化从而压迫脊髓和神经根,产生肢体感觉和运动障碍及内脏植物神经功能紊乱的疾病,多见于亚洲人群[1, 2],发病率高达2%~4%[3]。韧带骨化进展缓慢,临床表现为从没有症状的单纯影像学改变到严重的脊髓压迫导致不可逆的四肢瘫,患者症状的严重程度与骨化物的大小及范围密切相关。由于易与普通颈椎病相混淆及其本身外科治疗的复杂程度,韧带骨化仍是临床上难以处理的问题。
目前研究表明韧带骨化是一种与遗传及环境等多种因素相关的疾病,其中遗传背景被认为是最主要的致病因素。近年来单核苷酸多态性(single nucleotide polymorphism,SNP)研究逐渐成为探索OPLL相关易感基因位点的主要方法。在日本人群中,COL6A1基因已被证实是OPLL发生发展的易感基因[4, 5],并且BMP-2、VDR、Runx2等基因也相继被报道与韧带骨化相关[6, 7, 8]。由于在不同的种族人群中候选基因及其多态性位点可能存在不同,本研究对汉族人群中上述4个候选基因的19个SNP位点进行分析,旨在寻找汉族人群中OPLL患者的易感基因及与疾病相关的SNP位点。 1 资料和方法 1.1 病例选择
本研究由第二军医大学伦理委员会审查批准。选取2011年1月至2012年12月间我科收治的脊柱疾患患者,所有病例入院时均签署知情同意书。实验组病例为同一组医生通过临床症状及影像学检查诊断为OPLL的患者,并通过CT及MRI判断脊髓受压、椎管形态、骨化物大小及其范围。对照组为同期入院的非OPLL患者。两组患者中,非汉族人群、强直性脊柱炎、弥漫性特发性骨肥大(diffuse idiopathic skeletal hyperostosis,DISH)、脊柱感染及肿瘤、长期服用影响骨代谢药物的患者排除在外。本研究共纳入180例患者,实验组78例,其中男性60例、女性18例,平均年龄(53.31±9.73)岁;对照组102例,男性75例、女性27例,平均年龄(53.79±10.06)岁。 1.2 基因组DNA准备
获取所有患者的外周血,EDTA抗凝保存。通过标准程序应用TIANamp Blood DNA 试剂盒 (天根科技生化有限公司,DP318-03)抽提血液标本中的白细胞DNA,-80℃保存以待后续分析。 1.3 基因组分型
使用基质辅助激光解吸电离飞行时间(MALDI-TOF)质谱技术对SNP分型进行分析,参照MassArray系统 (Sequenom,San Diego,USA) 标准操作程序。根据前人的研究结果,选择COL6A1、BMP-2、VDR、Runx2基因为SNP分型的研究对象。先对含有目的SNP位点的基因片段进行多重PCR扩增,多重PCR反应体系使用Assay Design software 3.1 (Sequenom Inc.,San Diego,CA)设计。反应体系包含10 ng/μL基因组DNA,12.5 nmol/L引物,25 mmol/L MgCl2,25 mmol/L dNTPs和0.15 U HotStar Taq DNA polymerase (Qiagen,Hilden,Germany),反应体系共10 μL。PCR反应条件:94℃ 15 min,然后扩增45个循环(94℃变性20 s、56℃退火30 s、72℃延伸1 min),最后72℃延伸3 min。扩增产物以北极虾碱性磷酸酶(SAP)处理(85℃ 5 min,37℃ 40 min),将残余核苷酸去磷酸化,即去除反应体系中剩余的dNTPs。在单碱基延伸反应中,将PCR反应产物与0.625~1.25 μmol/L的 hME引物组合混合(延伸引物序列及未延伸和延伸后引物的相对分子质量见表 1),反应体系含有1×iPLEX terminator、1×iPLEX enzyme 和 1×iPLEX 缓冲液。
|
|
表 1 延伸引物序列及未延伸和延伸后引物的相对分子质量 Tab 1 Sequence of extented primers,molecular weight of non-extended primers and extended primers |
用Hardy-Weinberg (HW)平衡定律描述人群中基因位点稳定情况[9],各等位基因的分布频率和等位基因的基因分型分布频率通过直接计数方法计算。等位基因位点与疾病之间的关系通过χ2检验计算获得(以OR及其95%可信区间描述)。用Haploview软件(Massachusetts Institute of Technology,Cambridge,MA,USA)计算基因位点之间的连锁不均衡(linkage disequilibrium,LD),以LD系数(D’)和r2描述位点与疾病的关系。采取可信区间方法设定单体型域(haplotype block),每一个对比组定义为“强相关”、“不相关”、“再结合”。如果D’的95%可信值上界大于0.98且下界大于0.7,则认为这对SNP为“强连锁不均衡”;反之,如果D’的95%可信值上界小于0.9,则认为SNP为“强复合”(参考Gabriel等[10]的方法进行计算)。若95%的对比组为“强连锁不均衡”,则建立一个域,在每一个域中,单体型发生的频率用EM方法估计。对于单元型在两个域中的频率均大于1%的病例,将利用χ2检验及风险估计预测单体型与疾病的关系。检验水准(α)为0.05。统计分析利用Haploview、SHEsis(上海Bio-X 生命科学研究中心)和Microsoft Excel 2003 (Microsoft Corp,Redmond,WA,USA)完成。 2 结 果
筛选的4个候选基因共19个SNP位点全部满足HW平衡(P>0.001),次要等位基因频率(MAF)>0.001。等位基因分析未发现与疾病相关的位点,但等位基因的基因分型分析发现rs1321075与rs12333172在实验组与对照组之间差异有统计学意义(P=0.033 9,P=0.042 8),两个基因位点均分布在6号染色体Runx2基因中(图 1)。COL6A1、BMP-2、VDR基因未发现与疾病发生显著相关的多态性位点(表 2)。
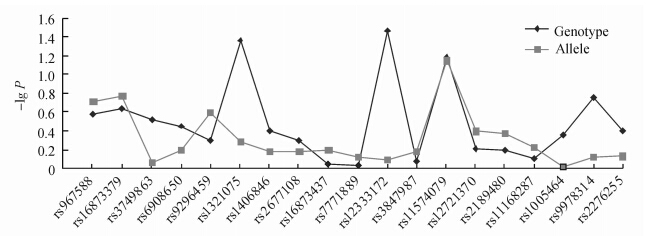 | 图 1 19个SNP位点统计分布图 Fig 1 Statistical distribution of 19 SNP loci Two SNP loci on chromosome 6 (rs1321075 and rs12333172) showed disease correlation in genotyping. No disease correlations were indentified in allelic genes |
|
|
表 2 位点与疾病相关性 Tab 2 Correlation of locus and disease |
位于12号染色体上的5个SNP位点之间的D’值范围在0.043~1之间、r2值为0~0.185,这5个SNP位点并不能形成一个域(表 3)。
|
|
表 3 12号染色体各位点的相互LD分析 Tab 3 Mutual linkage disequilibrium (LD) of loci on chromosome 12 |
位于21号染色体上的2个SNP位点之间D’= 0.92,r2=0.57,21号染色体上未发现单体型域。6号染色体上的11个SNP位点之间形成了2个单体型域(图 2),单体型域1由 rs967588、rs16873379、rs3749863、rs6908650组成,范围达51 kb,4个位点之间的D’值为0.964~1、r2值为0.299~1(表 4),4个位点形成了4个频率>0.01的单体型,单体型CTGG分别在14%的实验组患者和8%的对照组患者中出现,实验组/对照组比值为1.75 (RR值0.807 35,P=0.259),表明该单体型有可能增加疾病发生比率(表 5);单体型域2由 rs1321075、rs1406846、rs2677108组成,范围11 kb,3个位点之间的D’值为0.951~1、r2值为0.348~0.753(表 6),3个位点形成了4个频率>0.01的单体型,从每个单体型的RR值可以得出它们与疾病无关。
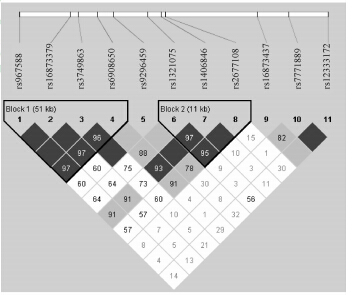 | 图 2 6号染色体上11个SNP位点LD分析 Fig 2 Linkage disequilibrium (LD) of 11 SNP loci on chromosome 6 The values in box stand for D’of loci in the upper left and right. The blank in box means D’=1. SNP locus would be excluded before LD analysis when MAF is less than 0.05 and HW is less than 0.001. The dark squares represent the linkage disequilibrium between the two loci; the darker the color,the greater the linkage disequilibrium. The white squares indicate no linkage disequilibrium between two loci. Bold wireframe is the haplotype block |
|
|
表 4 6号染色体上单体型域1中各位点的相互LD分析 Tab 4 Mutual linkage disequilibrium(LD) of loci on haplotype block 1 of chromosome 6 |
|
|
表 5 6号染色体单体型域1在病例组和对照组的风险分析 Tab 5 Linkage disequilibrium (LD) analysis,risk and significance analysis of individual locus on haplotype block 1 of chromosome 6 |
|
|
表 6 6号染色体单体型域2中各位点的相互LD分析 Tab 6 Mutual linkage disequilibrium(LD)of loci on block 2 of chrmosome 6 |
OPLL已被许多研究证明为多基因、多因素导致的遗传学疾病,近年来对其遗传学的研究将其致病 归结于候选基因SNP位点的变化增加了个体在 特定环境下的患病概率。近10年来,不同的候选基因先后被发现与OPLL的发病相关,多数研究来自日本,新的基因、新的多态性位点被一一发现。
Terayama[11]通过遗传学研究调查了347个OPLL患者家庭,发现在1 030个家庭成员中,单纯影像学表现有OPLL的患者父母达26.5%,兄弟姐妹中发现OPLL的占28.89%。Sakou等[12]于1991年对33个OPLL家庭进行人类白细胞抗原(HLA)单倍型频率研究发现,患者兄弟姐妹中有全部HLA单倍型的56%表现出OPLL,而仅有单个HLA抗原的并未表现出OPLL。Matsunaga等[13]研究发现,OPLL患者的家庭成员中,若HLA单倍型为双线性,OPLL发生率为53%;若为单线性单倍体,发生OPLL的比例为24%。上述研究提示导致OPLL的异常基因可能存在于6号染色体上HLA基因附近。
近年来OPLL在分子遗传学方面的研究进展深入到其候选基因的筛选及SNP位点的确定。双胞胎6号染色体HLA区域的配对研究确定了编码胶原Ⅺ α的基因COL11A2[14]与韧带骨化发病相关。直立鼠的OPLL动物模型研究发现NPPS基因与疾病发生及其严重程度相关[15]。Kawaguchi等[16]研究发现TGF-β1基因与骨化形成的范围有关。Tanaka等[4]对日本人群OPLL患者的基因组应用GENEHUNTER-PLUS进行多点连锁分析,确认了21号染色体上的7个基因与疾病相关,进一步LD分析发现COL6A1基因上的SNP与OPLL密切相关,单点(single-point)P值为0.000 000 7。Wang等[6]研究发现BMP-2基因中的Ser37Ala和Ser87Ser SNP与中国人OPLL遗传易感及严重程度相关。
Kamiya 等[17]2001年对日本319例非相关个体进行TGF-β的基因型分型研究,其中OPLL患者46例,对照组273例。结果发现,TGF-β基因的T869位点上C等位基因是OPLL遗传易感性的危险因素,多元回归分析显示OPLL患者该位点是C等位基因的比例远远高于对照组。
Tsukahara等[5]2005年对候选基因COL6A1做了进一步研究,他们采用直接测序的方法对COL6A1基因上的7个SNP进行了基因分型。OPLL患者的等位基因频率与97例日本DISH患者及298例对照组人群进行了比较,同时再与96例捷克DISH 患者及96名捷克对照人群比较,结果发现,COL6A1的内含子32(-29)SNP与日本DISH患者显著相关,而与捷克DISH患者没有明显关系,从而提示该基因与日本人群的OPLL和DISH发病相关,是导致脊柱韧带异位骨化的重要基因。
Horikoshi等[18]2006年对日本OPLL患者进行了大样本病例对照研究,针对文献已报道过的35个候选基因共109个SNP位点进行了基因分型,结果发现,TGF2基因上的SNP的一个内含子与疾病密切相关,而以前报道的相关基因COL11A2、NPPS及TGF-β1基因都未能重复,另外,进一步基于性别、年龄与骨化椎体的分层研究亦未显示出有明显关联。
国内学者Kong等[19]2007年针对中国汉族人群OPLL与OLF患者的COL6A1 SNP研究发现,在该基因的4个已知SNP位点中,启动子(-572T)的等位基因频率在OPLL组及OLF组要明显高于对照组,在启动子(-572)、内含子32(-29)及内含子33(+20)上的总单体型频率,OPLL与OLF组均与对照组显著不同,提示COL6A1基因可能是中国汉族人群脊柱韧带钙化致病的易感基因。
Kishiya等[7]2008年报道,他们通过DNA微阵列(microarray)检测Runx2基因表达,结果发现韧带骨化症患者的Runx2基因表达增强。Runx蛋白是一组转录因子蛋白家族,研究表明Runx2与骨分化和代谢过程中起重要作用的细胞及细胞因子关系密切,如成骨细胞、破骨细胞、骨基质蛋白、成纤维细胞生长因子(fibroblast growth factor,FGF)、TGF-β、血管内皮生长因子(VEGF)等[8, 20, 21]。体内外实验表明Runx2是骨髓间充质干细胞(MSCs)成骨分化和骨发育所必需的关键因子,对MSCs具有明显的促成骨效应[20, 21]。有实验证明Runx2表达下调会抑制成骨细胞的分化及骨质的矿化[22, 23]。近年,Iwasaki等[24]通过突变OPLL小鼠模型的Runx2的一个等位基因使Runx2的表达发生明显下调后,发现模型小鼠后纵韧带的骨化形成范围得到显著改善。本研究中,6号染色体Runx2基因中两个位点rs12333172、rs1321075基因分型与疾病有显著相关;由rs967588、rs16873379、rs3749863、rs6908650形成的单倍体CTCG 实验组与对照组比率为1.75,实验结果与Kishiya等[7]结果有一定的关联性,提示Runx2基因与OPLL发病有密切关系。
本研究通过大样本OPLL SNP分析,首次报道汉族人群中OPLL患者Runx2基因中SNP的变化导致OPLL发病可能性增加。6号染色体Runx2基因中两个位点rs12333172、rs1321075基因分型与疾病相关。而以往文献报道的COL6A1、BMP-2、VDR基因中的多态性位点未显现出与疾病显著相关。今后仍需进行更大范围的基因分型及更多样本的深入研究,以明确OPLL患者Runx2基因的SNP位点变化规律,并通过功能学研究加以验证。 4 利益冲突
所有作者声明本文不涉及任何利益冲突。
| [1] | Inamasu J, Guiot B H, Sachs D C. Ossification of the posterior longitudinal ligament: an update on its biology, epidemiology, and natural history[J]. Neurosurgery,2006,58:1027-1039. |
| [2] | Kobashi G, Washio M, Okamoto K, Sasaki S, Yokoyama T, Miyake Y,et al. High body mass index after age 20 and diabetes mellitus are independent risk factors for ossification of the posterior longitudinal ligament of the spine in Japanese subjects: a case-control study in multiple hospitals[J]. Spine (Phila Pa 1976),2004,29:1006-1010. |
| [3] | Matsunaga S, Sakou T. Ossification of the posterior longitudinal ligament[M]. Berlin: Springer,1997:3-7. |
| [4] | Tanaka T, Ikari K, Furushima K, Okada A, Tanaka H, Furukawa K,et al. Genomewide linkage and linkage disequilibrium analyses identify COL6A1, on chromosome 21, as the locus for ossification of the posterior longitudinal ligament of the spine[J].Am J Hum Genet,2003,73:812-822. |
| [5] | Tsukahara S, Miyazawa N, Akagawa H, Forejtova S, Pavelka K, Tanaka T,et al. COL6A1,the candidate gene for ossification of the posterior longitudinal ligament, is associated with diffuse idiopathic skeletal hyperostosis in Japanese[J]. Spine(Phila Pa 1976), 2005, 30:2321-2324. |
| [6] | Wang H, Liu D, Yang Z, Tian B, Li J, Meng X, et al. Association of bone morphogenetic protein-2 gene polymorphisms with susceptibility to ossification of the posterior longitudinal ligament of the spine and its severity in Chinese patients[J]. Eur Spine J,2008, 17:956-964. |
| [7] | Kishiya M, Sawada T, Kanemaru K, Kudo H, Numasawa T, Yokoyama T, et al. A functional RNAi screen for Runx2-regulated genes associated with ectopic bone formation in human spinal ligaments[J]. J Pharmacol Sci,2008, 106:404-414. |
| [8] | Stein G S, Lian J B, van Wijnen A J, Stein J L, Montecino M, Javed A,et al. Runx2 control of organization, assembly and activity of the regulatory machinery for skeletal gene expression[J]. Oncogene, 2004, 23: 4315-4329. |
| [9] | Salanti G, Amountza G, Ntzani E E, Ioannidis J P. Hardy-Weinberg equilibrium in genetic association studies: an empirical evaluation of reporting, deviations, and power[J]. Eur J Hum Genet, 2005,13:840-848. |
| [10] | Gabriel S B, Schaffner S F, Nguyen H, Moore J M, Roy J, Blumenstiel B,et al. The structure of haplotype blocks in the human genome[J]. Science,2002,296:2225-2229. |
| [11] | Terayama K. Genetic studies on ossification of the posterior longitudinal ligament of the spine[J]. Spine (Phila Pa 1976),1989,14:1184-1191. |
| [12] | Sakou T, Taketomi E, Matsunaga S, Yamaguchi M, Sonoda S, Yashiki S. Genetic study of ossification of the posterior longitudinal ligament in the cervical spine with human leukocyte antigen haplotype[J]. Spine(Phila Pa 1976),1991,16:1249-1252. |
| [13] | Matsunaga S, Yamaguchi M, Hayashi K, Sakou T. Genetic analysis of ossification of the posterior longitudinal ligament[J]. Spine (Phila Pa 1976),1999,24:937-939. |
| [14] | Koga H, Sakou T, Taketomi E, Hayashi K, Numasawa T, Harata S,et al. Genetic mapping of ossification of the posterior longitudinal ligament of the spine[J]. Am J Hum Genet,1998,62:1460-1467. |
| [15] | Koshizuka Y, Kawaguchi H, Ogata N, Ikeda T, Mabuchi A, Seichi A,et al. Nucleotide pyrophosphatase gene polymorphism associated with ossification of the posterior longitudinal ligament of the spine[J]. J Bone Miner Res,2002, 17:138-144. |
| [16] | Kawaguchi Y, Furushima K, Sugimori K, Inoue I, Kimura T.Association between polymorphism of the transforming growth factor-beta1 gene with the radiologic characteristic and ossification of the posterior longitudinal ligament[J]. Spine(Phila Pa 1976), 2003,28:1424-1426. |
| [17] | Kamiya M, Harada A, Mizuno M, Iwata H, Yamada Y.Association between a polymorphism of the transforming growth factor-beta1 gene and genetic susceptibility to ossification of the posterior longitudinal ligament in Japanese patients[J]. Spine(Phila Pa 1976),2001,26: 1264-1267. |
| [18] | Horikoshi T, Maeda K, Kawaguchi Y, Chiba K, Mori K, Koshizuka Y,et al. A large-scale genetic association study of ossification of the posterior longitudinal ligament of the spine[J]. Hum Genet, 2006,119:611-616. |
| [19] | Kong Q, Ma X, Li F, Guo Z, Qi Q, Li W,et al. COL6A1 polymorphisms associated with ossification of the ligamentum flavum and ossification of the posterior longitudinal ligament[J]. Spine (Phila Pa 1976), 2007, 32:2834-2838. |
| [20] | Saito T, Ogawa M, Hata Y, Bessho K.Acceleration effect of human recombinant bone morphogenetic protein-2 on differentiation of human pulp cells into odontoblasts[J]. J Endod, 2004, 30: 205-208. |
| [21] | Zhang X, Aubin J E, Inman R D.Molecular and cellular biology of new bone formation: insights into the ankylosis of ankylosing spondylitis[J]. Curr Opin Rheumatol, 2003, 15: 387-393. |
| [22] | Derynck R, Zhang Y E. Smad-dependent and Smad-independent pathways in TGF-beta family signalling[J]. Nature, 2003, 425: 577-584. |
| [23] | Xue T, Mao Z, Lin L, Hou Y, Wei X, Fu X,et al. Non-virus-mediated transfer of siRNAs against Runx2 and Smad4 inhibit heterotopic ossification in rats[J]. Gene Ther, 2010, 17: 370-379. |
| [24] | Iwasaki M, Piao J, Kimura A, Sato S, Inose H, Ochi H,et al.Runx2 haploinsufficiency ameliorates the development of ossification of the posterior longitudinal ligament[J]. PLoS One, 2012, 7: e43372. |
 2015, Vol. 36
2015, Vol. 36


